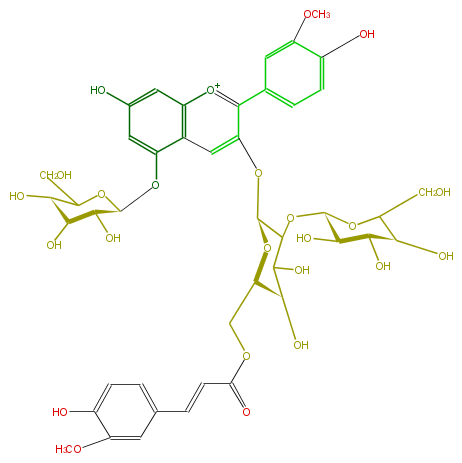
Mol:FL7AADGL0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 68 74 0 0 0 0 0 0 0 0999 V2000 | + | 68 74 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8470 2.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8470 2.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8470 2.1137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8470 2.1137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2907 1.7925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2907 1.7925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7344 2.1137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7344 2.1137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7344 2.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7344 2.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2907 3.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2907 3.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1781 1.7925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1781 1.7925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3782 2.1137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3782 2.1137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3782 2.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3782 2.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1781 3.0773 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.1781 3.0773 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9343 3.0771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9343 3.0771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5012 2.7498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5012 2.7498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0682 3.0771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0682 3.0771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0682 3.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0682 3.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5012 4.0592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5012 4.0592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9343 3.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9343 3.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4031 3.0771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4031 3.0771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9342 4.2318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9342 4.2318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2907 1.1504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2907 1.1504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7943 1.3930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7943 1.3930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2728 0.0789 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.2728 0.0789 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.7657 0.4666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.7657 0.4666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.9753 -0.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9753 -0.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7222 -0.8295 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.7222 -0.8295 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.2728 -1.1474 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.2728 -1.1474 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.0981 -0.5361 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.0981 -0.5361 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.4476 0.4838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4476 0.4838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5996 -0.5800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5996 -0.5800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5773 -2.0999 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5773 -2.0999 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1976 -1.6808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1976 -1.6808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1172 0.5812 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.1172 0.5812 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.4207 -0.0107 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4207 -0.0107 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.0295 0.1104 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.0295 0.1104 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.5802 0.0194 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.5802 0.0194 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.2512 0.4894 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.2512 0.4894 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.7064 0.3142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7064 0.3142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7987 0.0680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7987 0.0680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2592 -0.4951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2592 -0.4951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5335 -0.2828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5335 -0.2828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5600 -2.3149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5600 -2.3149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4495 0.8762 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.4495 0.8762 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.0783 0.3863 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.0783 0.3863 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.5438 0.5941 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.5438 0.5941 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.0281 0.5997 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.0281 0.5997 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.4029 0.9745 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4029 0.9745 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8705 0.7278 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.8705 0.7278 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.0404 0.9280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0404 0.9280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4287 -0.0750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4287 -0.0750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2376 0.0800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2376 0.0800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2221 -2.9001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2221 -2.9001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5498 -3.4677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5498 -3.4677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3447 -2.9001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3447 -2.9001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6688 -3.4614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6688 -3.4614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2957 -3.4614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2957 -3.4614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6082 -2.9202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6082 -2.9202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2331 -2.9202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2331 -2.9202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5456 -3.4614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5456 -3.4614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2331 -4.0027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2331 -4.0027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6082 -4.0027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6082 -4.0027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1699 -3.4614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1699 -3.4614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7846 4.6353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7846 4.6353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2260 5.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2260 5.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5866 1.3563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5866 1.3563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1679 1.9417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1679 1.9417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1101 1.0015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1101 1.0015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9831 0.5137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9831 0.5137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8886 -4.2228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8886 -4.2228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1742 -4.6353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1742 -4.6353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 30 40 1 0 0 0 0 | + | 30 40 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 19 1 0 0 0 0 | + | 44 19 1 0 0 0 0 |
| − | 40 50 1 0 0 0 0 | + | 40 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 50 52 1 0 0 0 0 | + | 50 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 2 0 0 0 0 | + | 58 59 2 0 0 0 0 |
| − | 59 54 1 0 0 0 0 | + | 59 54 1 0 0 0 0 |
| − | 57 60 1 0 0 0 0 | + | 57 60 1 0 0 0 0 |
| − | 15 61 1 0 0 0 0 | + | 15 61 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 46 63 1 0 0 0 0 | + | 46 63 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 35 65 1 0 0 0 0 | + | 35 65 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | 58 67 1 0 0 0 0 | + | 58 67 1 0 0 0 0 |
| − | 67 68 1 0 0 0 0 | + | 67 68 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 65 66 | + | M SAL 4 2 65 66 |
| − | M SBL 4 1 71 | + | M SBL 4 1 71 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 71 3.9633 0.5103 | + | M SVB 4 71 3.9633 0.5103 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 63 64 | + | M SAL 3 2 63 64 |
| − | M SBL 3 1 69 | + | M SBL 3 1 69 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 69 -3.2539 1.6001 | + | M SVB 3 69 -3.2539 1.6001 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 67 68 | + | M SAL 2 2 67 68 |
| − | M SBL 2 1 73 | + | M SBL 2 1 73 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 73 -2.8886 -4.2228 | + | M SVB 2 73 -2.8886 -4.2228 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 61 62 | + | M SAL 1 2 61 62 |
| − | M SBL 1 1 67 | + | M SBL 1 1 67 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 67 1.7846 4.6353 | + | M SVB 1 67 1.7846 4.6353 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AADGL0023 | + | ID FL7AADGL0023 |
| − | KNApSAcK_ID C00006871 | + | KNApSAcK_ID C00006871 |
| − | NAME Peonidin 3-ferulyldiglucoside-5-glucoside | + | NAME Peonidin 3-ferulyldiglucoside-5-glucoside |
| − | CAS_RN 144093-82-3 | + | CAS_RN 144093-82-3 |
| − | FORMULA C44H51O24 | + | FORMULA C44H51O24 |
| − | EXACTMASS 963.27702756 | + | EXACTMASS 963.27702756 |
| − | AVERAGEMASS 963.86134 | + | AVERAGEMASS 963.86134 |
| − | SMILES c(c(OC)7)c(ccc7O)C=CC(=O)OC[C@H]([C@H](O)1)O[C@@H](Oc(c(c(c6)cc(OC)c(O)c6)5)cc(c([o+1]5)4)c(cc(c4)O)O[C@H](O3)[C@H]([C@@H](O)[C@@H](O)C3CO)O)C(O[C@@H](O2)[C@@H]([C@@H]([C@@H](C2CO)O)O)O)C1O | + | SMILES c(c(OC)7)c(ccc7O)C=CC(=O)OC[C@H]([C@H](O)1)O[C@@H](Oc(c(c(c6)cc(OC)c(O)c6)5)cc(c([o+1]5)4)c(cc(c4)O)O[C@H](O3)[C@H]([C@@H](O)[C@@H](O)C3CO)O)C(O[C@@H](O2)[C@@H]([C@@H]([C@@H](C2CO)O)O)O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
68 74 0 0 0 0 0 0 0 0999 V2000
-1.8470 2.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8470 2.1137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2907 1.7925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7344 2.1137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7344 2.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2907 3.0773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1781 1.7925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3782 2.1137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3782 2.7561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1781 3.0773 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.9343 3.0771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5012 2.7498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0682 3.0771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0682 3.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5012 4.0592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9343 3.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4031 3.0771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9342 4.2318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2907 1.1504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7943 1.3930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2728 0.0789 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.7657 0.4666 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.9753 -0.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7222 -0.8295 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.2728 -1.1474 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.0981 -0.5361 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.4476 0.4838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5996 -0.5800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5773 -2.0999 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1976 -1.6808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1172 0.5812 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.4207 -0.0107 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.0295 0.1104 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.5802 0.0194 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.2512 0.4894 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.7064 0.3142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7987 0.0680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2592 -0.4951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5335 -0.2828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5600 -2.3149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4495 0.8762 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.0783 0.3863 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.5438 0.5941 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.0281 0.5997 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.4029 0.9745 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8705 0.7278 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.0404 0.9280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4287 -0.0750 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2376 0.0800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2221 -2.9001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5498 -3.4677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3447 -2.9001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6688 -3.4614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2957 -3.4614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6082 -2.9202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2331 -2.9202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5456 -3.4614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2331 -4.0027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6082 -4.0027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1699 -3.4614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7846 4.6353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2260 5.5326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5866 1.3563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1679 1.9417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1101 1.0015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9831 0.5137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8886 -4.2228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1742 -4.6353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
34 39 1 0 0 0 0
27 31 1 0 0 0 0
22 20 1 0 0 0 0
30 40 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 19 1 0 0 0 0
40 50 1 0 0 0 0
50 51 2 0 0 0 0
50 52 1 0 0 0 0
52 53 2 0 0 0 0
53 54 1 0 0 0 0
54 55 2 0 0 0 0
55 56 1 0 0 0 0
56 57 2 0 0 0 0
57 58 1 0 0 0 0
58 59 2 0 0 0 0
59 54 1 0 0 0 0
57 60 1 0 0 0 0
15 61 1 0 0 0 0
61 62 1 0 0 0 0
46 63 1 0 0 0 0
63 64 1 0 0 0 0
35 65 1 0 0 0 0
65 66 1 0 0 0 0
58 67 1 0 0 0 0
67 68 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 65 66
M SBL 4 1 71
M SMT 4 CH2OH
M SVB 4 71 3.9633 0.5103
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 63 64
M SBL 3 1 69
M SMT 3 CH2OH
M SVB 3 69 -3.2539 1.6001
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 67 68
M SBL 2 1 73
M SMT 2 OCH3
M SVB 2 73 -2.8886 -4.2228
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 61 62
M SBL 1 1 67
M SMT 1 OCH3
M SVB 1 67 1.7846 4.6353
S SKP 8
ID FL7AADGL0023
KNApSAcK_ID C00006871
NAME Peonidin 3-ferulyldiglucoside-5-glucoside
CAS_RN 144093-82-3
FORMULA C44H51O24
EXACTMASS 963.27702756
AVERAGEMASS 963.86134
SMILES c(c(OC)7)c(ccc7O)C=CC(=O)OC[C@H]([C@H](O)1)O[C@@H](Oc(c(c(c6)cc(OC)c(O)c6)5)cc(c([o+1]5)4)c(cc(c4)O)O[C@H](O3)[C@H]([C@@H](O)[C@@H](O)C3CO)O)C(O[C@@H](O2)[C@@H]([C@@H]([C@@H](C2CO)O)O)O)C1O
M END