Mol:FL7AAGGL0034
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 110120 0 0 0 0 0 0 0 0999 V2000 | + | 110120 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0902 -0.4675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0902 -0.4675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4892 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4892 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4892 0.5735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4892 0.5735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0902 0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0902 0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6912 0.5735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6912 0.5735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6912 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6912 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8882 -0.4675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8882 -0.4675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2871 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2871 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2871 0.5735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2871 0.5735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8882 0.9205 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -0.8882 0.9205 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3487 0.9406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3487 0.9406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9171 0.6123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9171 0.6123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4856 0.9406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4856 0.9406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4856 1.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4856 1.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9171 1.9252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9171 1.9252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3487 1.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3487 1.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9997 1.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9997 1.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2522 0.8974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2522 0.8974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0902 -1.0945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0902 -1.0945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0391 -0.7462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0391 -0.7462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9171 2.4530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9171 2.4530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0038 0.6414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0038 0.6414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9299 0.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9299 0.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3589 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3589 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6337 0.6245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6337 0.6245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8093 0.5934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8093 0.5934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3802 1.1890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3802 1.1890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1055 0.7956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1055 0.7956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3686 1.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3686 1.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8252 1.4069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8252 1.4069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2062 0.3704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2062 0.3704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7956 0.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7956 0.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3683 0.1416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3683 0.1416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0510 -0.6596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0510 -0.6596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5143 -1.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5143 -1.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3357 -1.2089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3357 -1.2089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0818 -1.5611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0818 -1.5611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6186 -0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6186 -0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7971 -1.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7971 -1.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6064 -0.7787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6064 -0.7787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2211 -0.5877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2211 -0.5877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9764 -1.2923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9764 -1.2923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6249 -1.4156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6249 -1.4156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0365 -1.8272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0365 -1.8272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7222 2.9179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7222 2.9179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3097 2.2034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3097 2.2034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1029 2.4301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1029 2.4301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9010 2.2210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9010 2.2210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3136 2.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3136 2.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5203 2.7088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5203 2.7088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1207 2.7568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1207 2.7568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7895 3.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7895 3.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7224 2.6994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7224 2.6994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4046 2.5117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4046 2.5117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9810 2.1789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9810 2.1789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1558 0.8347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1558 0.8347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5921 0.2323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5921 0.2323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4161 0.2734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4161 0.2734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1460 -0.1112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1460 -0.1112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7098 0.4912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7098 0.4912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8857 0.4502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8857 0.4502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5335 0.8141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5335 0.8141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2537 0.8435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2537 0.8435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0545 0.1687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0545 0.1687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7024 0.0579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7024 0.0579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1884 -0.3969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1884 -0.3969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6164 2.5458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6164 2.5458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6164 3.1658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6164 3.1658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2272 2.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2272 2.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8780 2.5688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8780 2.5688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4983 2.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4983 2.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4983 1.5448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4983 1.5448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0749 1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0749 1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6515 1.5448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6515 1.5448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6515 2.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6515 2.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0749 2.5435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0749 2.5435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0625 1.3075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0625 1.3075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9867 -0.1830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9867 -0.1830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9867 0.4262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9867 0.4262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6401 -0.5602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6401 -0.5602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2563 -0.2045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2563 -0.2045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8381 -0.5404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8381 -0.5404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8381 -1.1936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8381 -1.1936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4038 -1.5202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4038 -1.5202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9695 -1.1936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9695 -1.1936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9695 -0.5404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9695 -0.5404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4038 -0.2138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4038 -0.2138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.4073 -1.4464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.4073 -1.4464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3559 1.9376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3559 1.9376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3559 2.5851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3559 2.5851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.8850 1.4085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.8850 1.4085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.4842 2.0077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.4842 2.0077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -8.0910 1.4009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -8.0910 1.4009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -8.7469 1.5767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -8.7469 1.5767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -9.2270 1.0965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -9.2270 1.0965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -9.0513 0.4406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -9.0513 0.4406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -8.3954 0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -8.3954 0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.9152 0.7450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.9152 0.7450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -9.4073 0.0846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -9.4073 0.0846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8893 -1.8380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8893 -1.8380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8893 -1.2287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8893 -1.2287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5428 -2.2152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5428 -2.2152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1589 -1.8595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1589 -1.8595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7407 -2.1954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7407 -2.1954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7407 -2.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7407 -2.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3064 -3.1752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3064 -3.1752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8721 -2.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8721 -2.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8721 -2.1954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8721 -2.1954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3064 -1.8688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3064 -1.8688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3099 -3.1014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3099 -3.1014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 3 1 0 0 0 0 | + | 10 3 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 5 18 1 0 0 0 0 | + | 5 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 13 22 1 0 0 0 0 | + | 13 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 18 26 1 0 0 0 0 | + | 18 26 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 20 35 1 0 0 0 0 | + | 20 35 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 49 48 1 1 0 0 0 | + | 49 48 1 1 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 50 52 1 0 0 0 0 | + | 50 52 1 0 0 0 0 |
| − | 49 53 1 0 0 0 0 | + | 49 53 1 0 0 0 0 |
| − | 48 54 1 0 0 0 0 | + | 48 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 21 46 1 0 0 0 0 | + | 21 46 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 1 1 0 0 0 | + | 57 58 1 1 0 0 0 |
| − | 59 58 1 1 0 0 0 | + | 59 58 1 1 0 0 0 |
| − | 60 59 1 1 0 0 0 | + | 60 59 1 1 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 61 56 1 0 0 0 0 | + | 61 56 1 0 0 0 0 |
| − | 56 62 1 0 0 0 0 | + | 56 62 1 0 0 0 0 |
| − | 61 63 1 0 0 0 0 | + | 61 63 1 0 0 0 0 |
| − | 60 64 1 0 0 0 0 | + | 60 64 1 0 0 0 0 |
| − | 59 65 1 0 0 0 0 | + | 59 65 1 0 0 0 0 |
| − | 65 66 1 0 0 0 0 | + | 65 66 1 0 0 0 0 |
| − | 22 57 1 0 0 0 0 | + | 22 57 1 0 0 0 0 |
| − | 55 67 1 0 0 0 0 | + | 55 67 1 0 0 0 0 |
| − | 67 68 2 0 0 0 0 | + | 67 68 2 0 0 0 0 |
| − | 67 69 1 0 0 0 0 | + | 67 69 1 0 0 0 0 |
| − | 69 70 2 0 0 0 0 | + | 69 70 2 0 0 0 0 |
| − | 70 71 1 0 0 0 0 | + | 70 71 1 0 0 0 0 |
| − | 71 72 2 0 0 0 0 | + | 71 72 2 0 0 0 0 |
| − | 72 73 1 0 0 0 0 | + | 72 73 1 0 0 0 0 |
| − | 73 74 2 0 0 0 0 | + | 73 74 2 0 0 0 0 |
| − | 74 75 1 0 0 0 0 | + | 74 75 1 0 0 0 0 |
| − | 75 76 2 0 0 0 0 | + | 75 76 2 0 0 0 0 |
| − | 76 71 1 0 0 0 0 | + | 76 71 1 0 0 0 0 |
| − | 74 77 1 0 0 0 0 | + | 74 77 1 0 0 0 0 |
| − | 66 78 1 0 0 0 0 | + | 66 78 1 0 0 0 0 |
| − | 78 79 2 0 0 0 0 | + | 78 79 2 0 0 0 0 |
| − | 78 80 1 0 0 0 0 | + | 78 80 1 0 0 0 0 |
| − | 80 81 2 0 0 0 0 | + | 80 81 2 0 0 0 0 |
| − | 81 82 1 0 0 0 0 | + | 81 82 1 0 0 0 0 |
| − | 82 83 2 0 0 0 0 | + | 82 83 2 0 0 0 0 |
| − | 83 84 1 0 0 0 0 | + | 83 84 1 0 0 0 0 |
| − | 84 85 2 0 0 0 0 | + | 84 85 2 0 0 0 0 |
| − | 85 86 1 0 0 0 0 | + | 85 86 1 0 0 0 0 |
| − | 86 87 2 0 0 0 0 | + | 86 87 2 0 0 0 0 |
| − | 87 82 1 0 0 0 0 | + | 87 82 1 0 0 0 0 |
| − | 85 88 1 0 0 0 0 | + | 85 88 1 0 0 0 0 |
| − | 30 89 1 0 0 0 0 | + | 30 89 1 0 0 0 0 |
| − | 89 90 2 0 0 0 0 | + | 89 90 2 0 0 0 0 |
| − | 89 91 1 0 0 0 0 | + | 89 91 1 0 0 0 0 |
| − | 91 92 2 0 0 0 0 | + | 91 92 2 0 0 0 0 |
| − | 92 93 1 0 0 0 0 | + | 92 93 1 0 0 0 0 |
| − | 93 94 2 0 0 0 0 | + | 93 94 2 0 0 0 0 |
| − | 94 95 1 0 0 0 0 | + | 94 95 1 0 0 0 0 |
| − | 95 96 2 0 0 0 0 | + | 95 96 2 0 0 0 0 |
| − | 96 97 1 0 0 0 0 | + | 96 97 1 0 0 0 0 |
| − | 97 98 2 0 0 0 0 | + | 97 98 2 0 0 0 0 |
| − | 98 93 1 0 0 0 0 | + | 98 93 1 0 0 0 0 |
| − | 96 99 1 0 0 0 0 | + | 96 99 1 0 0 0 0 |
| − | 100101 2 0 0 0 0 | + | 100101 2 0 0 0 0 |
| − | 100102 1 0 0 0 0 | + | 100102 1 0 0 0 0 |
| − | 102103 2 0 0 0 0 | + | 102103 2 0 0 0 0 |
| − | 103104 1 0 0 0 0 | + | 103104 1 0 0 0 0 |
| − | 104105 2 0 0 0 0 | + | 104105 2 0 0 0 0 |
| − | 105106 1 0 0 0 0 | + | 105106 1 0 0 0 0 |
| − | 106107 2 0 0 0 0 | + | 106107 2 0 0 0 0 |
| − | 107108 1 0 0 0 0 | + | 107108 1 0 0 0 0 |
| − | 108109 2 0 0 0 0 | + | 108109 2 0 0 0 0 |
| − | 109104 1 0 0 0 0 | + | 109104 1 0 0 0 0 |
| − | 107110 1 0 0 0 0 | + | 107110 1 0 0 0 0 |
| − | 100 44 1 0 0 0 0 | + | 100 44 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAGGL0034 | + | ID FL7AAGGL0034 |
| − | KNApSAcK_ID C00011105 | + | KNApSAcK_ID C00011105 |
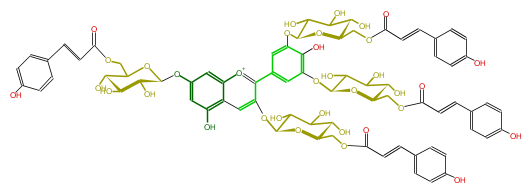
| − | NAME Delphinidin 3,7,3',5'-tetra(6-O-p-coumaroyl-beta-glucoside) | + | NAME Delphinidin 3,7,3',5'-tetra(6-O-p-coumaroyl-beta-glucoside) |
| − | CAS_RN 395680-65-6 | + | CAS_RN 395680-65-6 |
| − | FORMULA C75H75O35 | + | FORMULA C75H75O35 |
| − | EXACTMASS 1535.40888917 | + | EXACTMASS 1535.40888917 |
| − | AVERAGEMASS 1536.377 | + | AVERAGEMASS 1536.377 |
| − | SMILES c([o+1]3)(c9)c(c(cc9OC(C%11O)OC(C(C%11O)O)COC(=O)C=Cc(c%10)ccc(O)c%10)O)cc(c3c(c6)cc(c(O)c6OC(O7)C(O)C(C(C7COC(C=Cc(c8)ccc(c8)O)=O)O)O)OC(O4)C(O)C(C(C4COC(C=Cc(c5)ccc(c5)O)=O)O)O)OC(O1)C(C(O)C(O)C1COC(C=Cc(c2)ccc(O)c2)=O)O | + | SMILES c([o+1]3)(c9)c(c(cc9OC(C%11O)OC(C(C%11O)O)COC(=O)C=Cc(c%10)ccc(O)c%10)O)cc(c3c(c6)cc(c(O)c6OC(O7)C(O)C(C(C7COC(C=Cc(c8)ccc(c8)O)=O)O)O)OC(O4)C(O)C(C(C4COC(C=Cc(c5)ccc(c5)O)=O)O)O)OC(O1)C(C(O)C(O)C1COC(C=Cc(c2)ccc(O)c2)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
110120 0 0 0 0 0 0 0 0999 V2000
-2.0902 -0.4675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4892 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4892 0.5735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0902 0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6912 0.5735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6912 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8882 -0.4675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2871 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2871 0.5735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8882 0.9205 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.3487 0.9406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9171 0.6123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4856 0.9406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4856 1.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9171 1.9252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3487 1.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9997 1.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2522 0.8974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0902 -1.0945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0391 -0.7462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9171 2.4530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0038 0.6414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9299 0.8266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3589 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6337 0.6245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8093 0.5934 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3802 1.1890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1055 0.7956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3686 1.2742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8252 1.4069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.2062 0.3704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7956 0.2403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3683 0.1416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0510 -0.6596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5143 -1.2861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3357 -1.2089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0818 -1.5611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6186 -0.9345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7971 -1.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6064 -0.7787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2211 -0.5877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9764 -1.2923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6249 -1.4156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0365 -1.8272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7222 2.9179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3097 2.2034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1029 2.4301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9010 2.2210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3136 2.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5203 2.7088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1207 2.7568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7895 3.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7224 2.6994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4046 2.5117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9810 2.1789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1558 0.8347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5921 0.2323 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4161 0.2734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1460 -0.1112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7098 0.4912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8857 0.4502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5335 0.8141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2537 0.8435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0545 0.1687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7024 0.0579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1884 -0.3969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6164 2.5458 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6164 3.1658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2272 2.1931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.8780 2.5688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4983 2.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.4983 1.5448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0749 1.2119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6515 1.5448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.6515 2.2106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0749 2.5435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.0625 1.3075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9867 -0.1830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9867 0.4262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.6401 -0.5602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2563 -0.2045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.8381 -0.5404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.8381 -1.1936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.4038 -1.5202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9695 -1.1936 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.9695 -0.5404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.4038 -0.2138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.4073 -1.4464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.3559 1.9376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.3559 2.5851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.8850 1.4085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.4842 2.0077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-8.0910 1.4009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-8.7469 1.5767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-9.2270 1.0965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-9.0513 0.4406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-8.3954 0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.9152 0.7450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-9.4073 0.0846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8893 -1.8380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8893 -1.2287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5428 -2.2152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1589 -1.8595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7407 -2.1954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7407 -2.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3064 -3.1752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8721 -2.8486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.8721 -2.1954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3064 -1.8688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3099 -3.1014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 3 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
5 18 1 0 0 0 0
1 19 1 0 0 0 0
8 20 1 0 0 0 0
15 21 1 0 0 0 0
13 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
18 26 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
20 35 1 0 0 0 0
34 40 1 0 0 0 0
39 41 1 0 0 0 0
38 42 1 0 0 0 0
37 43 1 0 0 0 0
43 44 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
49 48 1 1 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
45 51 1 0 0 0 0
50 52 1 0 0 0 0
49 53 1 0 0 0 0
48 54 1 0 0 0 0
54 55 1 0 0 0 0
21 46 1 0 0 0 0
56 57 1 0 0 0 0
57 58 1 1 0 0 0
59 58 1 1 0 0 0
60 59 1 1 0 0 0
60 61 1 0 0 0 0
61 56 1 0 0 0 0
56 62 1 0 0 0 0
61 63 1 0 0 0 0
60 64 1 0 0 0 0
59 65 1 0 0 0 0
65 66 1 0 0 0 0
22 57 1 0 0 0 0
55 67 1 0 0 0 0
67 68 2 0 0 0 0
67 69 1 0 0 0 0
69 70 2 0 0 0 0
70 71 1 0 0 0 0
71 72 2 0 0 0 0
72 73 1 0 0 0 0
73 74 2 0 0 0 0
74 75 1 0 0 0 0
75 76 2 0 0 0 0
76 71 1 0 0 0 0
74 77 1 0 0 0 0
66 78 1 0 0 0 0
78 79 2 0 0 0 0
78 80 1 0 0 0 0
80 81 2 0 0 0 0
81 82 1 0 0 0 0
82 83 2 0 0 0 0
83 84 1 0 0 0 0
84 85 2 0 0 0 0
85 86 1 0 0 0 0
86 87 2 0 0 0 0
87 82 1 0 0 0 0
85 88 1 0 0 0 0
30 89 1 0 0 0 0
89 90 2 0 0 0 0
89 91 1 0 0 0 0
91 92 2 0 0 0 0
92 93 1 0 0 0 0
93 94 2 0 0 0 0
94 95 1 0 0 0 0
95 96 2 0 0 0 0
96 97 1 0 0 0 0
97 98 2 0 0 0 0
98 93 1 0 0 0 0
96 99 1 0 0 0 0
100101 2 0 0 0 0
100102 1 0 0 0 0
102103 2 0 0 0 0
103104 1 0 0 0 0
104105 2 0 0 0 0
105106 1 0 0 0 0
106107 2 0 0 0 0
107108 1 0 0 0 0
108109 2 0 0 0 0
109104 1 0 0 0 0
107110 1 0 0 0 0
100 44 1 0 0 0 0
S SKP 8
ID FL7AAGGL0034
KNApSAcK_ID C00011105
NAME Delphinidin 3,7,3',5'-tetra(6-O-p-coumaroyl-beta-glucoside)
CAS_RN 395680-65-6
FORMULA C75H75O35
EXACTMASS 1535.40888917
AVERAGEMASS 1536.377
SMILES c([o+1]3)(c9)c(c(cc9OC(C%11O)OC(C(C%11O)O)COC(=O)C=Cc(c%10)ccc(O)c%10)O)cc(c3c(c6)cc(c(O)c6OC(O7)C(O)C(C(C7COC(C=Cc(c8)ccc(c8)O)=O)O)O)OC(O4)C(O)C(C(C4COC(C=Cc(c5)ccc(c5)O)=O)O)O)OC(O1)C(C(O)C(O)C1COC(C=Cc(c2)ccc(O)c2)=O)O
M END