Mol:FL7AAGGL0066
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 81 88 0 0 0 0 0 0 0 0999 V2000 | + | 81 88 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.0555 2.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0555 2.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0555 1.6227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0555 1.6227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3411 1.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3411 1.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3734 1.6227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3734 1.6227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3734 2.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3734 2.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3411 2.8602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3411 2.8602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0879 1.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0879 1.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8023 1.6227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8023 1.6227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8023 2.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8023 2.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0879 2.8602 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 1.0879 2.8602 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5789 2.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5789 2.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2934 2.4835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2934 2.4835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0078 2.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0078 2.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0078 3.7210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0078 3.7210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2934 4.1335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2934 4.1335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5789 3.7210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5789 3.7210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6480 4.0906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6480 4.0906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6608 1.1270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6608 1.1270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6355 2.7825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6355 2.7825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3411 0.4475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3411 0.4475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2934 4.8647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2934 4.8647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6728 2.5121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6728 2.5121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0676 0.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0676 0.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6550 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6550 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8566 0.2546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8566 0.2546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0631 0.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0631 0.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4757 0.7425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4757 0.7425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2741 0.5335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2741 0.5335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4311 1.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4311 1.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5508 0.2770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5508 0.2770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1036 0.3045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1036 0.3045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0281 -0.3853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0281 -0.3853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0274 1.4635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0274 1.4635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2492 -1.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2492 -1.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0387 -1.3296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0387 -1.3296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4790 -0.6316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4790 -0.6316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2136 -0.2555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2136 -0.2555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4241 -0.0151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4241 -0.0151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9837 -0.7131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9837 -0.7131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4689 -0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4689 -0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2262 -1.7723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2262 -1.7723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6283 -1.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6283 -1.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7908 -1.2162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7908 -1.2162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7368 -3.1273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7368 -3.1273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5263 -3.3676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5263 -3.3676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9666 -2.6696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9666 -2.6696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7012 -2.2935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7012 -2.2935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9117 -2.0531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9117 -2.0531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4712 -2.7510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4712 -2.7510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9565 -2.4303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9565 -2.4303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4168 -2.5556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4168 -2.5556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7138 -3.8103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7138 -3.8103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0216 -3.4848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0216 -3.4848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2784 -3.2542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2784 -3.2542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0713 -3.4367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0713 -3.4367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3429 -4.1541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3429 -4.1541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8952 -3.4367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8952 -3.4367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3071 -4.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3071 -4.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1310 -4.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1310 -4.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5435 -3.4358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5435 -3.4358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3685 -3.4358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3685 -3.4358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7810 -4.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7810 -4.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3685 -4.8647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3685 -4.8647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5435 -4.8647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5435 -4.8647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6049 -4.1503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6049 -4.1503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9467 -2.8576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9467 -2.8576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6728 -2.8576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6728 -2.8576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6950 -0.6250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6950 -0.6250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3577 -1.2092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3577 -1.2092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7570 -1.9008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7570 -1.9008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4662 -1.2092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4662 -1.2092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8781 -1.9228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8781 -1.9228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7020 -1.9228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7020 -1.9228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1145 -1.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1145 -1.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9395 -1.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9395 -1.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3520 -1.9228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3520 -1.9228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9395 -2.6372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9395 -2.6372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1145 -2.6372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1145 -2.6372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1759 -1.9228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1759 -1.9228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5177 -0.6301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5177 -0.6301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2438 -0.6301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2438 -0.6301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 13 22 1 0 0 0 0 | + | 13 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 33 29 1 0 0 0 0 | + | 33 29 1 0 0 0 0 |
| − | 26 20 1 0 0 0 0 | + | 26 20 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 47 46 1 1 0 0 0 | + | 47 46 1 1 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 44 52 1 0 0 0 0 | + | 44 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 46 54 1 0 0 0 0 | + | 46 54 1 0 0 0 0 |
| − | 47 43 1 0 0 0 0 | + | 47 43 1 0 0 0 0 |
| − | 37 18 1 0 0 0 0 | + | 37 18 1 0 0 0 0 |
| − | 55 56 2 0 0 0 0 | + | 55 56 2 0 0 0 0 |
| − | 55 57 1 0 0 0 0 | + | 55 57 1 0 0 0 0 |
| − | 57 58 2 0 0 0 0 | + | 57 58 2 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 2 0 0 0 0 | + | 59 60 2 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 61 62 2 0 0 0 0 | + | 61 62 2 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 63 64 2 0 0 0 0 | + | 63 64 2 0 0 0 0 |
| − | 64 59 1 0 0 0 0 | + | 64 59 1 0 0 0 0 |
| − | 62 65 1 0 0 0 0 | + | 62 65 1 0 0 0 0 |
| − | 61 66 1 0 0 0 0 | + | 61 66 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | 68 69 1 0 0 0 0 | + | 68 69 1 0 0 0 0 |
| − | 69 70 2 0 0 0 0 | + | 69 70 2 0 0 0 0 |
| − | 69 71 1 0 0 0 0 | + | 69 71 1 0 0 0 0 |
| − | 71 72 2 0 0 0 0 | + | 71 72 2 0 0 0 0 |
| − | 72 73 1 0 0 0 0 | + | 72 73 1 0 0 0 0 |
| − | 73 74 2 0 0 0 0 | + | 73 74 2 0 0 0 0 |
| − | 74 75 1 0 0 0 0 | + | 74 75 1 0 0 0 0 |
| − | 75 76 2 0 0 0 0 | + | 75 76 2 0 0 0 0 |
| − | 76 77 1 0 0 0 0 | + | 76 77 1 0 0 0 0 |
| − | 77 78 2 0 0 0 0 | + | 77 78 2 0 0 0 0 |
| − | 78 73 1 0 0 0 0 | + | 78 73 1 0 0 0 0 |
| − | 76 79 1 0 0 0 0 | + | 76 79 1 0 0 0 0 |
| − | 75 80 1 0 0 0 0 | + | 75 80 1 0 0 0 0 |
| − | 80 81 1 0 0 0 0 | + | 80 81 1 0 0 0 0 |
| − | 51 55 1 0 0 0 0 | + | 51 55 1 0 0 0 0 |
| − | 68 40 1 0 0 0 0 | + | 68 40 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAGGL0066 | + | ID FL7AAGGL0066 |
| − | KNApSAcK_ID C00014817 | + | KNApSAcK_ID C00014817 |
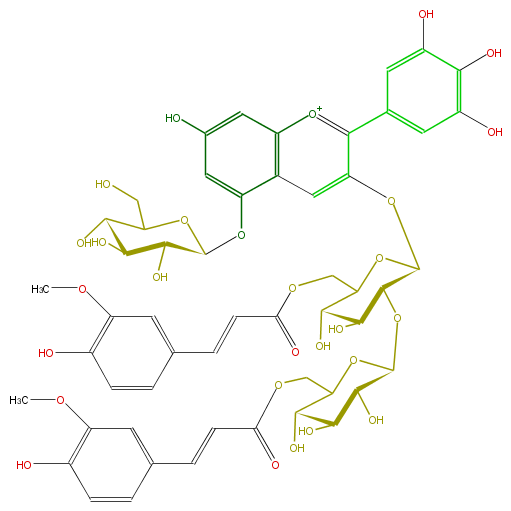
| − | NAME Delphinidin 3-(diferuloyl)sophoroside-5-glucoside | + | NAME Delphinidin 3-(diferuloyl)sophoroside-5-glucoside |
| − | CAS_RN 379232-06-1 | + | CAS_RN 379232-06-1 |
| − | FORMULA C53H57O28 | + | FORMULA C53H57O28 |
| − | EXACTMASS 1141.30363624 | + | EXACTMASS 1141.30363624 |
| − | AVERAGEMASS 1142.00288 | + | AVERAGEMASS 1142.00288 |
| − | SMILES Oc(c1)cc(OC(O8)C(C(O)C(O)C8CO)O)c(c3)c1[o+1]c(c3OC(O4)C(OC(O6)C(C(C(C6COC(=O)C=Cc(c7)cc(c(O)c7)OC)O)O)O)C(O)C(O)C4COC(=O)C=Cc(c5)ccc(O)c5OC)c(c2)cc(c(O)c2O)O | + | SMILES Oc(c1)cc(OC(O8)C(C(O)C(O)C8CO)O)c(c3)c1[o+1]c(c3OC(O4)C(OC(O6)C(C(C(C6COC(=O)C=Cc(c7)cc(c(O)c7)OC)O)O)O)C(O)C(O)C4COC(=O)C=Cc(c5)ccc(O)c5OC)c(c2)cc(c(O)c2O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
81 88 0 0 0 0 0 0 0 0999 V2000
-1.0555 2.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0555 1.6227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3411 1.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3734 1.6227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3734 2.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3411 2.8602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0879 1.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8023 1.6227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8023 2.4477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0879 2.8602 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
2.5789 2.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2934 2.4835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0078 2.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0078 3.7210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2934 4.1335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5789 3.7210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6480 4.0906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6608 1.1270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6355 2.7825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3411 0.4475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2934 4.8647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6728 2.5121 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0676 0.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6550 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8566 0.2546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0631 0.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4757 0.7425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2741 0.5335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4311 1.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5508 0.2770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1036 0.3045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0281 -0.3853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0274 1.4635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2492 -1.0893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0387 -1.3296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4790 -0.6316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2136 -0.2555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4241 -0.0151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9837 -0.7131 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4689 -0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2262 -1.7723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6283 -1.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7908 -1.2162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7368 -3.1273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5263 -3.3676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9666 -2.6696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7012 -2.2935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9117 -2.0531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4712 -2.7510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9565 -2.4303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4168 -2.5556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7138 -3.8103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0216 -3.4848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2784 -3.2542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0713 -3.4367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3429 -4.1541 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8952 -3.4367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3071 -4.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1310 -4.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5435 -3.4358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3685 -3.4358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7810 -4.1503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3685 -4.8647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5435 -4.8647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6049 -4.1503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9467 -2.8576 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6728 -2.8576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6950 -0.6250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3577 -1.2092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7570 -1.9008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4662 -1.2092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8781 -1.9228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7020 -1.9228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1145 -1.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9395 -1.2083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3520 -1.9228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9395 -2.6372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1145 -2.6372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1759 -1.9228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5177 -0.6301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2438 -0.6301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
8 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
15 21 1 0 0 0 0
13 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
33 29 1 0 0 0 0
26 20 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
39 40 1 0 0 0 0
34 41 1 0 0 0 0
35 42 1 0 0 0 0
36 43 1 0 0 0 0
44 45 1 1 0 0 0
45 46 1 1 0 0 0
47 46 1 1 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
44 52 1 0 0 0 0
45 53 1 0 0 0 0
46 54 1 0 0 0 0
47 43 1 0 0 0 0
37 18 1 0 0 0 0
55 56 2 0 0 0 0
55 57 1 0 0 0 0
57 58 2 0 0 0 0
58 59 1 0 0 0 0
59 60 2 0 0 0 0
60 61 1 0 0 0 0
61 62 2 0 0 0 0
62 63 1 0 0 0 0
63 64 2 0 0 0 0
64 59 1 0 0 0 0
62 65 1 0 0 0 0
61 66 1 0 0 0 0
66 67 1 0 0 0 0
68 69 1 0 0 0 0
69 70 2 0 0 0 0
69 71 1 0 0 0 0
71 72 2 0 0 0 0
72 73 1 0 0 0 0
73 74 2 0 0 0 0
74 75 1 0 0 0 0
75 76 2 0 0 0 0
76 77 1 0 0 0 0
77 78 2 0 0 0 0
78 73 1 0 0 0 0
76 79 1 0 0 0 0
75 80 1 0 0 0 0
80 81 1 0 0 0 0
51 55 1 0 0 0 0
68 40 1 0 0 0 0
S SKP 8
ID FL7AAGGL0066
KNApSAcK_ID C00014817
NAME Delphinidin 3-(diferuloyl)sophoroside-5-glucoside
CAS_RN 379232-06-1
FORMULA C53H57O28
EXACTMASS 1141.30363624
AVERAGEMASS 1142.00288
SMILES Oc(c1)cc(OC(O8)C(C(O)C(O)C8CO)O)c(c3)c1[o+1]c(c3OC(O4)C(OC(O6)C(C(C(C6COC(=O)C=Cc(c7)cc(c(O)c7)OC)O)O)O)C(O)C(O)C4COC(=O)C=Cc(c5)ccc(O)c5OC)c(c2)cc(c(O)c2O)O
M END