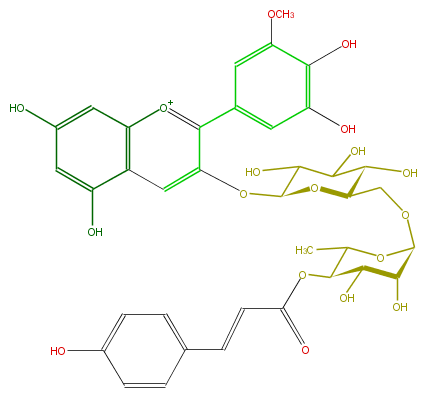
Mol:FL7AAHGL0012
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.2198 0.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2198 0.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2198 0.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2198 0.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5049 -0.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5049 -0.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7902 0.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7902 0.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7902 0.8505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7902 0.8505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5050 1.2631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5050 1.2631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0753 -0.3877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0753 -0.3877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3604 0.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3604 0.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3604 0.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3604 0.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0753 1.2632 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -1.0753 1.2632 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3541 1.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3541 1.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0828 0.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0828 0.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8112 1.2631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8112 1.2631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8112 2.1042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8112 2.1042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0828 2.5249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0828 2.5249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3541 2.1042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3541 2.1042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9344 1.2630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9344 1.2630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5657 2.5399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5657 2.5399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5050 -1.2128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5050 -1.2128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5263 -0.4488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5263 -0.4488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6047 -1.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6047 -1.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2600 -2.1364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2600 -2.1364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9228 -1.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9228 -1.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5896 -2.1364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5896 -2.1364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9344 -1.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9344 -1.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2716 -1.7288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2716 -1.7288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8597 -1.5823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8597 -1.5823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6283 0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6283 0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2838 -0.5271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2838 -0.5271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9465 -0.3377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9465 -0.3377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6132 -0.5271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6132 -0.5271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9581 0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9581 0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2953 -0.1194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2953 -0.1194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7914 -0.0211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7914 -0.0211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7360 0.4023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7360 0.4023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7824 -0.0346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7824 -0.0346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2466 -0.3428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2466 -0.3428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7714 -0.8772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7714 -0.8772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7098 -2.0662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7098 -2.0662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5185 -2.5379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5185 -2.5379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5896 -2.7181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5896 -2.7181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5397 0.8425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5397 0.8425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2942 -2.7603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2942 -2.7603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7622 -3.5711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7622 -3.5711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5027 -2.7640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5027 -2.7640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1090 -3.5889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1090 -3.5889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6361 -3.5924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6361 -3.5924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0490 -2.8868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0490 -2.8868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8744 -2.8906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8744 -2.8906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2873 -3.6000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2873 -3.6000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8744 -4.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8744 -4.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0489 -4.3017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0489 -4.3017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1120 -3.6036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1120 -3.6036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0828 3.1525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0828 3.1525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6501 4.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6501 4.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 24 41 1 0 0 0 0 | + | 24 41 1 0 0 0 0 |
| − | 29 20 1 0 0 0 0 | + | 29 20 1 0 0 0 0 |
| − | 13 42 1 0 0 0 0 | + | 13 42 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 43 39 1 0 0 0 0 | + | 43 39 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 15 54 1 0 0 0 0 | + | 15 54 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 54 55 | + | M SAL 1 2 54 55 |
| − | M SBL 1 1 60 | + | M SBL 1 1 60 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 60 0.0000 -0.6275 | + | M SBV 1 60 0.0000 -0.6275 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AAHGL0012 | + | ID FL7AAHGL0012 |
| − | FORMULA C37H39O18 | + | FORMULA C37H39O18 |
| − | EXACTMASS 771.213639444 | + | EXACTMASS 771.213639444 |
| − | AVERAGEMASS 771.69476 | + | AVERAGEMASS 771.69476 |
| − | SMILES c(c6)(c([o+1]c(c65)cc(O)cc5O)c(c4)cc(c(c(O)4)O)OC)OC(O1)C(O)C(O)C(C1COC(C(O)2)OC(C(OC(=O)C=Cc(c3)ccc(O)c3)C(O)2)C)O | + | SMILES c(c6)(c([o+1]c(c65)cc(O)cc5O)c(c4)cc(c(c(O)4)O)OC)OC(O1)C(O)C(O)C(C1COC(C(O)2)OC(C(OC(=O)C=Cc(c3)ccc(O)c3)C(O)2)C)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
-3.2198 0.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2198 0.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5049 -0.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7902 0.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7902 0.8505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5050 1.2631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0753 -0.3877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3604 0.0250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3604 0.8504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0753 1.2632 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
0.3541 1.2630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0828 0.8423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8112 1.2631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8112 2.1042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0828 2.5249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3541 2.1042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9344 1.2630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5657 2.5399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5050 -1.2128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5263 -0.4488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6047 -1.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2600 -2.1364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9228 -1.9470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5896 -2.1364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9344 -1.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2716 -1.7288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8597 -1.5823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6283 0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2838 -0.5271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9465 -0.3377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6132 -0.5271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9581 0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2953 -0.1194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7914 -0.0211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7360 0.4023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7824 -0.0346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2466 -0.3428 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7714 -0.8772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7098 -2.0662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5185 -2.5379 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5896 -2.7181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5397 0.8425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2942 -2.7603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7622 -3.5711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5027 -2.7640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1090 -3.5889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6361 -3.5924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0490 -2.8868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8744 -2.8906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2873 -3.6000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8744 -4.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0489 -4.3017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1120 -3.6036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0828 3.1525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6501 4.3055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
25 38 1 0 0 0 0
37 38 1 0 0 0 0
22 39 1 0 0 0 0
23 40 1 0 0 0 0
24 41 1 0 0 0 0
29 20 1 0 0 0 0
13 42 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
43 39 1 0 0 0 0
54 55 1 0 0 0 0
15 54 1 0 0 0 0
M CHG 1 10 1
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 54 55
M SBL 1 1 60
M SMT 1 OCH3
M SBV 1 60 0.0000 -0.6275
S SKP 5
ID FL7AAHGL0012
FORMULA C37H39O18
EXACTMASS 771.213639444
AVERAGEMASS 771.69476
SMILES c(c6)(c([o+1]c(c65)cc(O)cc5O)c(c4)cc(c(c(O)4)O)OC)OC(O1)C(O)C(O)C(C1COC(C(O)2)OC(C(OC(=O)C=Cc(c3)ccc(O)c3)C(O)2)C)O
M END