Mol:FLIA1CGS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4830 1.0715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4830 1.0715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0733 0.7503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0733 0.7503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6296 1.0715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6296 1.0715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1857 0.7504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1857 0.7504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1857 0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1857 0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7624 -0.2485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7624 -0.2485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3392 0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3392 0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3392 0.7504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3392 0.7504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7624 1.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7624 1.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0735 0.1083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0735 0.1083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6296 -0.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6296 -0.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7624 -0.9140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7624 -0.9140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9155 -0.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9155 -0.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9155 -0.8849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9155 -0.8849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4669 -1.2032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4669 -1.2032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0182 -0.8849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0182 -0.8849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0182 -0.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0182 -0.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4669 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4669 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5869 0.8677 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.5869 0.8677 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.2407 0.4107 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.2407 0.4107 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.7421 0.6046 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.7421 0.6046 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.2224 0.5989 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.2224 0.5989 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6106 0.9595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6106 0.9595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1199 0.7766 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.1199 0.7766 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.9739 1.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9739 1.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5675 -0.0195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5675 -0.0195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4564 0.1250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4564 0.1250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7294 -1.8074 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.7294 -1.8074 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.1568 -1.8360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.1568 -1.8360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.9848 -1.3294 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.9848 -1.3294 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.6464 -0.9350 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6464 -0.9350 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.1721 -1.0005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1721 -1.0005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3594 -1.5083 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.3594 -1.5083 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.0453 -2.1235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0453 -2.1235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0372 -2.3628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0372 -2.3628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4338 -1.4188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4338 -1.4188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3895 1.5037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3895 1.5037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7513 2.2736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7513 2.2736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8842 -1.3849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8842 -1.3849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7502 -1.8849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7502 -1.8849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5696 0.0701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5696 0.0701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2840 -0.3424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2840 -0.3424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0897 -1.2476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0897 -1.2476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8519 -1.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8519 -1.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 24 37 1 0 0 0 0 | + | 24 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 16 39 1 0 0 0 0 | + | 16 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 17 41 1 0 0 0 0 | + | 17 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 33 43 1 0 0 0 0 | + | 33 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 43 44 | + | M SAL 4 2 43 44 |
| − | M SBL 4 1 47 | + | M SBL 4 1 47 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 47 -4.6995 0.7612 | + | M SVB 4 47 -4.6995 0.7612 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 37 38 | + | M SAL 3 2 37 38 |
| − | M SBL 3 1 41 | + | M SBL 3 1 41 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 41 -2.3895 1.5037 | + | M SVB 3 41 -2.3895 1.5037 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 45 4.5696 0.0701 | + | M SVB 2 45 4.5696 0.0701 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 43 4.2123 -1.0912 | + | M SVB 1 43 4.2123 -1.0912 |
| − | S SKP 8 | + | S SKP 8 |
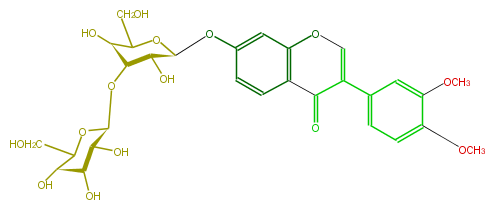
| − | ID FLIA1CGS0005 | + | ID FLIA1CGS0005 |
| − | KNApSAcK_ID C00010092 | + | KNApSAcK_ID C00010092 |
| − | NAME Cladrin 7-O-laminaribioside | + | NAME Cladrin 7-O-laminaribioside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C29H34O15 | + | FORMULA C29H34O15 |
| − | EXACTMASS 622.189770418 | + | EXACTMASS 622.189770418 |
| − | AVERAGEMASS 622.5712599999999 | + | AVERAGEMASS 622.5712599999999 |
| − | SMILES [C@H](O[C@H]([C@@H]2O)[C@@H](O)C(CO)O[C@H]2Oc(c3)ccc(C4=O)c3OC=C4c(c5)ccc(c(OC)5)OC)(O1)[C@H]([C@H]([C@H](C1CO)O)O)O | + | SMILES [C@H](O[C@H]([C@@H]2O)[C@@H](O)C(CO)O[C@H]2Oc(c3)ccc(C4=O)c3OC=C4c(c5)ccc(c(OC)5)OC)(O1)[C@H]([C@H]([C@H](C1CO)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-0.4830 1.0715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0733 0.7503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6296 1.0715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1857 0.7504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1857 0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7624 -0.2485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3392 0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3392 0.7504 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7624 1.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0735 0.1083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6296 -0.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7624 -0.9140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9155 -0.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9155 -0.8849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4669 -1.2032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0182 -0.8849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0182 -0.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4669 0.0701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5869 0.8677 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.2407 0.4107 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.7421 0.6046 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.2224 0.5989 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6106 0.9595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1199 0.7766 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.9739 1.0912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5675 -0.0195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4564 0.1250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7294 -1.8074 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.1568 -1.8360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.9848 -1.3294 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.6464 -0.9350 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.1721 -1.0005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3594 -1.5083 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.0453 -2.1235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0372 -2.3628 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4338 -1.4188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3895 1.5037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7513 2.2736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8842 -1.3849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7502 -1.8849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5696 0.0701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2840 -0.3424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0897 -1.2476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8519 -1.8949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 1 1 0 0 0 0
28 29 1 1 0 0 0
29 30 1 1 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 26 1 0 0 0 0
24 37 1 0 0 0 0
37 38 1 0 0 0 0
16 39 1 0 0 0 0
39 40 1 0 0 0 0
17 41 1 0 0 0 0
41 42 1 0 0 0 0
33 43 1 0 0 0 0
43 44 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 43 44
M SBL 4 1 47
M SMT 4 CH2OH
M SVB 4 47 -4.6995 0.7612
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 37 38
M SBL 3 1 41
M SMT 3 CH2OH
M SVB 3 41 -2.3895 1.5037
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 45
M SMT 2 OCH3
M SVB 2 45 4.5696 0.0701
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 43
M SMT 1 OCH3
M SVB 1 43 4.2123 -1.0912
S SKP 8
ID FLIA1CGS0005
KNApSAcK_ID C00010092
NAME Cladrin 7-O-laminaribioside
CAS_RN -
FORMULA C29H34O15
EXACTMASS 622.189770418
AVERAGEMASS 622.5712599999999
SMILES [C@H](O[C@H]([C@@H]2O)[C@@H](O)C(CO)O[C@H]2Oc(c3)ccc(C4=O)c3OC=C4c(c5)ccc(c(OC)5)OC)(O1)[C@H]([C@H]([C@H](C1CO)O)O)O
M END