Mol:FLIA1LNI0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 30 32 0 0 0 0 0 0 0 0999 V2000 | + | 30 32 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1832 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1832 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1832 -0.4773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1832 -0.4773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6269 -0.7985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6269 -0.7985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0706 -0.4773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0706 -0.4773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0706 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0706 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6269 0.4862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6269 0.4862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5143 -0.7985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5143 -0.7985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0420 -0.4773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0420 -0.4773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0420 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0420 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5143 0.4862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5143 0.4862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5981 -0.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5981 -0.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5981 -1.4852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5981 -1.4852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1929 -1.8286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1929 -1.8286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7877 -1.4852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7877 -1.4852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7877 -0.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7877 -0.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1929 -0.4550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1929 -0.4550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5143 -1.4406 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5143 -1.4406 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6269 1.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6269 1.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1830 1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1830 1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1830 2.0915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1830 2.0915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7391 2.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7391 2.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6269 2.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6269 2.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7755 -2.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7755 -2.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4900 -2.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4900 -2.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6537 -1.9852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6537 -1.9852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5198 -2.4852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5198 -2.4852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5404 0.7838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5404 0.7838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0403 1.6499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0403 1.6499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4900 0.1291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4900 0.1291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2045 -0.2834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2045 -0.2834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 11 2 0 0 0 0 | + | 16 11 2 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 2 0 0 0 0 | + | 19 20 2 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 13 23 1 0 0 0 0 | + | 13 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 14 25 1 0 0 0 0 | + | 14 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 1 27 1 0 0 0 0 | + | 1 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 16 29 1 0 0 0 0 | + | 16 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 29 30 | + | M SAL 4 2 29 30 |
| − | M SBL 4 1 31 | + | M SBL 4 1 31 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 31 1.49 0.1291 | + | M SVB 4 31 1.49 0.1291 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 27 28 | + | M SAL 3 2 27 28 |
| − | M SBL 3 1 29 | + | M SBL 3 1 29 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 29 -2.5404 0.7838 | + | M SVB 3 29 -2.5404 0.7838 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 25 26 | + | M SAL 2 2 25 26 |
| − | M SBL 2 1 27 | + | M SBL 2 1 27 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 27 2.0246 -1.6914 | + | M SVB 2 27 2.0246 -1.6914 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 23 24 | + | M SAL 1 2 23 24 |
| − | M SBL 1 1 25 | + | M SBL 1 1 25 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 25 0.7755 -2.0001 | + | M SVB 1 25 0.7755 -2.0001 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIA1LNI0005 | + | ID FLIA1LNI0005 |
| − | KNApSAcK_ID C00009884 | + | KNApSAcK_ID C00009884 |
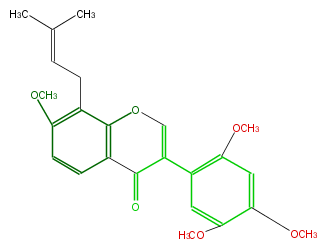
| − | NAME Prebarbigerone;7,2',4',5'-Tetramethoxy-8-prenylisoflavone | + | NAME Prebarbigerone;7,2',4',5'-Tetramethoxy-8-prenylisoflavone |
| − | CAS_RN 130286-70-3 | + | CAS_RN 130286-70-3 |
| − | FORMULA C24H26O6 | + | FORMULA C24H26O6 |
| − | EXACTMASS 410.172938564 | + | EXACTMASS 410.172938564 |
| − | AVERAGEMASS 410.45964 | + | AVERAGEMASS 410.45964 |
| − | SMILES O(C=2)c(c(CC=C(C)C)1)c(C(=O)C2c(c3)c(OC)cc(OC)c3OC)ccc1OC | + | SMILES O(C=2)c(c(CC=C(C)C)1)c(C(=O)C2c(c3)c(OC)cc(OC)c3OC)ccc1OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
30 32 0 0 0 0 0 0 0 0999 V2000
-2.1832 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1832 -0.4773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6269 -0.7985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0706 -0.4773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0706 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6269 0.4862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5143 -0.7985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0420 -0.4773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0420 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5143 0.4862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5981 -0.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5981 -1.4852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1929 -1.8286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7877 -1.4852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7877 -0.7984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1929 -0.4550 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5143 -1.4406 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6269 1.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1830 1.4494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1830 2.0915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7391 2.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6269 2.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7755 -2.0001 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4900 -2.4126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6537 -1.9852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5198 -2.4852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5404 0.7838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0403 1.6499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4900 0.1291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2045 -0.2834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
8 11 1 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 11 2 0 0 0 0
1 2 1 0 0 0 0
7 17 2 0 0 0 0
6 18 1 0 0 0 0
18 19 1 0 0 0 0
19 20 2 0 0 0 0
20 21 1 0 0 0 0
20 22 1 0 0 0 0
13 23 1 0 0 0 0
23 24 1 0 0 0 0
14 25 1 0 0 0 0
25 26 1 0 0 0 0
1 27 1 0 0 0 0
27 28 1 0 0 0 0
16 29 1 0 0 0 0
29 30 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 29 30
M SBL 4 1 31
M SMT 4 OCH3
M SVB 4 31 1.49 0.1291
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 27 28
M SBL 3 1 29
M SMT 3 OCH3
M SVB 3 29 -2.5404 0.7838
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 25 26
M SBL 2 1 27
M SMT 2 OCH3
M SVB 2 27 2.0246 -1.6914
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 23 24
M SBL 1 1 25
M SMT 1 OCH3
M SVB 1 25 0.7755 -2.0001
S SKP 8
ID FLIA1LNI0005
KNApSAcK_ID C00009884
NAME Prebarbigerone;7,2',4',5'-Tetramethoxy-8-prenylisoflavone
CAS_RN 130286-70-3
FORMULA C24H26O6
EXACTMASS 410.172938564
AVERAGEMASS 410.45964
SMILES O(C=2)c(c(CC=C(C)C)1)c(C(=O)C2c(c3)c(OC)cc(OC)c3OC)ccc1OC
M END