Mol:FLIAABGS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.9448 0.5739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9448 0.5739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2286 0.1604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2286 0.1604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4876 0.5739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4876 0.5739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2036 0.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2036 0.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2036 -0.6968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2036 -0.6968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9461 -1.1256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9461 -1.1256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6886 -0.6968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6886 -0.6968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6886 0.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6886 0.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9461 0.5892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9461 0.5892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2283 -0.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2283 -0.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4876 -1.0795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4876 -1.0795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9461 -1.7837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9461 -1.7837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4307 -1.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4307 -1.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4307 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4307 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1405 -2.3547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1405 -2.3547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8504 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8504 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8504 -1.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8504 -1.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1405 -0.7154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1405 -0.7154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6371 0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6371 0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0754 -0.0768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0754 -0.0768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4334 0.1729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4334 0.1729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6364 0.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6364 0.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2641 0.6298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2641 0.6298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9198 0.3943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9198 0.3943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4367 0.3648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4367 0.3648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8513 -0.0295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8513 -0.0295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7269 -0.3214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7269 -0.3214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5025 0.8661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5025 0.8661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5133 1.4760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5133 1.4760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4876 -1.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4876 -1.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7302 2.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7302 2.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7046 2.3571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7046 2.3571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3250 1.8559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3250 1.8559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2046 1.2904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2046 1.2904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0908 1.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0908 1.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7046 2.9404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7046 2.9404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7302 2.9201 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7302 2.9201 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4596 -2.2966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4596 -2.2966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5747 -2.9404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5747 -2.9404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7046 1.9088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7046 1.9088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.5747 2.5181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.5747 2.5181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 29 28 1 0 0 0 0 | + | 29 28 1 0 0 0 0 |
| − | 11 30 1 0 0 0 0 | + | 11 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 31 1 0 0 0 0 | + | 35 31 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 35 29 1 0 0 0 0 | + | 35 29 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 16 38 1 0 0 0 0 | + | 16 38 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 38 39 | + | M SAL 1 2 38 39 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 43 -0.6092 0.3517 | + | M SBV 1 43 -0.6092 0.3517 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 40 41 | + | M SAL 2 2 40 41 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 45 0.0000 0.4483 | + | M SBV 2 45 0.0000 0.4483 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FLIAABGS0005 | + | ID FLIAABGS0005 |
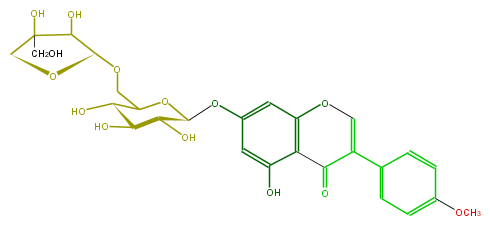
| − | FORMULA C27H30O14 | + | FORMULA C27H30O14 |
| − | EXACTMASS 578.163555668 | + | EXACTMASS 578.163555668 |
| − | AVERAGEMASS 578.5187000000001 | + | AVERAGEMASS 578.5187000000001 |
| − | SMILES C(C1OCC(O2)C(O)C(C(O)C2Oc(c5)cc(c(c54)C(=O)C(=CO4)c(c3)ccc(OC)c3)O)O)(C(CO)(CO1)O)O | + | SMILES C(C1OCC(O2)C(O)C(C(O)C2Oc(c5)cc(c(c54)C(=O)C(=CO4)c(c3)ccc(OC)c3)O)O)(C(CO)(CO1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-0.9448 0.5739 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2286 0.1604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4876 0.5739 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2036 0.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2036 -0.6968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9461 -1.1256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6886 -0.6968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6886 0.1605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9461 0.5892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2283 -0.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4876 -1.0795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9461 -1.7837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4307 -1.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4307 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1405 -2.3547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8504 -1.9449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8504 -1.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1405 -0.7154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6371 0.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0754 -0.0768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4334 0.1729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6364 0.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2641 0.6298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9198 0.3943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4367 0.3648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8513 -0.0295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7269 -0.3214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5025 0.8661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5133 1.4760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4876 -1.7625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7302 2.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7046 2.3571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.3250 1.8559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2046 1.2904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0908 1.8679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7046 2.9404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7302 2.9201 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4596 -2.2966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.5747 -2.9404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7046 1.9088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.5747 2.5181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
24 28 1 0 0 0 0
22 1 1 0 0 0 0
29 28 1 0 0 0 0
11 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 31 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
35 29 1 0 0 0 0
38 39 1 0 0 0 0
16 38 1 0 0 0 0
40 41 1 0 0 0 0
32 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 38 39
M SBL 1 1 43
M SMT 1 OCH3
M SBV 1 43 -0.6092 0.3517
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 40 41
M SBL 2 1 45
M SMT 2 CH2OH
M SBV 2 45 0.0000 0.4483
S SKP 5
ID FLIAABGS0005
FORMULA C27H30O14
EXACTMASS 578.163555668
AVERAGEMASS 578.5187000000001
SMILES C(C1OCC(O2)C(O)C(C(O)C2Oc(c5)cc(c(c54)C(=O)C(=CO4)c(c3)ccc(OC)c3)O)O)(C(CO)(CO1)O)O
M END