Mol:FLIAEGGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 37 40 0 0 0 0 0 0 0 0999 V2000 | + | 37 40 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4594 1.1322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4594 1.1322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9031 0.8111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9031 0.8111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3468 1.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3468 1.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2093 0.8112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2093 0.8112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2093 0.1452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2093 0.1452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7861 -0.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7861 -0.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3628 0.1452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3628 0.1452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3628 0.8112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3628 0.8112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7861 1.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7861 1.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9029 0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9029 0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3468 -0.1520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3468 -0.1520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7861 -0.8533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7861 -0.8533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9391 -0.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9391 -0.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9391 -0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9391 -0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4905 -1.1425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4905 -1.1425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0418 -0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0418 -0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0418 -0.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0418 -0.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4905 0.1308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4905 0.1308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5633 0.9285 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.5633 0.9285 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.2170 0.4714 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.2170 0.4714 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.7184 0.6653 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7184 0.6653 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1988 0.6596 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1988 0.6596 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.5869 1.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5869 1.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0963 0.8373 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.0963 0.8373 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.9503 1.1520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9503 1.1520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5439 0.0412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5439 0.0412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4328 0.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4328 0.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3573 -0.7927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3573 -0.7927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5932 0.1308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5932 0.1308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6173 0.2611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6173 0.2611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6173 -0.5638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6173 -0.5638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9078 -1.3242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9078 -1.3242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7738 -1.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7738 -1.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2957 1.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2957 1.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2510 2.0096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2510 2.0096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0517 -1.3016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0517 -1.3016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7662 -1.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7662 -1.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 11 28 1 0 0 0 0 | + | 11 28 1 0 0 0 0 |
| − | 17 29 1 0 0 0 0 | + | 17 29 1 0 0 0 0 |
| − | 10 30 1 0 0 0 0 | + | 10 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 16 32 1 0 0 0 0 | + | 16 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 24 34 1 0 0 0 0 | + | 24 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 15 36 1 0 0 0 0 | + | 15 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 34 35 | + | M SAL 4 2 34 35 |
| − | M SBL 4 1 37 | + | M SBL 4 1 37 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 37 -3.2957 1.7141 | + | M SVB 4 37 -3.2957 1.7141 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 36 37 | + | M SAL 3 2 36 37 |
| − | M SBL 3 1 39 | + | M SBL 3 1 39 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 39 2.0517 -1.3016 | + | M SVB 3 39 2.0517 -1.3016 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 32 33 | + | M SAL 2 2 32 33 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 35 3.2359 -1.0304 | + | M SVB 2 35 3.2359 -1.0304 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 33 -1.6173 0.2611 | + | M SVB 1 33 -1.6173 0.2611 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIAEGGS0001 | + | ID FLIAEGGS0001 |
| − | KNApSAcK_ID C00010137 | + | KNApSAcK_ID C00010137 |
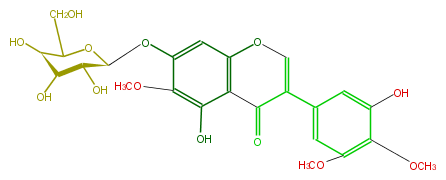
| − | NAME Irigenin 7-O-glucoside;Iridin | + | NAME Irigenin 7-O-glucoside;Iridin |
| − | CAS_RN 491-74-7 | + | CAS_RN 491-74-7 |
| − | FORMULA C24H26O13 | + | FORMULA C24H26O13 |
| − | EXACTMASS 522.137340918 | + | EXACTMASS 522.137340918 |
| − | AVERAGEMASS 522.45544 | + | AVERAGEMASS 522.45544 |
| − | SMILES [C@@H](Oc(c2OC)cc(O4)c(C(=O)C(=C4)c(c3)cc(c(OC)c(OC)3)O)c2O)([C@H]1O)OC(CO)[C@H](O)[C@@H]1O | + | SMILES [C@@H](Oc(c2OC)cc(O4)c(C(=O)C(=C4)c(c3)cc(c(OC)c(OC)3)O)c2O)([C@H]1O)OC(CO)[C@H](O)[C@@H]1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
37 40 0 0 0 0 0 0 0 0999 V2000
-1.4594 1.1322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9031 0.8111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3468 1.1322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2093 0.8112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2093 0.1452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7861 -0.1878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3628 0.1452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3628 0.8112 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7861 1.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9029 0.1691 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3468 -0.1520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7861 -0.8533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9391 -0.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9391 -0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4905 -1.1425 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0418 -0.8242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0418 -0.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4905 0.1308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5633 0.9285 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.2170 0.4714 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.7184 0.6653 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1988 0.6596 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.5869 1.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0963 0.8373 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.9503 1.1520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5439 0.0412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4328 0.1857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3573 -0.7927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5932 0.1308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6173 0.2611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6173 -0.5638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9078 -1.3242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7738 -1.8243 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2957 1.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2510 2.0096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0517 -1.3016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7662 -1.7141 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 1 1 0 0 0 0
11 28 1 0 0 0 0
17 29 1 0 0 0 0
10 30 1 0 0 0 0
30 31 1 0 0 0 0
16 32 1 0 0 0 0
32 33 1 0 0 0 0
24 34 1 0 0 0 0
34 35 1 0 0 0 0
15 36 1 0 0 0 0
36 37 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 34 35
M SBL 4 1 37
M SMT 4 CH2OH
M SVB 4 37 -3.2957 1.7141
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 36 37
M SBL 3 1 39
M SMT 3 OCH3
M SVB 3 39 2.0517 -1.3016
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 32 33
M SBL 2 1 35
M SMT 2 OCH3
M SVB 2 35 3.2359 -1.0304
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 33
M SMT 1 OCH3
M SVB 1 33 -1.6173 0.2611
S SKP 8
ID FLIAEGGS0001
KNApSAcK_ID C00010137
NAME Irigenin 7-O-glucoside;Iridin
CAS_RN 491-74-7
FORMULA C24H26O13
EXACTMASS 522.137340918
AVERAGEMASS 522.45544
SMILES [C@@H](Oc(c2OC)cc(O4)c(C(=O)C(=C4)c(c3)cc(c(OC)c(OC)3)O)c2O)([C@H]1O)OC(CO)[C@H](O)[C@@H]1O
M END