Mol:FLIAFLGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 53 0 0 0 0 0 0 0 0999 V2000 | + | 49 53 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.0466 0.9672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0466 0.9672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5097 0.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5097 0.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0660 0.9672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0660 0.9672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6221 0.6462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6221 0.6462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6221 -0.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6221 -0.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1989 -0.3528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1989 -0.3528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7756 -0.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7756 -0.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7756 0.6462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7756 0.6462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1989 0.9792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1989 0.9792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5099 0.0041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5099 0.0041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0660 -0.3170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0660 -0.3170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1989 -1.0183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1989 -1.0183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3519 -0.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3519 -0.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3519 -0.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3519 -0.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9033 -1.3075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9033 -1.3075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4546 -0.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4546 -0.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4546 -0.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4546 -0.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9033 -0.0342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9033 -0.0342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1505 0.7635 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.1505 0.7635 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.8042 0.3064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.8042 0.3064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.3056 0.5003 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.3056 0.5003 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.7860 0.4946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.7860 0.4946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.1741 0.8552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1741 0.8552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6834 0.6723 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.6834 0.6723 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.5956 0.8025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5956 0.8025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9187 -0.2217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9187 -0.2217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0200 0.0207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0200 0.0207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0453 1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0453 1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0556 -0.9577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0556 -0.9577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9761 1.0935 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.9761 1.0935 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -4.6299 0.6364 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.6299 0.6364 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -4.1313 0.8303 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.1313 0.8303 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.6116 0.8246 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.6116 0.8246 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.9998 1.1852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9998 1.1852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5091 1.0023 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -4.5091 1.0023 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -5.3631 1.3170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3631 1.3170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9568 0.2063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9568 0.2063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8456 0.3507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8456 0.3507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0386 1.3976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0386 1.3976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3441 1.5403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3441 1.5403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7807 2.4400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7807 2.4400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4645 -1.4666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4645 -1.4666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1790 -1.8791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1790 -1.8791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1790 0.5375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1790 0.5375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8934 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8934 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3206 -1.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3206 -1.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1866 -1.9893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1866 -1.9893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7085 1.8791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7085 1.8791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6638 2.1746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6638 2.1746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 2 10 1 0 0 0 0 | + | 2 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 5 1 0 0 0 0 | + | 11 5 1 0 0 0 0 |
| − | 6 12 2 0 0 0 0 | + | 6 12 2 0 0 0 0 |
| − | 7 13 1 0 0 0 0 | + | 7 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 22 1 1 0 0 0 0 | + | 22 1 1 0 0 0 0 |
| − | 11 29 1 0 0 0 0 | + | 11 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 39 28 1 0 0 0 0 | + | 39 28 1 0 0 0 0 |
| − | 3 40 1 0 0 0 0 | + | 3 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 15 42 1 0 0 0 0 | + | 15 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 18 44 1 0 0 0 0 | + | 18 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 16 46 1 0 0 0 0 | + | 16 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 35 48 1 0 0 0 0 | + | 35 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 48 49 | + | M SAL 5 2 48 49 |
| − | M SBL 5 1 52 | + | M SBL 5 1 52 |
| − | M SMT 5 CH2OH | + | M SMT 5 CH2OH |
| − | M SVB 5 52 -4.7085 1.8791 | + | M SVB 5 52 -4.7085 1.8791 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 46 47 | + | M SAL 4 2 46 47 |
| − | M SBL 4 1 50 | + | M SBL 4 1 50 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 50 4.6487 -1.1954 | + | M SVB 4 50 4.6487 -1.1954 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 44 45 | + | M SAL 3 2 44 45 |
| − | M SBL 3 1 48 | + | M SBL 3 1 48 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 48 4.179 0.5375 | + | M SVB 3 48 4.179 0.5375 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 46 3.4645 -1.4666 | + | M SVB 2 46 3.4645 -1.4666 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 44 1.3441 1.5403 | + | M SVB 1 44 1.3441 1.5403 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIAFLGS0002 | + | ID FLIAFLGS0002 |
| − | KNApSAcK_ID C00010140 | + | KNApSAcK_ID C00010140 |
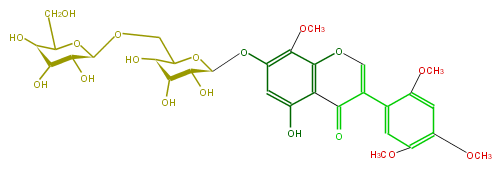
| − | NAME Isocaviunin 7-O-gentiobioside | + | NAME Isocaviunin 7-O-gentiobioside |
| − | CAS_RN 74517-73-0 | + | CAS_RN 74517-73-0 |
| − | FORMULA C31H38O18 | + | FORMULA C31H38O18 |
| − | EXACTMASS 698.205814412 | + | EXACTMASS 698.205814412 |
| − | AVERAGEMASS 698.62262 | + | AVERAGEMASS 698.62262 |
| − | SMILES C(CO[C@@H]([C@@H](O)5)OC([C@H](O)[C@@H]5O)CO)(O1)[C@H](O)[C@H](O)[C@H](O)[C@@H]1Oc(c4)c(c(c3c4O)OC=C(C3=O)c(c2OC)cc(OC)c(OC)c2)OC | + | SMILES C(CO[C@@H]([C@@H](O)5)OC([C@H](O)[C@@H]5O)CO)(O1)[C@H](O)[C@H](O)[C@H](O)[C@@H]1Oc(c4)c(c(c3c4O)OC=C(C3=O)c(c2OC)cc(OC)c(OC)c2)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 53 0 0 0 0 0 0 0 0999 V2000
-0.0466 0.9672 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5097 0.6461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0660 0.9672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6221 0.6462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6221 -0.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1989 -0.3528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7756 -0.0198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7756 0.6462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1989 0.9792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5099 0.0041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0660 -0.3170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1989 -1.0183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3519 -0.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3519 -0.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9033 -1.3075 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4546 -0.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4546 -0.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9033 -0.0342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1505 0.7635 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.8042 0.3064 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.3056 0.5003 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.7860 0.4946 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.1741 0.8552 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6834 0.6723 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.5956 0.8025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9187 -0.2217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0200 0.0207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0453 1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0556 -0.9577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9761 1.0935 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-4.6299 0.6364 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-4.1313 0.8303 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.6116 0.8246 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.9998 1.1852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5091 1.0023 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-5.3631 1.3170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9568 0.2063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8456 0.3507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0386 1.3976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3441 1.5403 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7807 2.4400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4645 -1.4666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1790 -1.8791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1790 0.5375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8934 0.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3206 -1.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1866 -1.9893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7085 1.8791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6638 2.1746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
2 10 1 0 0 0 0
10 11 2 0 0 0 0
11 5 1 0 0 0 0
6 12 2 0 0 0 0
7 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
24 28 1 0 0 0 0
22 1 1 0 0 0 0
11 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
39 28 1 0 0 0 0
3 40 1 0 0 0 0
40 41 1 0 0 0 0
15 42 1 0 0 0 0
42 43 1 0 0 0 0
18 44 1 0 0 0 0
44 45 1 0 0 0 0
16 46 1 0 0 0 0
46 47 1 0 0 0 0
35 48 1 0 0 0 0
48 49 1 0 0 0 0
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 48 49
M SBL 5 1 52
M SMT 5 CH2OH
M SVB 5 52 -4.7085 1.8791
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 46 47
M SBL 4 1 50
M SMT 4 OCH3
M SVB 4 50 4.6487 -1.1954
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 44 45
M SBL 3 1 48
M SMT 3 OCH3
M SVB 3 48 4.179 0.5375
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 OCH3
M SVB 2 46 3.4645 -1.4666
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 OCH3
M SVB 1 44 1.3441 1.5403
S SKP 8
ID FLIAFLGS0002
KNApSAcK_ID C00010140
NAME Isocaviunin 7-O-gentiobioside
CAS_RN 74517-73-0
FORMULA C31H38O18
EXACTMASS 698.205814412
AVERAGEMASS 698.62262
SMILES C(CO[C@@H]([C@@H](O)5)OC([C@H](O)[C@@H]5O)CO)(O1)[C@H](O)[C@H](O)[C@H](O)[C@@H]1Oc(c4)c(c(c3c4O)OC=C(C3=O)c(c2OC)cc(OC)c(OC)c2)OC
M END