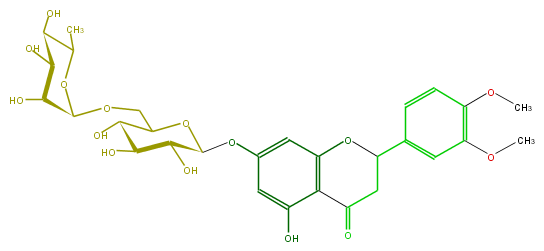
Mol:FL2FAFGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.2713 -0.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2713 -0.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4413 -0.3304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4413 -0.3304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2681 -1.5710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2681 -1.5710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4486 -1.9804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4486 -1.9804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1613 -1.5647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1613 -1.5647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1576 -0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1576 -0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8775 -1.9740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8775 -1.9740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5902 -1.5584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5902 -1.5584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5865 -0.7334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5865 -0.7334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8702 -0.3240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8702 -0.3240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2403 -0.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2403 -0.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9564 -0.7623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9564 -0.7623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6692 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6692 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6660 0.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6660 0.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9500 0.8877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9500 0.8877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2371 0.4724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2371 0.4724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8786 -2.6173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8786 -2.6173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8968 -0.3849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8968 -0.3849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4486 -2.6885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4486 -2.6885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3128 -0.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3128 -0.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2971 0.8423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2971 0.8423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9949 -0.3248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9949 -0.3248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9346 0.4742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9346 0.4742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6139 0.0723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6139 0.0723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2012 -0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2012 -0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4029 -0.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4029 -0.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6094 -0.6601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6094 -0.6601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0220 0.0547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0220 0.0547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8204 -0.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8204 -0.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1203 0.3269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1203 0.3269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9110 0.4197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9110 0.4197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1370 -0.2473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1370 -0.2473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7915 -0.6336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7915 -0.6336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9749 -1.0746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9749 -1.0746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7264 1.7795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7264 1.7795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4411 2.1921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4411 2.1921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2143 1.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2143 1.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4235 0.6003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4235 0.6003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7088 0.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7088 0.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9355 0.9812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9355 0.9812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9949 0.5843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9949 0.5843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7942 1.8322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7942 1.8322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2881 2.6885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2881 2.6885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8079 2.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8079 2.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 4 19 1 0 0 0 0 | + | 4 19 1 0 0 0 0 |
| − | 13 20 1 0 0 0 0 | + | 13 20 1 0 0 0 0 |
| − | 14 21 1 0 0 0 0 | + | 14 21 1 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 27 18 1 0 0 0 0 | + | 27 18 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 37 42 1 0 0 0 0 | + | 37 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 35 44 1 0 0 0 0 | + | 35 44 1 0 0 0 0 |
| − | 39 31 1 0 0 0 0 | + | 39 31 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FAFGS0002 | + | ID FL2FAFGS0002 |
| − | FORMULA C29H36O15 | + | FORMULA C29H36O15 |
| − | EXACTMASS 624.2054204819999 | + | EXACTMASS 624.2054204819999 |
| − | AVERAGEMASS 624.58714 | + | AVERAGEMASS 624.58714 |
| − | SMILES COc(c(OC)1)ccc(C(O5)CC(c(c54)c(cc(c4)OC(C(O)2)OC(COC(O3)C(C(C(O)C(C)3)O)O)C(O)C(O)2)O)=O)c1 | + | SMILES COc(c(OC)1)ccc(C(O5)CC(c(c54)c(cc(c4)OC(C(O)2)OC(COC(O3)C(C(C(O)C(C)3)O)O)C(O)C(O)2)O)=O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-0.2713 -0.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4413 -0.3304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2681 -1.5710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4486 -1.9804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1613 -1.5647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1576 -0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8775 -1.9740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5902 -1.5584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5865 -0.7334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8702 -0.3240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2403 -0.3525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9564 -0.7623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6692 -0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6660 0.4780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9500 0.8877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2371 0.4724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8786 -2.6173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8968 -0.3849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4486 -2.6885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3128 -0.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2971 0.8423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9949 -0.3248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9346 0.4742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6139 0.0723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2012 -0.6424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4029 -0.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6094 -0.6601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0220 0.0547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8204 -0.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1203 0.3269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9110 0.4197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1370 -0.2473 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7915 -0.6336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9749 -1.0746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7264 1.7795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4411 2.1921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2143 1.3986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4235 0.6003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7088 0.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9355 0.9812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9949 0.5843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7942 1.8322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2881 2.6885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8079 2.2944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
1 18 1 0 0 0 0
4 19 1 0 0 0 0
13 20 1 0 0 0 0
14 21 1 0 0 0 0
20 22 1 0 0 0 0
21 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
27 18 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
38 41 1 0 0 0 0
37 42 1 0 0 0 0
36 43 1 0 0 0 0
35 44 1 0 0 0 0
39 31 1 0 0 0 0
S SKP 5
ID FL2FAFGS0002
FORMULA C29H36O15
EXACTMASS 624.2054204819999
AVERAGEMASS 624.58714
SMILES COc(c(OC)1)ccc(C(O5)CC(c(c54)c(cc(c4)OC(C(O)2)OC(COC(O3)C(C(C(O)C(C)3)O)O)C(O)C(O)2)O)=O)c1
M END