Mol:FL3FACGS0076
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.5010 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5010 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5010 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5010 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2154 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2154 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9298 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9298 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9298 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9298 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2154 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2154 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6443 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6443 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3588 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3588 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3588 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3588 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6443 0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6443 0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0732 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0732 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7877 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7877 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5021 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5021 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5021 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5021 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7877 1.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7877 1.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0732 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0732 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6443 -1.8397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6443 -1.8397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2186 0.5697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2186 0.5697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2154 -1.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2154 -1.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1250 1.8197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1250 1.8197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2079 0.2277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2079 0.2277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9829 -0.1273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9829 -0.1273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2884 -0.5731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2884 -0.5731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6749 -0.0211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6749 -0.0211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8658 0.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8658 0.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5602 0.5872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5602 0.5872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1298 0.0325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1298 0.0325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6115 0.5162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6115 0.5162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4910 0.5382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4910 0.5382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9590 0.2417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9590 0.2417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0798 -0.7653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0798 -0.7653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9230 -0.3218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9230 -0.3218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0854 1.0954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0854 1.0954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8826 0.8819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8826 0.8819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1611 0.4812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1611 0.4812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7447 -0.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7447 -0.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9474 -0.0178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9474 -0.0178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6689 0.3830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6689 0.3830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2361 -0.6615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2361 -0.6615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1098 0.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1098 0.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2079 1.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2079 1.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6342 1.4123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6342 1.4123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 37 29 1 0 0 0 0 | + | 37 29 1 0 0 0 0 |
| − | 18 25 1 0 0 0 0 | + | 18 25 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
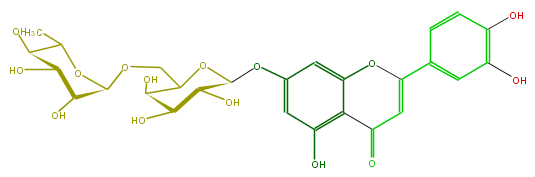
| − | ID FL3FACGS0076 | + | ID FL3FACGS0076 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C(Oc(c5)cc(O)c(c54)C(=O)C=C(O4)c(c3)cc(c(c3)O)O)2)O)C(C(O)C(O)1)O | + | SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C(Oc(c5)cc(O)c(c54)C(=O)C=C(O4)c(c3)cc(c(c3)O)O)2)O)C(C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
0.5010 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5010 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2154 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9298 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9298 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2154 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6443 -1.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3588 -0.6023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3588 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6443 0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0732 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7877 0.2227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5021 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5021 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7877 1.8726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0732 1.4602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6443 -1.8397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2186 0.5697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2154 -1.8726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1250 1.8197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.2079 0.2277 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9829 -0.1273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2884 -0.5731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6749 -0.0211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8658 0.1412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5602 0.5872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1298 0.0325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6115 0.5162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4910 0.5382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9590 0.2417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0798 -0.7653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9230 -0.3218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0854 1.0954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8826 0.8819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1611 0.4812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7447 -0.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9474 -0.0178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6689 0.3830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2361 -0.6615 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.1098 0.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.2079 1.4341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6342 1.4123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
14 20 1 0 0 0 0
13 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
34 41 1 0 0 0 0
33 42 1 0 0 0 0
37 29 1 0 0 0 0
18 25 1 0 0 0 0
S SKP 5
ID FL3FACGS0076
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C(Oc(c5)cc(O)c(c54)C(=O)C=C(O4)c(c3)cc(c(c3)O)O)2)O)C(C(O)C(O)1)O
M END