Mol:FL3FADGS0012
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.1847 -0.6880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1847 -0.6880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1846 -1.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1846 -1.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5164 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5164 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2172 -1.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2172 -1.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2172 -0.6878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2172 -0.6878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5164 -0.2832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5164 -0.2832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9183 -1.9019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9183 -1.9019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6193 -1.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6193 -1.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6193 -0.6880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6193 -0.6880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9183 -0.2830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9183 -0.2830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9183 -2.6265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9183 -2.6265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3200 -0.2833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3200 -0.2833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0345 -0.6958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0345 -0.6958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7487 -0.2834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7487 -0.2834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7488 0.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7488 0.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0345 0.9540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0345 0.9540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3199 0.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3199 0.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5165 -2.7096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5165 -2.7096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5288 0.9920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5288 0.9920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8841 -0.2840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8841 -0.2840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9680 -0.3301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9680 -0.3301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3671 -1.1233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3671 -1.1233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5016 -0.7868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5016 -0.7868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6666 -0.7778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6666 -0.7778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2735 -0.1708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2735 -0.1708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1574 -0.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1574 -0.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6399 -0.7181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6399 -0.7181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3038 -1.1690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3038 -1.1690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7054 -1.3678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7054 -1.3678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2102 -2.8345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2102 -2.8345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5237 -2.2988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5237 -2.2988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7380 -2.9096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7380 -2.9096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9892 -2.3606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9892 -2.3606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1849 -2.1358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1849 -2.1358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9281 -1.7065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9281 -1.7065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6999 -2.2420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6999 -2.2420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5638 -3.0681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5638 -3.0681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9485 -2.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9485 -2.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5437 -1.8216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5437 -1.8216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4470 -1.4926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4470 -1.4926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5288 -2.4444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5288 -2.4444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6751 0.0544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6751 0.0544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6365 0.0546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6365 0.0546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1926 0.8901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1926 0.8901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0388 1.6362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0388 1.6362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6423 3.0681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6423 3.0681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 26 42 1 0 0 0 0 | + | 26 42 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 16 45 1 0 0 0 0 | + | 16 45 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 39 40 41 | + | M SAL 1 3 39 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^ COOH | + | M SMT 1 ^ COOH |
| − | M SBV 1 45 0.8438 -0.4204 | + | M SBV 1 45 0.8438 -0.4204 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 3 42 43 44 | + | M SAL 2 3 42 43 44 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 ^ COOH | + | M SMT 2 ^ COOH |
| − | M SBV 2 48 0.5177 -0.5428 | + | M SBV 2 48 0.5177 -0.5428 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 45 46 | + | M SAL 3 2 45 46 |
| − | M SBL 3 1 50 | + | M SBL 3 1 50 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 50 -0.0043 -0.6822 | + | M SBV 3 50 -0.0043 -0.6822 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FADGS0012 | + | ID FL3FADGS0012 |
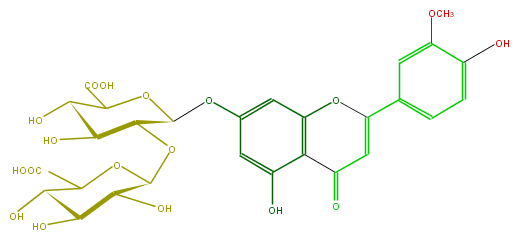
| − | FORMULA C28H28O18 | + | FORMULA C28H28O18 |
| − | EXACTMASS 652.1275640920001 | + | EXACTMASS 652.1275640920001 |
| − | AVERAGEMASS 652.51112 | + | AVERAGEMASS 652.51112 |
| − | SMILES Oc(c43)cc(cc(OC(c(c5)cc(c(c5)O)OC)=CC4=O)3)OC(C1OC(O2)C(O)C(O)C(C(C(O)=O)2)O)OC(C(O)=O)C(C1O)O | + | SMILES Oc(c43)cc(cc(OC(c(c5)cc(c(c5)O)OC)=CC4=O)3)OC(C1OC(O2)C(O)C(O)C(C(C(O)=O)2)O)OC(C(O)=O)C(C1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-0.1847 -0.6880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1846 -1.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5164 -1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2172 -1.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2172 -0.6878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5164 -0.2832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9183 -1.9019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6193 -1.4972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6193 -0.6880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9183 -0.2830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9183 -2.6265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3200 -0.2833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0345 -0.6958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7487 -0.2834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7488 0.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0345 0.9540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3199 0.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5165 -2.7096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5288 0.9920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8841 -0.2840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9680 -0.3301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3671 -1.1233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5016 -0.7868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6666 -0.7778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2735 -0.1708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1574 -0.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6399 -0.7181 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3038 -1.1690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7054 -1.3678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2102 -2.8345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5237 -2.2988 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7380 -2.9096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9892 -2.3606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1849 -2.1358 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9281 -1.7065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6999 -2.2420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5638 -3.0681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9485 -2.6700 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5437 -1.8216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4470 -1.4926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5288 -2.4444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6751 0.0544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6365 0.0546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1926 0.8901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0388 1.6362 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6423 3.0681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
30 31 1 0 0 0 0
34 29 1 0 0 0 0
24 20 1 0 0 0 0
39 40 2 0 0 0 0
39 41 1 0 0 0 0
36 39 1 0 0 0 0
42 43 2 0 0 0 0
42 44 1 0 0 0 0
26 42 1 0 0 0 0
45 46 1 0 0 0 0
16 45 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 39 40 41
M SBL 1 1 45
M SMT 1 ^ COOH
M SBV 1 45 0.8438 -0.4204
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 3 42 43 44
M SBL 2 1 48
M SMT 2 ^ COOH
M SBV 2 48 0.5177 -0.5428
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 45 46
M SBL 3 1 50
M SMT 3 OCH3
M SBV 3 50 -0.0043 -0.6822
S SKP 5
ID FL3FADGS0012
FORMULA C28H28O18
EXACTMASS 652.1275640920001
AVERAGEMASS 652.51112
SMILES Oc(c43)cc(cc(OC(c(c5)cc(c(c5)O)OC)=CC4=O)3)OC(C1OC(O2)C(O)C(O)C(C(C(O)=O)2)O)OC(C(O)=O)C(C1O)O
M END