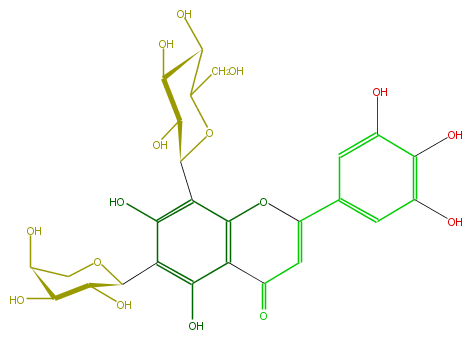
Mol:FL3FAGCS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5945 -1.0535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5945 -1.0535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5945 -1.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5945 -1.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8801 -2.2908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8801 -2.2908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1656 -1.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1656 -1.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1656 -1.0535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1656 -1.0535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8801 -0.6410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8801 -0.6410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5488 -2.2908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5488 -2.2908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2632 -1.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2632 -1.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2632 -1.0535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2632 -1.0535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5488 -0.6410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5488 -0.6410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5488 -2.9341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5488 -2.9341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3087 -0.6411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3087 -0.6411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6887 2.3940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6887 2.3940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4664 1.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4664 1.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1365 0.9564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1365 0.9564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1276 0.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1276 0.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5326 0.7327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5326 0.7327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9243 1.4750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9243 1.4750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1053 3.1156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1053 3.1156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4664 2.5059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4664 2.5059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5919 0.5009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5919 0.5009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8801 -3.1156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8801 -3.1156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0569 -0.6146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0569 -0.6146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8097 -1.0493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8097 -1.0493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5625 -0.6146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5625 -0.6146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5625 0.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5625 0.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8097 0.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8097 0.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0569 0.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0569 0.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1172 -1.9666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1172 -1.9666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6406 -2.5958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6406 -2.5958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9542 -2.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9542 -2.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2918 -2.3217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2918 -2.3217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7731 -1.8402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7731 -1.8402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3737 -2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3737 -2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1172 -1.2250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1172 -1.2250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3150 -2.5958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3150 -2.5958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3069 -2.7026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3069 -2.7026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3150 0.6890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3150 0.6890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3150 -1.0491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3150 -1.0491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8097 1.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8097 1.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4332 1.9661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4332 1.9661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4843 1.4364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4843 1.4364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 3 22 1 0 0 0 0 | + | 3 22 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 9 1 0 0 0 0 | + | 23 9 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 2 1 0 0 0 0 | + | 32 2 1 0 0 0 0 |
| − | 26 38 1 0 0 0 0 | + | 26 38 1 0 0 0 0 |
| − | 25 39 1 0 0 0 0 | + | 25 39 1 0 0 0 0 |
| − | 27 40 1 0 0 0 0 | + | 27 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 18 41 1 0 0 0 0 | + | 18 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 46 -0.4912 -0.4912 | + | M SBV 1 46 -0.4912 -0.4912 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAGCS0004 | + | ID FL3FAGCS0004 |
| − | FORMULA C26H28O16 | + | FORMULA C26H28O16 |
| − | EXACTMASS 596.137734848 | + | EXACTMASS 596.137734848 |
| − | AVERAGEMASS 596.49092 | + | AVERAGEMASS 596.49092 |
| − | SMILES c(C(O5)C(C(C(O)C5CO)O)O)(c31)c(c(C(C4O)OCC(C4O)O)c(c1C(C=C(O3)c(c2)cc(c(c(O)2)O)O)=O)O)O | + | SMILES c(C(O5)C(C(C(O)C5CO)O)O)(c31)c(c(C(C4O)OCC(C4O)O)c(c1C(C=C(O3)c(c2)cc(c(c(O)2)O)O)=O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.5945 -1.0535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5945 -1.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8801 -2.2908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1656 -1.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1656 -1.0535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8801 -0.6410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5488 -2.2908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2632 -1.8785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2632 -1.0535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5488 -0.6410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5488 -2.9341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3087 -0.6411 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6887 2.3940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4664 1.8049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1365 0.9564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1276 0.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5326 0.7327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9243 1.4750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1053 3.1156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4664 2.5059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5919 0.5009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8801 -3.1156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0569 -0.6146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8097 -1.0493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5625 -0.6146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5625 0.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8097 0.6893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0569 0.2547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1172 -1.9666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6406 -2.5958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9542 -2.3289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2918 -2.3217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7731 -1.8402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3737 -2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1172 -1.2250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3150 -2.5958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3069 -2.7026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3150 0.6890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3150 -1.0491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8097 1.5581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4332 1.9661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4843 1.4364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
3 22 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 23 1 0 0 0 0
23 9 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 2 1 0 0 0 0
26 38 1 0 0 0 0
25 39 1 0 0 0 0
27 40 1 0 0 0 0
41 42 1 0 0 0 0
18 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 CH2OH
M SBV 1 46 -0.4912 -0.4912
S SKP 5
ID FL3FAGCS0004
FORMULA C26H28O16
EXACTMASS 596.137734848
AVERAGEMASS 596.49092
SMILES c(C(O5)C(C(C(O)C5CO)O)O)(c31)c(c(C(C4O)OCC(C4O)O)c(c1C(C=C(O3)c(c2)cc(c(c(O)2)O)O)=O)O)O
M END