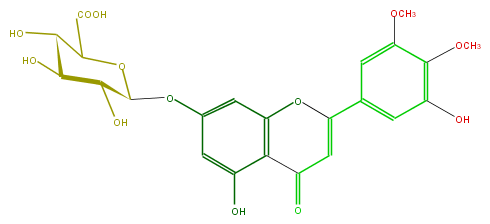
Mol:FL3FAKGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4798 -0.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4798 -0.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4798 -1.6917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4798 -1.6917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7789 -2.0964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7789 -2.0964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0778 -1.6917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0778 -1.6917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0778 -0.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0778 -0.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7789 -0.4775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7789 -0.4775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6232 -2.0964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6232 -2.0964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3241 -1.6917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3241 -1.6917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3241 -0.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3241 -0.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6232 -0.4775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6232 -0.4775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6232 -2.8854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6232 -2.8854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0248 -0.4777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0248 -0.4777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7394 -0.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7394 -0.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4538 -0.4777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4538 -0.4777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4538 0.3472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4538 0.3472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7394 0.7597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7394 0.7597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0248 0.3472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0248 0.3472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7789 -2.9042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7789 -2.9042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1680 -0.8902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1680 -0.8902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2145 -0.4103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2145 -0.4103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7226 0.9623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7226 0.9623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6071 0.0407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6071 0.0407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7505 -0.0911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7505 -0.0911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0712 -0.4735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0712 -0.4735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2785 0.3003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2785 0.3003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1412 0.4562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1412 0.4562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5206 1.0258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5206 1.0258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2319 0.4313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2319 0.4313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3861 -0.9349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3861 -0.9349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7394 1.4722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7394 1.4722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3430 2.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3430 2.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1746 0.7501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1746 0.7501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5206 1.5272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5206 1.5272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2971 1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2971 1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3847 2.5175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3847 2.5175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2246 1.6150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2246 1.6150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 14 19 1 0 0 0 0 | + | 14 19 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 16 30 1 0 0 0 0 | + | 16 30 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 15 32 1 0 0 0 0 | + | 15 32 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 34 | + | M SBL 1 1 34 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 34 0.0000 -0.7125 | + | M SBV 1 34 0.0000 -0.7125 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 32 33 | + | M SAL 2 2 32 33 |
| − | M SBL 2 1 36 | + | M SBL 2 1 36 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 36 -0.7208 -0.4029 | + | M SBV 2 36 -0.7208 -0.4029 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 3 34 35 36 | + | M SAL 3 3 34 35 36 |
| − | M SBL 3 1 39 | + | M SBL 3 1 39 |
| − | M SMT 3 ^ COOH | + | M SMT 3 ^ COOH |
| − | M SBV 3 39 0.1559 -0.9771 | + | M SBV 3 39 0.1559 -0.9771 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAKGS0001 | + | ID FL3FAKGS0001 |
| − | FORMULA C23H22O13 | + | FORMULA C23H22O13 |
| − | EXACTMASS 506.10604078999995 | + | EXACTMASS 506.10604078999995 |
| − | AVERAGEMASS 506.41298000000006 | + | AVERAGEMASS 506.41298000000006 |
| − | SMILES OC(C4C(O)=O)C(C(O)C(O4)Oc(c3)cc(c(c3O)1)OC(c(c2)cc(OC)c(OC)c2O)=CC1=O)O | + | SMILES OC(C4C(O)=O)C(C(O)C(O4)Oc(c3)cc(c(c3O)1)OC(c(c2)cc(OC)c(OC)c2O)=CC1=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-1.4798 -0.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4798 -1.6917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7789 -2.0964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0778 -1.6917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0778 -0.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7789 -0.4775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6232 -2.0964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3241 -1.6917 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3241 -0.8824 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6232 -0.4775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6232 -2.8854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0248 -0.4777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7394 -0.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4538 -0.4777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4538 0.3472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7394 0.7597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0248 0.3472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7789 -2.9042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1680 -0.8902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2145 -0.4103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7226 0.9623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6071 0.0407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7505 -0.0911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0712 -0.4735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2785 0.3003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1412 0.4562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5206 1.0258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2319 0.4313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3861 -0.9349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7394 1.4722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3430 2.9042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1746 0.7501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5206 1.5272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2971 1.4333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3847 2.5175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2246 1.6150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
14 19 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 20 1 0 0 0 0
20 1 1 0 0 0 0
30 31 1 0 0 0 0
16 30 1 0 0 0 0
32 33 1 0 0 0 0
15 32 1 0 0 0 0
34 35 2 0 0 0 0
34 36 1 0 0 0 0
26 34 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 34
M SMT 1 OCH3
M SBV 1 34 0.0000 -0.7125
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 32 33
M SBL 2 1 36
M SMT 2 OCH3
M SBV 2 36 -0.7208 -0.4029
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 3 34 35 36
M SBL 3 1 39
M SMT 3 ^ COOH
M SBV 3 39 0.1559 -0.9771
S SKP 5
ID FL3FAKGS0001
FORMULA C23H22O13
EXACTMASS 506.10604078999995
AVERAGEMASS 506.41298000000006
SMILES OC(C4C(O)=O)C(C(O)C(O4)Oc(c3)cc(c(c3O)1)OC(c(c2)cc(OC)c(OC)c2O)=CC1=O)O
M END