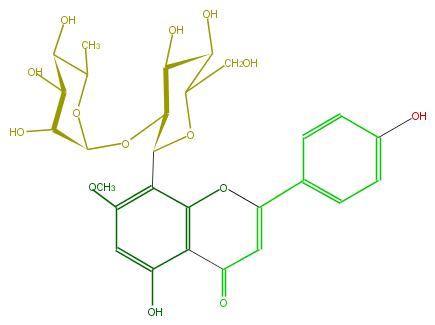
Mol:FL3FCACS0021
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0371 -0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0371 -0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0371 -1.7422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0371 -1.7422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3227 -2.1547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3227 -2.1547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6082 -1.7422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6082 -1.7422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6082 -0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6082 -0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3227 -0.5048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3227 -0.5048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1062 -2.1547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1062 -2.1547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8206 -1.7422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8206 -1.7422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8206 -0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8206 -0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1062 -0.5048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1062 -0.5048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1062 -2.7978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1062 -2.7978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1243 2.1747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1243 2.1747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0565 1.8870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0565 1.8870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0367 0.9768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0367 0.9768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3084 0.2046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3084 0.2046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5458 0.5601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5458 0.5601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6601 1.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6601 1.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2395 2.9793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2395 2.9793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9069 2.6609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9069 2.6609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8428 0.3815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8428 0.3815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3227 -2.9793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3227 -2.9793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6937 -0.3724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6937 -0.3724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4465 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4465 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1993 -0.3724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1993 -0.3724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1993 0.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1993 0.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4465 0.9315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4465 0.9315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6937 0.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6937 0.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9517 0.9312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9517 0.9312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3008 0.6436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3008 0.6436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6263 0.2542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6263 0.2542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8404 1.0031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8404 1.0031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6263 1.7564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6263 1.7564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3008 2.1459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3008 2.1459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0868 1.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0868 1.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0675 2.8571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0675 2.8571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6577 2.3693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6577 2.3693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7334 1.8264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7334 1.8264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9517 0.6133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9517 0.6133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6183 -0.4746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6183 -0.4746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2606 0.6375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2606 0.6375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2040 1.9926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2040 1.9926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8851 1.1810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8851 1.1810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 12 13 1 1 0 0 0 | + | 12 13 1 1 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 15 14 1 1 0 0 0 | + | 15 14 1 1 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 12 18 1 0 0 0 0 | + | 12 18 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 6 15 1 0 0 0 0 | + | 6 15 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 9 22 1 0 0 0 0 | + | 9 22 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 29 1 1 0 0 0 | + | 34 29 1 1 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 29 38 1 0 0 0 0 | + | 29 38 1 0 0 0 0 |
| − | 30 20 1 0 0 0 0 | + | 30 20 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 1 39 1 0 0 0 0 | + | 1 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 17 41 1 0 0 0 0 | + | 17 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 44 0.5812 -0.4426 | + | M SBV 1 44 0.5812 -0.4426 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 46 -0.8641 -0.6010 | + | M SBV 2 46 -0.8641 -0.6010 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FCACS0021 | + | ID FL3FCACS0021 |
| − | FORMULA C28H32O14 | + | FORMULA C28H32O14 |
| − | EXACTMASS 592.179205732 | + | EXACTMASS 592.179205732 |
| − | AVERAGEMASS 592.54528 | + | AVERAGEMASS 592.54528 |
| − | SMILES c(O1)(c(C(C(OC(O5)C(O)C(O)C(O)C(C)5)4)OC(C(C4O)O)CO)3)c(c(cc(OC)3)O)C(C=C1c(c2)ccc(c2)O)=O | + | SMILES c(O1)(c(C(C(OC(O5)C(O)C(O)C(O)C(C)5)4)OC(C(C4O)O)CO)3)c(c(cc(OC)3)O)C(C=C1c(c2)ccc(c2)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-2.0371 -0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0371 -1.7422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3227 -2.1547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6082 -1.7422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6082 -0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3227 -0.5048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1062 -2.1547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8206 -1.7422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8206 -0.9172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1062 -0.5048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1062 -2.7978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1243 2.1747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0565 1.8870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0367 0.9768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3084 0.2046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5458 0.5601 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6601 1.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2395 2.9793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9069 2.6609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8428 0.3815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3227 -2.9793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6937 -0.3724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4465 -0.8071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1993 -0.3724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1993 0.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4465 0.9315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6937 0.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9517 0.9312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3008 0.6436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6263 0.2542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8404 1.0031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6263 1.7564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3008 2.1459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0868 1.3971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0675 2.8571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6577 2.3693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7334 1.8264 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9517 0.6133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6183 -0.4746 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2606 0.6375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2040 1.9926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8851 1.1810 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
12 13 1 1 0 0 0
13 14 1 1 0 0 0
15 14 1 1 0 0 0
15 16 1 0 0 0 0
16 17 1 0 0 0 0
17 12 1 0 0 0 0
12 18 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
6 15 1 0 0 0 0
3 21 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 22 1 0 0 0 0
9 22 1 0 0 0 0
25 28 1 0 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 29 1 1 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
34 37 1 0 0 0 0
29 38 1 0 0 0 0
30 20 1 0 0 0 0
39 40 1 0 0 0 0
1 39 1 0 0 0 0
41 42 1 0 0 0 0
17 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 ^ OCH3
M SBV 1 44 0.5812 -0.4426
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 CH2OH
M SBV 2 46 -0.8641 -0.6010
S SKP 5
ID FL3FCACS0021
FORMULA C28H32O14
EXACTMASS 592.179205732
AVERAGEMASS 592.54528
SMILES c(O1)(c(C(C(OC(O5)C(O)C(O)C(O)C(C)5)4)OC(C(C4O)O)CO)3)c(c(cc(OC)3)O)C(C=C1c(c2)ccc(c2)O)=O
M END