Mol:FL3FCAGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 32 35 0 0 0 0 0 0 0 0999 V2000 | + | 32 35 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.3443 0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3443 0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0456 0.3586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0456 0.3586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2657 -0.1134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2657 -0.1134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7846 -0.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7846 -0.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0833 0.2678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0833 0.2678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8632 0.7399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8632 0.7399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0047 -0.6309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0047 -0.6309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5235 -0.6763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5235 -0.6763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8223 -0.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8223 -0.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6022 0.2224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6022 0.2224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9343 -1.0307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9343 -1.0307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3410 -0.2950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3410 -0.2950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5654 -0.7761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5654 -0.7761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0942 -0.8224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0942 -0.8224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3986 -0.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3986 -0.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1743 0.0935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1743 0.0935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6454 0.1398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6454 0.1398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9273 -0.4338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9273 -0.4338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0326 -0.4463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0326 -0.4463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6393 -0.6919 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.6393 -0.6919 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.1236 -1.3725 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1236 -1.3725 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.3811 -1.0837 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.3811 -1.0837 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.6647 -1.0760 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6647 -1.0760 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.1853 -0.5553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1853 -0.5553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9438 -0.8276 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.9438 -0.8276 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.2158 -1.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2158 -1.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9274 -1.4116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9274 -1.4116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9557 -1.7979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9557 -1.7979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4066 1.4970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4066 1.4970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4937 2.4932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4937 2.4932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5528 -0.2207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5528 -0.2207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9654 0.4938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9654 0.4938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 19 3 1 0 0 0 0 | + | 19 3 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 1 29 1 0 0 0 0 | + | 1 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 31 32 | + | M SAL 2 2 31 32 |
| − | M SBL 2 1 34 | + | M SBL 2 1 34 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 34 -2.2393 -0.0202 | + | M SVB 2 34 -2.2393 -0.0202 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 29 30 | + | M SAL 1 2 29 30 |
| − | M SBL 1 1 32 | + | M SBL 1 1 32 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 32 -0.7757 1.3601 | + | M SVB 1 32 -0.7757 1.3601 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FCAGS0001 | + | ID FL3FCAGS0001 |
| − | KNApSAcK_ID C00004198 | + | KNApSAcK_ID C00004198 |
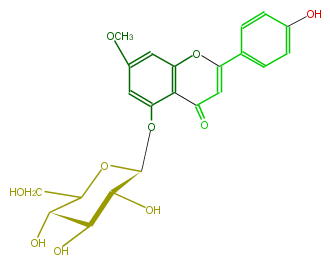
| − | NAME Apigenin 7-methyl ether 5-glucoside | + | NAME Apigenin 7-methyl ether 5-glucoside |
| − | CAS_RN 552-52-3 | + | CAS_RN 552-52-3 |
| − | FORMULA C22H22O10 | + | FORMULA C22H22O10 |
| − | EXACTMASS 446.121296924 | + | EXACTMASS 446.121296924 |
| − | AVERAGEMASS 446.40408 | + | AVERAGEMASS 446.40408 |
| − | SMILES O(c(c32)cc(OC)cc2OC(c(c4)ccc(c4)O)=CC3=O)[C@@H]([C@@H](O)1)OC(CO)[C@@H]([C@@H]1O)O | + | SMILES O(c(c32)cc(OC)cc2OC(c(c4)ccc(c4)O)=CC3=O)[C@@H]([C@@H](O)1)OC(CO)[C@@H]([C@@H]1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
32 35 0 0 0 0 0 0 0 0999 V2000
0.3443 0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0456 0.3586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2657 -0.1134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7846 -0.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0833 0.2678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8632 0.7399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0047 -0.6309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5235 -0.6763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8223 -0.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6022 0.2224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9343 -1.0307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3410 -0.2950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5654 -0.7761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0942 -0.8224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3986 -0.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1743 0.0935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6454 0.1398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9273 -0.4338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0326 -0.4463 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6393 -0.6919 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.1236 -1.3725 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.3811 -1.0837 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.6647 -1.0760 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.1853 -0.5553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9438 -0.8276 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.2158 -1.0247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9274 -1.4116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9557 -1.7979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4066 1.4970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4937 2.4932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5528 -0.2207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9654 0.4938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
19 3 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 19 1 0 0 0 0
1 29 1 0 0 0 0
29 30 1 0 0 0 0
25 31 1 0 0 0 0
31 32 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 31 32
M SBL 2 1 34
M SMT 2 CH2OH
M SVB 2 34 -2.2393 -0.0202
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 29 30
M SBL 1 1 32
M SMT 1 OCH3
M SVB 1 32 -0.7757 1.3601
S SKP 8
ID FL3FCAGS0001
KNApSAcK_ID C00004198
NAME Apigenin 7-methyl ether 5-glucoside
CAS_RN 552-52-3
FORMULA C22H22O10
EXACTMASS 446.121296924
AVERAGEMASS 446.40408
SMILES O(c(c32)cc(OC)cc2OC(c(c4)ccc(c4)O)=CC3=O)[C@@H]([C@@H](O)1)OC(CO)[C@@H]([C@@H]1O)O
M END