Mol:FL3FCAGS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 38 41 0 0 0 0 0 0 0 0999 V2000 | + | 38 41 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.3924 0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3924 0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3924 0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3924 0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8435 -0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8435 -0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2946 0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2946 0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2946 0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2946 0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8435 0.9718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8435 0.9718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7456 -0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7456 -0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1967 0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1967 0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1967 0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1967 0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7456 0.9718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7456 0.9718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9173 -0.4378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9173 -0.4378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6476 0.9717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6476 0.9717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1073 0.7063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1073 0.7063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5670 0.9717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5670 0.9717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5670 1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5670 1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1073 1.7680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1073 1.7680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6476 1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6476 1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0266 1.7679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0266 1.7679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8435 -0.4762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8435 -0.4762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5190 -0.8456 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -1.5190 -0.8456 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.0034 -1.5262 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.0034 -1.5262 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.2609 -1.2374 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.2609 -1.2374 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.4555 -1.2297 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.4555 -1.2297 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.0651 -0.7090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0651 -0.7090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8235 -0.9813 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.8235 -0.9813 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.0955 -1.1784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0955 -1.1784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8072 -1.5653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8072 -1.5653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2773 -0.4244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2773 -0.4244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2696 -1.7680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2696 -1.7680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8548 -0.4038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8548 -0.4038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3910 0.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3910 0.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0304 -0.3007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0304 -0.3007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5460 0.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5460 0.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0266 -0.2961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0266 -0.2961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5460 0.7721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5460 0.7721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3910 0.6495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3910 0.6495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0352 1.3302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0352 1.3302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4647 2.1963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4647 2.1963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 19 3 1 0 0 0 0 | + | 19 3 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 31 36 2 0 0 0 0 | + | 31 36 2 0 0 0 0 |
| − | 1 37 1 0 0 0 0 | + | 1 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 37 38 | + | M SAL 2 2 37 38 |
| − | M SBL 2 1 40 | + | M SBL 2 1 40 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 40 0.0352 1.3302 | + | M SVB 2 40 0.0352 1.3302 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 33 34 35 | + | M SAL 1 3 33 34 35 |
| − | M SBL 1 1 36 | + | M SBL 1 1 36 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SVB 1 36 -3.546 0.0706 | + | M SVB 1 36 -3.546 0.0706 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FCAGS0003 | + | ID FL3FCAGS0003 |
| − | KNApSAcK_ID C00004200 | + | KNApSAcK_ID C00004200 |
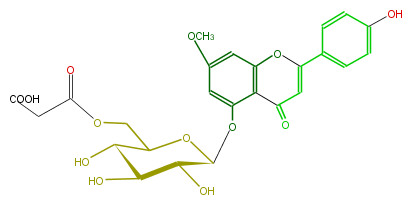
| − | NAME Apigenin 7-methyl ether 5-(6''-malonylglucoside) | + | NAME Apigenin 7-methyl ether 5-(6''-malonylglucoside) |
| − | CAS_RN 130733-29-8 | + | CAS_RN 130733-29-8 |
| − | FORMULA C25H24O13 | + | FORMULA C25H24O13 |
| − | EXACTMASS 532.121690854 | + | EXACTMASS 532.121690854 |
| − | AVERAGEMASS 532.45026 | + | AVERAGEMASS 532.45026 |
| − | SMILES O=C(CC(O)=O)OCC([C@H](O)4)O[C@H]([C@H]([C@@H](O)4)O)Oc(c12)cc(cc1OC(c(c3)ccc(c3)O)=CC2=O)OC | + | SMILES O=C(CC(O)=O)OCC([C@H](O)4)O[C@H]([C@H]([C@@H](O)4)O)Oc(c12)cc(cc1OC(c(c3)ccc(c3)O)=CC2=O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
38 41 0 0 0 0 0 0 0 0999 V2000
0.3924 0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3924 0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8435 -0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2946 0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2946 0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8435 0.9718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7456 -0.0699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1967 0.1906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1967 0.7114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7456 0.9718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9173 -0.4378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6476 0.9717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1073 0.7063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5670 0.9717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5670 1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1073 1.7680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6476 1.5026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0266 1.7679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8435 -0.4762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5190 -0.8456 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.0034 -1.5262 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.2609 -1.2374 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.4555 -1.2297 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.0651 -0.7090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8235 -0.9813 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.0955 -1.1784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8072 -1.5653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2773 -0.4244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2696 -1.7680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8548 -0.4038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3910 0.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0304 -0.3007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5460 0.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0266 -0.2961 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5460 0.7721 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3910 0.6495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0352 1.3302 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4647 2.1963 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
19 3 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
25 28 1 0 0 0 0
19 23 1 0 0 0 0
22 29 1 0 0 0 0
28 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
31 36 2 0 0 0 0
1 37 1 0 0 0 0
37 38 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 37 38
M SBL 2 1 40
M SMT 2 OCH3
M SVB 2 40 0.0352 1.3302
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 33 34 35
M SBL 1 1 36
M SMT 1 COOH
M SVB 1 36 -3.546 0.0706
S SKP 8
ID FL3FCAGS0003
KNApSAcK_ID C00004200
NAME Apigenin 7-methyl ether 5-(6''-malonylglucoside)
CAS_RN 130733-29-8
FORMULA C25H24O13
EXACTMASS 532.121690854
AVERAGEMASS 532.45026
SMILES O=C(CC(O)=O)OCC([C@H](O)4)O[C@H]([C@H]([C@@H](O)4)O)Oc(c12)cc(cc1OC(c(c3)ccc(c3)O)=CC2=O)OC
M END