Mol:FL4DAAGS0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.1564 -0.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1564 -0.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5579 -1.1717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5579 -1.1717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2724 -0.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2724 -0.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2724 0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2724 0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5579 0.4782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5579 0.4782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1564 0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1564 0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9868 -1.1717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9868 -1.1717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7012 -0.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7012 -0.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7012 0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7012 0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9868 0.4782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9868 0.4782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9868 -1.8903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9868 -1.8903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4962 0.4763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4962 0.4763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2215 0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2215 0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9466 0.4763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9466 0.4763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9466 1.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9466 1.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2215 1.7324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2215 1.7324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4962 1.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4962 1.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8658 0.3215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8658 0.3215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3434 -1.5503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3434 -1.5503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6717 1.7324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6717 1.7324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5579 -1.9958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5579 -1.9958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4153 -0.5905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4153 -0.5905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6370 -1.0932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6370 -1.0932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9872 -0.5233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9872 -0.5233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2596 -0.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2596 -0.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9836 0.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9836 0.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6558 -0.4710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6558 -0.4710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5768 -1.4154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5768 -1.4154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1086 -0.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1086 -0.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4826 -0.1669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4826 -0.1669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5220 1.2135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5220 1.2135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3528 1.4806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3528 1.4806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9415 0.7109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9415 0.7109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9796 -0.1610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9796 -0.1610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1488 -0.4282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1488 -0.4282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5602 0.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5602 0.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5680 -0.3425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5680 -0.3425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6717 0.9469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6717 0.9469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1965 1.9958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1965 1.9958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6857 1.9231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6857 1.9231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0845 0.2432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0845 0.2432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8931 1.0584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8931 1.0584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 6 0 0 0 | + | 9 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 23 28 1 0 0 0 0 | + | 23 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 18 25 1 0 0 0 0 | + | 18 25 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 30 35 1 0 0 0 0 | + | 30 35 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 31 40 1 0 0 0 0 | + | 31 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 27 41 1 0 0 0 0 | + | 27 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 46 0.4287 -0.7142 | + | M SBV 1 46 0.4287 -0.7142 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL4DAAGS0011 | + | ID FL4DAAGS0011 |
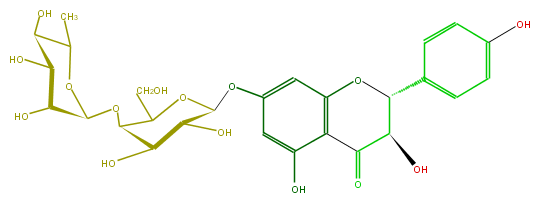
| − | FORMULA C27H32O15 | + | FORMULA C27H32O15 |
| − | EXACTMASS 596.174120354 | + | EXACTMASS 596.174120354 |
| − | AVERAGEMASS 596.5339799999999 | + | AVERAGEMASS 596.5339799999999 |
| − | SMILES Oc(c5)ccc(c5)C(C(O)4)Oc(c(C4=O)1)cc(OC(O3)C(C(O)C(C3CO)OC(O2)C(O)C(O)C(O)C(C)2)O)cc1O | + | SMILES Oc(c5)ccc(c5)C(C(O)4)Oc(c(C4=O)1)cc(OC(O3)C(C(O)C(C3CO)OC(O2)C(O)C(O)C(O)C(C)2)O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.1564 -0.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5579 -1.1717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2724 -0.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2724 0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5579 0.4782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1564 0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9868 -1.1717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7012 -0.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7012 0.0657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9868 0.4782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9868 -1.8903 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4962 0.4763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2215 0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9466 0.4763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9466 1.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2215 1.7324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4962 1.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8658 0.3215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3434 -1.5503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6717 1.7324 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5579 -1.9958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4153 -0.5905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6370 -1.0932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9872 -0.5233 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2596 -0.2496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9836 0.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6558 -0.4710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5768 -1.4154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1086 -0.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4826 -0.1669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5220 1.2135 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3528 1.4806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9415 0.7109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9796 -0.1610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1488 -0.4282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5602 0.3416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5680 -0.3425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6717 0.9469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1965 1.9958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6857 1.9231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0845 0.2432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8931 1.0584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
6 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
2 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
23 28 1 0 0 0 0
24 29 1 0 0 0 0
18 25 1 0 0 0 0
22 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
30 35 1 0 0 0 0
34 37 1 0 0 0 0
33 38 1 0 0 0 0
32 39 1 0 0 0 0
31 40 1 0 0 0 0
41 42 1 0 0 0 0
27 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ CH2OH
M SBV 1 46 0.4287 -0.7142
S SKP 5
ID FL4DAAGS0011
FORMULA C27H32O15
EXACTMASS 596.174120354
AVERAGEMASS 596.5339799999999
SMILES Oc(c5)ccc(c5)C(C(O)4)Oc(c(C4=O)1)cc(OC(O3)C(C(O)C(C3CO)OC(O2)C(O)C(O)C(O)C(C)2)O)cc1O
M END