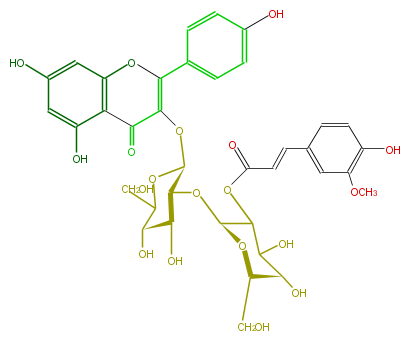
Mol:FL5FAAGL0081
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1317 1.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1317 1.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1317 1.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1317 1.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5754 0.8630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5754 0.8630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0190 1.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0190 1.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0190 1.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0190 1.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5754 2.1477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5754 2.1477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4627 0.8630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4627 0.8630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9064 1.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9064 1.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9064 1.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9064 1.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4627 2.1477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4627 2.1477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4627 0.3621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4627 0.3621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3503 2.1476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3503 2.1476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2166 1.8202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2166 1.8202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7836 2.1476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7836 2.1476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7836 2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7836 2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2166 3.1296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2166 3.1296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3503 2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3503 2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6878 2.1476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6878 2.1476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3504 3.1295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3504 3.1295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5123 0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5123 0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5754 0.2208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5754 0.2208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2541 -1.2024 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -1.2541 -1.2024 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.6720 -1.0522 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6720 -1.0522 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.6998 -0.4575 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6998 -0.4575 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.4091 0.0557 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.4091 0.0557 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.0476 -0.1696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0476 -0.1696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9913 -0.7893 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.9913 -0.7893 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -1.2541 -1.6752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2541 -1.6752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6720 -1.8086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6720 -1.8086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1665 -0.4502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1665 -0.4502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9335 -1.0712 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.9335 -1.0712 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.2947 -1.0712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.2947 -1.0712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.7538 -1.5154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7538 -1.5154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9108 -2.1383 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.9108 -2.1383 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.5497 -2.1384 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.5497 -2.1384 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.0905 -1.6941 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.0905 -1.6941 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.5490 -1.4803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5490 -1.4803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8087 -2.4471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8087 -2.4471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8463 0.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8463 0.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4432 -0.3821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4432 -0.3821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5603 0.5165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5603 0.5165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3713 0.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3713 0.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5984 0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5984 0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0521 0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0521 0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3249 -0.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3249 -0.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8705 -0.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8705 -0.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1433 0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1433 0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8705 0.8867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8705 0.8867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3249 0.8867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3249 0.8867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6878 0.4142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6878 0.4142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9837 -0.4321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9837 -0.4321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8056 -0.5041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8056 -0.5041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5989 -0.3764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5989 -0.3764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4992 -0.8117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4992 -0.8117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7215 -3.1296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7215 -3.1296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1381 -2.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1381 -2.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 32 30 1 0 0 0 0 | + | 32 30 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 31 1 0 0 0 0 | + | 40 31 1 0 0 0 0 |
| − | 39 41 2 0 0 0 0 | + | 39 41 2 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 47 50 1 0 0 0 0 | + | 47 50 1 0 0 0 0 |
| − | 46 51 1 0 0 0 0 | + | 46 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 27 53 1 0 0 0 0 | + | 27 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 34 55 1 0 0 0 0 | + | 34 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 55 56 | + | M SAL 3 2 55 56 |
| − | M SBL 3 1 60 | + | M SBL 3 1 60 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 60 0.7215 -3.1296 | + | M SVB 3 60 0.7215 -3.1296 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 53 54 | + | M SAL 2 2 53 54 |
| − | M SBL 2 1 58 | + | M SBL 2 1 58 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 58 -1.5989 -0.3764 | + | M SVB 2 58 -1.5989 -0.3764 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 56 | + | M SBL 1 1 56 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 56 2.815 -0.445 | + | M SVB 1 56 2.815 -0.445 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGL0081 | + | ID FL5FAAGL0081 |
| − | KNApSAcK_ID C00005882 | + | KNApSAcK_ID C00005882 |
| − | NAME Petunoside | + | NAME Petunoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C37H38O19 | + | FORMULA C37H38O19 |
| − | EXACTMASS 786.200729034 | + | EXACTMASS 786.200729034 |
| − | AVERAGEMASS 786.68622 | + | AVERAGEMASS 786.68622 |
| − | SMILES c(C=CC(=O)OC([C@H](O[C@H]([C@H](OC(=C(c(c6)ccc(O)c6)5)C(c(c(O5)4)c(O)cc(O)c4)=O)3)[C@H]([C@@H](O)C(CO)O3)O)2)C([C@@H](O)[C@@H](CO)O2)O)(c1)ccc(c1OC)O | + | SMILES c(C=CC(=O)OC([C@H](O[C@H]([C@H](OC(=C(c(c6)ccc(O)c6)5)C(c(c(O5)4)c(O)cc(O)c4)=O)3)[C@H]([C@@H](O)C(CO)O3)O)2)C([C@@H](O)[C@@H](CO)O2)O)(c1)ccc(c1OC)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
-3.1317 1.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1317 1.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5754 0.8630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0190 1.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0190 1.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5754 2.1477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4627 0.8630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9064 1.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9064 1.8265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4627 2.1477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4627 0.3621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3503 2.1476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2166 1.8202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7836 2.1476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7836 2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2166 3.1296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3503 2.8023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6878 2.1476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3504 3.1295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5123 0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5754 0.2208 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2541 -1.2024 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.6720 -1.0522 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.6998 -0.4575 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.4091 0.0557 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.0476 -0.1696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9913 -0.7893 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-1.2541 -1.6752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6720 -1.8086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1665 -0.4502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9335 -1.0712 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.2947 -1.0712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.7538 -1.5154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9108 -2.1383 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.5497 -2.1384 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.0905 -1.6941 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.5490 -1.4803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8087 -2.4471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8463 0.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4432 -0.3821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5603 0.5165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3713 0.0210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5984 0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0521 0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3249 -0.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8705 -0.0582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1433 0.4142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8705 0.8867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3249 0.8867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6878 0.4142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9837 -0.4321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8056 -0.5041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5989 -0.3764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4992 -0.8117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7215 -3.1296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1381 -2.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 20 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
36 37 1 0 0 0 0
35 38 1 0 0 0 0
32 30 1 0 0 0 0
39 40 1 0 0 0 0
40 31 1 0 0 0 0
39 41 2 0 0 0 0
39 42 1 0 0 0 0
42 43 2 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 44 1 0 0 0 0
47 50 1 0 0 0 0
46 51 1 0 0 0 0
51 52 1 0 0 0 0
27 53 1 0 0 0 0
53 54 1 0 0 0 0
34 55 1 0 0 0 0
55 56 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 55 56
M SBL 3 1 60
M SMT 3 CH2OH
M SVB 3 60 0.7215 -3.1296
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 53 54
M SBL 2 1 58
M SMT 2 CH2OH
M SVB 2 58 -1.5989 -0.3764
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 56
M SMT 1 OCH3
M SVB 1 56 2.815 -0.445
S SKP 8
ID FL5FAAGL0081
KNApSAcK_ID C00005882
NAME Petunoside
CAS_RN -
FORMULA C37H38O19
EXACTMASS 786.200729034
AVERAGEMASS 786.68622
SMILES c(C=CC(=O)OC([C@H](O[C@H]([C@H](OC(=C(c(c6)ccc(O)c6)5)C(c(c(O5)4)c(O)cc(O)c4)=O)3)[C@H]([C@@H](O)C(CO)O3)O)2)C([C@@H](O)[C@@H](CO)O2)O)(c1)ccc(c1OC)O
M END