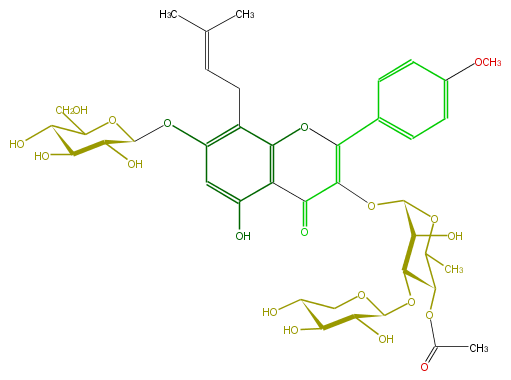
Mol:FL5FABGI0020
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 60 65 0 0 0 0 0 0 0 0999 V2000 | + | 60 65 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4047 0.9788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4047 0.9788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4047 0.1516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4047 0.1516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6883 -0.2620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6883 -0.2620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0280 0.1516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0280 0.1516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0280 0.9788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0280 0.9788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6883 1.3924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6883 1.3924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7443 -0.2620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7443 -0.2620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4606 0.1516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4606 0.1516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4606 0.9788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4606 0.9788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7443 1.3924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7443 1.3924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7443 -0.9069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7443 -0.9069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3626 1.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3626 1.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0926 1.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0926 1.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8227 1.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8227 1.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8227 2.3946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8227 2.3946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0926 2.8160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0926 2.8160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3626 2.3946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3626 2.3946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2603 1.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2603 1.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6883 -0.9866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6883 -0.9866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2125 -0.3492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2125 -0.3492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9080 -1.7704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9080 -1.7704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5974 -2.1684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5974 -2.1684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3786 -1.4030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3786 -1.4030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5974 -0.6330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5974 -0.6330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9080 -0.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9080 -0.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1268 -1.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1268 -1.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9721 -1.0003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9721 -1.0003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0852 -2.4313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0852 -2.4313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4738 -2.7286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4738 -2.7286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8751 -1.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8751 -1.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6883 2.2192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6883 2.2192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4044 2.6326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4044 2.6326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4044 3.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4044 3.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6899 3.8702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6899 3.8702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1189 3.8702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1189 3.8702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6552 -2.4042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6552 -2.4042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1331 -3.0350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1331 -3.0350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8213 -2.7674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8213 -2.7674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4855 -2.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4855 -2.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0029 -2.2775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0029 -2.2775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4008 -2.5953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4008 -2.5953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0567 -2.6637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0567 -2.6637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5338 -3.0521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5338 -3.0521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4440 -3.2452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4440 -3.2452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6086 -3.4405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6086 -3.4405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3606 -3.8702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3606 -3.8702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4335 -3.4405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4335 -3.4405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8110 1.4087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8110 1.4087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3330 0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3330 0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6448 1.0454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6448 1.0454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9806 1.0525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9806 1.0525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4632 1.5352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4632 1.5352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0653 1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0653 1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4726 1.0266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4726 1.0266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9323 0.7607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9323 0.7607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0427 0.5882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0427 0.5882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5526 2.8159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5526 2.8159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4726 2.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4726 2.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6376 1.7707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6376 1.7707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6114 1.4957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6114 1.4957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 19 3 1 0 0 0 0 | + | 19 3 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 21 1 1 0 0 0 | + | 26 21 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 6 31 1 0 0 0 0 | + | 6 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 38 44 1 0 0 0 0 | + | 38 44 1 0 0 0 0 |
| − | 39 28 1 0 0 0 0 | + | 39 28 1 0 0 0 0 |
| − | 29 45 1 0 0 0 0 | + | 29 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 48 49 1 1 0 0 0 | + | 48 49 1 1 0 0 0 |
| − | 49 50 1 1 0 0 0 | + | 49 50 1 1 0 0 0 |
| − | 51 50 1 1 0 0 0 | + | 51 50 1 1 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 48 1 0 0 0 0 | + | 53 48 1 0 0 0 0 |
| − | 48 54 1 0 0 0 0 | + | 48 54 1 0 0 0 0 |
| − | 49 55 1 0 0 0 0 | + | 49 55 1 0 0 0 0 |
| − | 50 56 1 0 0 0 0 | + | 50 56 1 0 0 0 0 |
| − | 51 18 1 0 0 0 0 | + | 51 18 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 15 57 1 0 0 0 0 | + | 15 57 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 53 59 1 0 0 0 0 | + | 53 59 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 57 58 | + | M SAL 1 2 57 58 |
| − | M SBL 1 1 63 | + | M SBL 1 1 63 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 63 -0.7298 -0.4213 | + | M SBV 1 63 -0.7298 -0.4213 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 59 60 | + | M SAL 2 2 59 60 |
| − | M SBL 2 1 65 | + | M SBL 2 1 65 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 65 0.5723 -0.5533 | + | M SBV 2 65 0.5723 -0.5533 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FABGI0020 | + | ID FL5FABGI0020 |
| − | FORMULA C40H50O20 | + | FORMULA C40H50O20 |
| − | EXACTMASS 850.28954404 | + | EXACTMASS 850.28954404 |
| − | AVERAGEMASS 850.8130000000001 | + | AVERAGEMASS 850.8130000000001 |
| − | SMILES C(C5OC(C)=O)(OC(C(C5OC(C6O)OCC(C6O)O)O)OC(C(=O)4)=C(Oc(c42)c(c(OC(C3O)OC(C(O)C3O)CO)cc2O)CC=C(C)C)c(c1)ccc(OC)c1)C | + | SMILES C(C5OC(C)=O)(OC(C(C5OC(C6O)OCC(C6O)O)O)OC(C(=O)4)=C(Oc(c42)c(c(OC(C3O)OC(C(O)C3O)CO)cc2O)CC=C(C)C)c(c1)ccc(OC)c1)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
60 65 0 0 0 0 0 0 0 0999 V2000
-1.4047 0.9788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4047 0.1516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6883 -0.2620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0280 0.1516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0280 0.9788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6883 1.3924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7443 -0.2620 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4606 0.1516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4606 0.9788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7443 1.3924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7443 -0.9069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3626 1.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0926 1.1301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8227 1.5515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8227 2.3946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0926 2.8160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3626 2.3946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2603 1.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6883 -0.9866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2125 -0.3492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9080 -1.7704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5974 -2.1684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3786 -1.4030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5974 -0.6330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9080 -0.2348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1268 -1.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9721 -1.0003 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0852 -2.4313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4738 -2.7286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8751 -1.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6883 2.2192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4044 2.6326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4044 3.4576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6899 3.8702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1189 3.8702 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6552 -2.4042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1331 -3.0350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8213 -2.7674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4855 -2.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0029 -2.2775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4008 -2.5953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0567 -2.6637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5338 -3.0521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4440 -3.2452 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6086 -3.4405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3606 -3.8702 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4335 -3.4405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8110 1.4087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3330 0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6448 1.0454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9806 1.0525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4632 1.5352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0653 1.2175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4726 1.0266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9323 0.7607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0427 0.5882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5526 2.8159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4726 2.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6376 1.7707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6114 1.4957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
4 3 1 0 0 0 0
1 18 1 0 0 0 0
19 3 1 0 0 0 0
8 20 1 0 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 21 1 1 0 0 0
26 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
25 20 1 0 0 0 0
6 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
33 35 1 0 0 0 0
36 37 1 1 0 0 0
37 38 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 36 1 0 0 0 0
36 42 1 0 0 0 0
37 43 1 0 0 0 0
38 44 1 0 0 0 0
39 28 1 0 0 0 0
29 45 1 0 0 0 0
45 46 2 0 0 0 0
45 47 1 0 0 0 0
48 49 1 1 0 0 0
49 50 1 1 0 0 0
51 50 1 1 0 0 0
51 52 1 0 0 0 0
52 53 1 0 0 0 0
53 48 1 0 0 0 0
48 54 1 0 0 0 0
49 55 1 0 0 0 0
50 56 1 0 0 0 0
51 18 1 0 0 0 0
57 58 1 0 0 0 0
15 57 1 0 0 0 0
59 60 1 0 0 0 0
53 59 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 57 58
M SBL 1 1 63
M SMT 1 OCH3
M SBV 1 63 -0.7298 -0.4213
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 59 60
M SBL 2 1 65
M SMT 2 ^CH2OH
M SBV 2 65 0.5723 -0.5533
S SKP 5
ID FL5FABGI0020
FORMULA C40H50O20
EXACTMASS 850.28954404
AVERAGEMASS 850.8130000000001
SMILES C(C5OC(C)=O)(OC(C(C5OC(C6O)OCC(C6O)O)O)OC(C(=O)4)=C(Oc(c42)c(c(OC(C3O)OC(C(O)C3O)CO)cc2O)CC=C(C)C)c(c1)ccc(OC)c1)C
M END