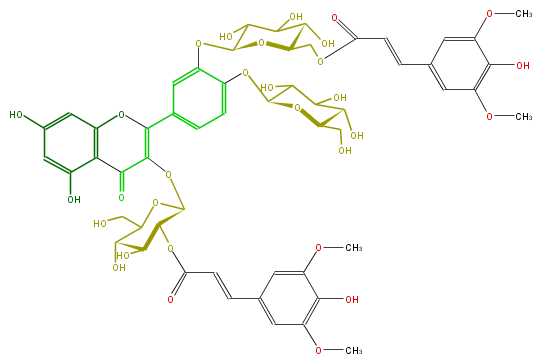
Mol:FL5FACGS0120
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 85 92 0 0 0 0 0 0 0 0999 V2000 | + | 85 92 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.9573 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9573 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6718 1.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6718 1.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6718 2.7160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6718 2.7160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9573 3.1285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9573 3.1285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2429 2.7160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2429 2.7160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2429 1.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2429 1.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3863 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3863 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1008 1.8910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1008 1.8910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8152 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8152 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8152 0.6535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8152 0.6535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1008 0.2410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1008 0.2410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3863 0.6535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3863 0.6535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5297 1.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5297 1.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2442 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2442 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2442 0.6535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2442 0.6535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5297 0.2410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5297 0.2410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1008 -0.4198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1008 -0.4198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.9586 1.8910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.9586 1.8910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7873 0.2080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7873 0.2080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5297 -0.4857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5297 -0.4857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6510 3.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6510 3.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9573 3.8543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9573 3.8543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6554 2.6799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6554 2.6799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0255 2.2137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0255 2.2137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7880 2.0753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7880 2.0753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4175 1.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4175 1.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0986 2.0078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0986 2.0078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2850 2.1463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2850 2.1463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0548 2.6337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0548 2.6337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8743 2.3537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8743 2.3537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3475 1.3008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3475 1.3008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9819 1.4360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9819 1.4360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0238 0.8912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0238 0.8912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5845 4.3696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5845 4.3696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9971 3.6549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9971 3.6549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2036 3.8817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2036 3.8817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5947 3.6724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5947 3.6724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0074 4.3871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0074 4.3871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2138 4.1604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2138 4.1604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1062 4.0684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1062 4.0684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6556 4.6022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6556 4.6022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5374 3.8571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5374 3.8571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1493 3.8210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1493 3.8210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4225 3.3478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4225 3.3478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4250 3.9723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4250 3.9723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8253 4.5720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8253 4.5720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2489 3.9723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2489 3.9723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6609 3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6609 3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4848 3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4848 3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8973 2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8973 2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7223 2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7223 2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1348 3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1348 3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7223 3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7223 3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8973 3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8973 3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9586 3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9586 3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1342 4.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1342 4.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9581 4.6867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9581 4.6867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1342 1.8308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1342 1.8308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9581 1.8308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9581 1.8308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7389 -1.8317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7389 -1.8317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3270 -2.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3270 -2.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7389 -3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7389 -3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5031 -2.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5031 -2.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0911 -3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0911 -3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2672 -3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2672 -3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1453 -3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1453 -3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9703 -3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9703 -3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3828 -3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3828 -3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9703 -2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9703 -2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1453 -2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1453 -2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2066 -3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2066 -3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3822 -1.8308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3822 -1.8308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2061 -1.8308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2061 -1.8308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3822 -4.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3822 -4.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2061 -4.6867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2061 -4.6867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2737 -1.6944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2737 -1.6944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4747 -1.9011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4747 -1.9011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0643 -1.1850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0643 -1.1850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3463 -0.7781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3463 -0.7781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1453 -0.5714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1453 -0.5714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5558 -1.2874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5558 -1.2874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0837 -0.9887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0837 -0.9887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6176 -1.1368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6176 -1.1368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2677 -2.3778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2677 -2.3778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9739 -2.0395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9739 -2.0395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 24 21 1 0 0 0 0 | + | 24 21 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 35 22 1 0 0 0 0 | + | 35 22 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 54 49 1 0 0 0 0 | + | 54 49 1 0 0 0 0 |
| − | 52 55 1 0 0 0 0 | + | 52 55 1 0 0 0 0 |
| − | 53 56 1 0 0 0 0 | + | 53 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 51 58 1 0 0 0 0 | + | 51 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 61 62 2 0 0 0 0 | + | 61 62 2 0 0 0 0 |
| − | 61 63 1 0 0 0 0 | + | 61 63 1 0 0 0 0 |
| − | 63 64 2 0 0 0 0 | + | 63 64 2 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 65 66 2 0 0 0 0 | + | 65 66 2 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | 67 68 2 0 0 0 0 | + | 67 68 2 0 0 0 0 |
| − | 68 69 1 0 0 0 0 | + | 68 69 1 0 0 0 0 |
| − | 69 70 2 0 0 0 0 | + | 69 70 2 0 0 0 0 |
| − | 70 65 1 0 0 0 0 | + | 70 65 1 0 0 0 0 |
| − | 68 71 1 0 0 0 0 | + | 68 71 1 0 0 0 0 |
| − | 69 72 1 0 0 0 0 | + | 69 72 1 0 0 0 0 |
| − | 72 73 1 0 0 0 0 | + | 72 73 1 0 0 0 0 |
| − | 67 74 1 0 0 0 0 | + | 67 74 1 0 0 0 0 |
| − | 74 75 1 0 0 0 0 | + | 74 75 1 0 0 0 0 |
| − | 76 77 1 1 0 0 0 | + | 76 77 1 1 0 0 0 |
| − | 77 78 1 1 0 0 0 | + | 77 78 1 1 0 0 0 |
| − | 79 78 1 1 0 0 0 | + | 79 78 1 1 0 0 0 |
| − | 79 80 1 0 0 0 0 | + | 79 80 1 0 0 0 0 |
| − | 80 81 1 0 0 0 0 | + | 80 81 1 0 0 0 0 |
| − | 81 76 1 0 0 0 0 | + | 81 76 1 0 0 0 0 |
| − | 81 82 1 0 0 0 0 | + | 81 82 1 0 0 0 0 |
| − | 82 83 1 0 0 0 0 | + | 82 83 1 0 0 0 0 |
| − | 76 84 1 0 0 0 0 | + | 76 84 1 0 0 0 0 |
| − | 77 85 1 0 0 0 0 | + | 77 85 1 0 0 0 0 |
| − | 79 19 1 0 0 0 0 | + | 79 19 1 0 0 0 0 |
| − | 60 78 1 0 0 0 0 | + | 60 78 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGS0120 | + | ID FL5FACGS0120 |
| − | KNApSAcK_ID C00013892 | + | KNApSAcK_ID C00013892 |
| − | NAME Quercetin 3-(2''-sinapoylglucoside)-3'-(6''-sinapoylglucoside)-4'-glucoside;2-[4-(beta-D-Glucopyranosyloxy)-3-[[6-O-[(2E)-3-(4-hydroxy-3,5-dimethoxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]phenyl]-5,7-dihydroxy-3-[[2-O-[(2E)-3-(4-hydroxy-3,5-dimethoxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-4H-1-benzopyran-4-one | + | NAME Quercetin 3-(2''-sinapoylglucoside)-3'-(6''-sinapoylglucoside)-4'-glucoside;2-[4-(beta-D-Glucopyranosyloxy)-3-[[6-O-[(2E)-3-(4-hydroxy-3,5-dimethoxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]phenyl]-5,7-dihydroxy-3-[[2-O-[(2E)-3-(4-hydroxy-3,5-dimethoxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-4H-1-benzopyran-4-one |
| − | CAS_RN 352639-77-1 | + | CAS_RN 352639-77-1 |
| − | FORMULA C55H60O30 | + | FORMULA C55H60O30 |
| − | EXACTMASS 1200.31694058 | + | EXACTMASS 1200.31694058 |
| − | AVERAGEMASS 1201.0468999999998 | + | AVERAGEMASS 1201.0468999999998 |
| − | SMILES C(=O)(OC(C(OC(=C4c(c5)ccc(OC(C(O)8)OC(C(O)C(O)8)CO)c5OC(C7O)OC(C(C7O)O)COC(C=Cc(c6)cc(OC)c(O)c(OC)6)=O)C(c(c(O)3)c(O4)cc(O)c3)=O)2)C(C(C(O2)CO)O)O)C=Cc(c1)cc(OC)c(c1OC)O | + | SMILES C(=O)(OC(C(OC(=C4c(c5)ccc(OC(C(O)8)OC(C(O)C(O)8)CO)c5OC(C7O)OC(C(C7O)O)COC(C=Cc(c6)cc(OC)c(O)c(OC)6)=O)C(c(c(O)3)c(O4)cc(O)c3)=O)2)C(C(C(O2)CO)O)O)C=Cc(c1)cc(OC)c(c1OC)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
85 92 0 0 0 0 0 0 0 0999 V2000
-1.9573 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6718 1.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6718 2.7160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9573 3.1285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2429 2.7160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2429 1.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3863 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1008 1.8910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8152 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8152 0.6535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1008 0.2410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3863 0.6535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5297 1.8910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.2442 1.4785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.2442 0.6535 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5297 0.2410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1008 -0.4198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.9586 1.8910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7873 0.2080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5297 -0.4857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6510 3.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9573 3.8543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6554 2.6799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0255 2.2137 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7880 2.0753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4175 1.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0986 2.0078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2850 2.1463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0548 2.6337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8743 2.3537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3475 1.3008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9819 1.4360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0238 0.8912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5845 4.3696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9971 3.6549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2036 3.8817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5947 3.6724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0074 4.3871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2138 4.1604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1062 4.0684 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6556 4.6022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5374 3.8571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1493 3.8210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4225 3.3478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4250 3.9723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8253 4.5720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2489 3.9723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6609 3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4848 3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8973 2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7223 2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1348 3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7223 3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8973 3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9586 3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1342 4.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.9581 4.6867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1342 1.8308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.9581 1.8308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7389 -1.8317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3270 -2.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7389 -3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5031 -2.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0911 -3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2672 -3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1453 -3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9703 -3.9732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3828 -3.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9703 -2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1453 -2.5443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2066 -3.2588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3822 -1.8308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2061 -1.8308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3822 -4.6867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2061 -4.6867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2737 -1.6944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4747 -1.9011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0643 -1.1850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3463 -0.7781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1453 -0.5714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5558 -1.2874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0837 -0.9887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6176 -1.1368 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2677 -2.3778 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9739 -2.0395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
28 30 1 0 0 0 0
27 31 1 0 0 0 0
26 32 1 0 0 0 0
32 33 1 0 0 0 0
24 21 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
34 40 1 0 0 0 0
39 41 1 0 0 0 0
38 42 1 0 0 0 0
37 43 1 0 0 0 0
43 44 1 0 0 0 0
35 22 1 0 0 0 0
45 46 2 0 0 0 0
45 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 53 1 0 0 0 0
53 54 2 0 0 0 0
54 49 1 0 0 0 0
52 55 1 0 0 0 0
53 56 1 0 0 0 0
56 57 1 0 0 0 0
51 58 1 0 0 0 0
58 59 1 0 0 0 0
44 45 1 0 0 0 0
60 61 1 0 0 0 0
61 62 2 0 0 0 0
61 63 1 0 0 0 0
63 64 2 0 0 0 0
64 65 1 0 0 0 0
65 66 2 0 0 0 0
66 67 1 0 0 0 0
67 68 2 0 0 0 0
68 69 1 0 0 0 0
69 70 2 0 0 0 0
70 65 1 0 0 0 0
68 71 1 0 0 0 0
69 72 1 0 0 0 0
72 73 1 0 0 0 0
67 74 1 0 0 0 0
74 75 1 0 0 0 0
76 77 1 1 0 0 0
77 78 1 1 0 0 0
79 78 1 1 0 0 0
79 80 1 0 0 0 0
80 81 1 0 0 0 0
81 76 1 0 0 0 0
81 82 1 0 0 0 0
82 83 1 0 0 0 0
76 84 1 0 0 0 0
77 85 1 0 0 0 0
79 19 1 0 0 0 0
60 78 1 0 0 0 0
S SKP 8
ID FL5FACGS0120
KNApSAcK_ID C00013892
NAME Quercetin 3-(2''-sinapoylglucoside)-3'-(6''-sinapoylglucoside)-4'-glucoside;2-[4-(beta-D-Glucopyranosyloxy)-3-[[6-O-[(2E)-3-(4-hydroxy-3,5-dimethoxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]phenyl]-5,7-dihydroxy-3-[[2-O-[(2E)-3-(4-hydroxy-3,5-dimethoxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-4H-1-benzopyran-4-one
CAS_RN 352639-77-1
FORMULA C55H60O30
EXACTMASS 1200.31694058
AVERAGEMASS 1201.0468999999998
SMILES C(=O)(OC(C(OC(=C4c(c5)ccc(OC(C(O)8)OC(C(O)C(O)8)CO)c5OC(C7O)OC(C(C7O)O)COC(C=Cc(c6)cc(OC)c(O)c(OC)6)=O)C(c(c(O)3)c(O4)cc(O)c3)=O)2)C(C(C(O2)CO)O)O)C=Cc(c1)cc(OC)c(c1OC)O
M END