Mol:FL5FADGL0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.5691 0.7184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5691 0.7184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5691 0.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5691 0.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0128 -0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0128 -0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4565 0.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4565 0.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4565 0.7184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4565 0.7184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0128 1.0396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0128 1.0396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9002 -0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9002 -0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3439 0.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3439 0.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3439 0.7184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3439 0.7184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9002 1.0396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9002 1.0396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9002 -0.7459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9002 -0.7459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3566 1.1633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3566 1.1633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9236 0.8359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9236 0.8359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4906 1.1633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4906 1.1633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4906 1.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4906 1.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9236 2.1453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9236 2.1453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3566 1.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3566 1.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0128 -0.8872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0128 -0.8872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3567 2.3178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3567 2.3178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2336 1.1021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2336 1.1021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5060 -0.4121 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.5060 -0.4121 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.1983 -0.9451 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.1983 -0.9451 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.7900 -0.7760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7900 -0.7760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3854 -0.9451 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.3854 -0.9451 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.6932 -0.4121 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.6932 -0.4121 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1014 -0.5812 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.1014 -0.5812 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.9345 -0.5653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9345 -0.5653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1438 -0.0957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1438 -0.0957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2336 -0.5569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2336 -0.5569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8967 -0.7067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8967 -0.7067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8557 -2.3162 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 4.8557 -2.3162 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 5.0687 -1.6550 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 5.0687 -1.6550 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.5298 -1.2950 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.5298 -1.2950 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.1906 -0.8213 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.1906 -0.8213 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.0863 -1.4111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0863 -1.4111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6217 -1.7894 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.6217 -1.7894 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 4.3655 -2.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3655 -2.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4698 -2.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4698 -2.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8075 -0.6785 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8075 -0.6785 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4129 -0.8894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4129 -0.8894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2679 -0.6958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2679 -0.6958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2070 2.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2070 2.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6484 3.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6484 3.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6797 -2.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6797 -2.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4995 -3.1087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4995 -3.1087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 40 30 1 0 0 0 0 | + | 40 30 1 0 0 0 0 |
| − | 22 41 1 0 0 0 0 | + | 22 41 1 0 0 0 0 |
| − | 41 8 1 0 0 0 0 | + | 41 8 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 44 45 | + | M SAL 2 2 44 45 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 48 2.3693 -1.6961 | + | M SVB 2 48 2.3693 -1.6961 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 46 1.207 2.7214 | + | M SVB 1 46 1.207 2.7214 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FADGL0006 | + | ID FL5FADGL0006 |
| − | KNApSAcK_ID C00005543 | + | KNApSAcK_ID C00005543 |
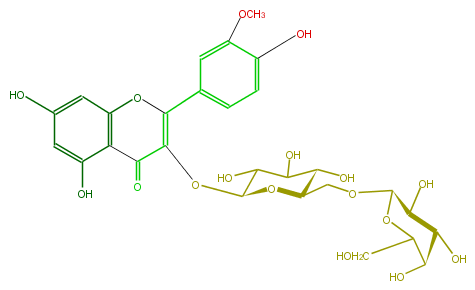
| − | NAME Isorhamnetin 3-galactosyl-(1->6)-glucoside | + | NAME Isorhamnetin 3-galactosyl-(1->6)-glucoside |
| − | CAS_RN 32472-27-8 | + | CAS_RN 32472-27-8 |
| − | FORMULA C28H32O17 | + | FORMULA C28H32O17 |
| − | EXACTMASS 640.163949598 | + | EXACTMASS 640.163949598 |
| − | AVERAGEMASS 640.54348 | + | AVERAGEMASS 640.54348 |
| − | SMILES O=C(c23)C(O[C@@H](C(O)4)O[C@H](CO[C@@H]([C@@H](O)5)OC(CO)[C@H](O)[C@@H]5O)[C@H](O)C4O)=C(Oc2cc(O)cc3O)c(c1)cc(OC)c(O)c1 | + | SMILES O=C(c23)C(O[C@@H](C(O)4)O[C@H](CO[C@@H]([C@@H](O)5)OC(CO)[C@H](O)[C@@H]5O)[C@H](O)C4O)=C(Oc2cc(O)cc3O)c(c1)cc(OC)c(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
-2.5691 0.7184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5691 0.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0128 -0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4565 0.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4565 0.7184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0128 1.0396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9002 -0.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3439 0.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3439 0.7184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9002 1.0396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9002 -0.7459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3566 1.1633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9236 0.8359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4906 1.1633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4906 1.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9236 2.1453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3566 1.8179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0128 -0.8872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3567 2.3178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2336 1.1021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5060 -0.4121 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.1983 -0.9451 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.7900 -0.7760 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3854 -0.9451 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.6932 -0.4121 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1014 -0.5812 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.9345 -0.5653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1438 -0.0957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2336 -0.5569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8967 -0.7067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8557 -2.3162 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
5.0687 -1.6550 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.5298 -1.2950 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.1906 -0.8213 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.0863 -1.4111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6217 -1.7894 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
4.3655 -2.5447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4698 -2.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8075 -0.6785 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4129 -0.8894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2679 -0.6958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2070 2.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6484 3.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6797 -2.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4995 -3.1087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
40 30 1 0 0 0 0
22 41 1 0 0 0 0
41 8 1 0 0 0 0
16 42 1 0 0 0 0
42 43 1 0 0 0 0
36 44 1 0 0 0 0
44 45 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 44 45
M SBL 2 1 48
M SMT 2 CH2OH
M SVB 2 48 2.3693 -1.6961
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 46
M SMT 1 OCH3
M SVB 1 46 1.207 2.7214
S SKP 8
ID FL5FADGL0006
KNApSAcK_ID C00005543
NAME Isorhamnetin 3-galactosyl-(1->6)-glucoside
CAS_RN 32472-27-8
FORMULA C28H32O17
EXACTMASS 640.163949598
AVERAGEMASS 640.54348
SMILES O=C(c23)C(O[C@@H](C(O)4)O[C@H](CO[C@@H]([C@@H](O)5)OC(CO)[C@H](O)[C@@H]5O)[C@H](O)C4O)=C(Oc2cc(O)cc3O)c(c1)cc(OC)c(O)c1
M END