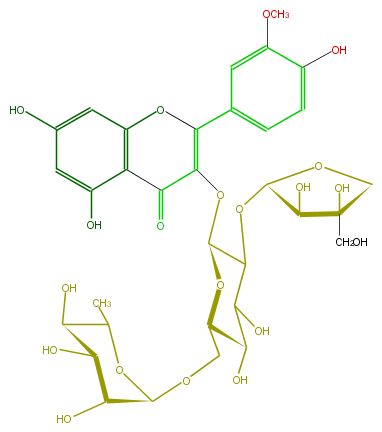
Mol:FL5FADGL0020
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.9815 1.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9815 1.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9815 0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9815 0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2805 -0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2805 -0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5795 0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5795 0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5795 1.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5795 1.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2805 1.5611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2805 1.5611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8784 -0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8784 -0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1774 0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1774 0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1774 1.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1774 1.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8784 1.5611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8784 1.5611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8784 -0.7559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8784 -0.7559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5233 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5233 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2378 1.1485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2378 1.1485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9523 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9523 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9523 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9523 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2378 2.7985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2378 2.7985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5233 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5233 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6823 1.5610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6823 1.5610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3193 -0.1431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3193 -0.1431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2805 -0.7358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2805 -0.7358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6169 2.7697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6169 2.7697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8115 -1.5517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8115 -1.5517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0771 -1.1277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0771 -1.1277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3101 -1.9431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3101 -1.9431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0771 -2.7633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0771 -2.7633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8219 -3.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8219 -3.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5785 -2.3720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5785 -2.3720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6982 -0.4258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6982 -0.4258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0719 -2.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0719 -2.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6438 -3.8328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6438 -3.8328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2211 0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2211 0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8944 -0.5496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8944 -0.5496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6782 -0.5289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6782 -0.5289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3382 0.0486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3382 0.0486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2863 0.4404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2863 0.4404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8944 0.0294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8944 0.0294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6782 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6782 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2847 -3.3602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2847 -3.3602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9652 -4.2770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9652 -4.2770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0511 -4.2770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0511 -4.2770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7081 -3.6415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7081 -3.6415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9327 -2.7501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9327 -2.7501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8469 -2.7499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8469 -2.7499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1899 -3.3857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1899 -3.3857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7837 -1.9873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7837 -1.9873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1752 -2.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1752 -2.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0244 -3.2272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0244 -3.2272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7421 -4.6237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7421 -4.6237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3711 -3.8556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3711 -3.8556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2378 3.4929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2378 3.4929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7939 4.6237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7939 4.6237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6782 -1.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6782 -1.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6823 -1.3560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6823 -1.3560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 31 1 0 0 0 0 | + | 35 31 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 40 39 1 1 0 0 0 | + | 40 39 1 1 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 39 1 1 0 0 0 | + | 44 39 1 1 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 42 46 1 0 0 0 0 | + | 42 46 1 0 0 0 0 |
| − | 44 47 1 0 0 0 0 | + | 44 47 1 0 0 0 0 |
| − | 39 48 1 0 0 0 0 | + | 39 48 1 0 0 0 0 |
| − | 40 49 1 0 0 0 0 | + | 40 49 1 0 0 0 0 |
| − | 49 38 1 0 0 0 0 | + | 49 38 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 16 50 1 0 0 0 0 | + | 16 50 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 33 52 1 0 0 0 0 | + | 33 52 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 56 | + | M SBL 1 1 56 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 56 0.0000 -0.6945 | + | M SBV 1 56 0.0000 -0.6945 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 52 53 | + | M SAL 2 2 52 53 |
| − | M SBL 2 1 58 | + | M SBL 2 1 58 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 58 0.0000 0.5573 | + | M SBV 2 58 0.0000 0.5573 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FADGL0020 | + | ID FL5FADGL0020 |
| − | FORMULA C33H40O20 | + | FORMULA C33H40O20 |
| − | EXACTMASS 756.21129372 | + | EXACTMASS 756.21129372 |
| − | AVERAGEMASS 756.6587 | + | AVERAGEMASS 756.6587 |
| − | SMILES C(OC(O6)C(C(O)(CO)C6)O)(C1OC(C(=O)4)=C(c(c5)ccc(O)c5OC)Oc(c43)cc(O)cc(O)3)C(O)C(C(COC(C(O)2)OC(C)C(O)C(O)2)O1)O | + | SMILES C(OC(O6)C(C(O)(CO)C6)O)(C1OC(C(=O)4)=C(c(c5)ccc(O)c5OC)Oc(c43)cc(O)cc(O)3)C(O)C(C(COC(C(O)2)OC(C)C(O)C(O)2)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-2.9815 1.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9815 0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2805 -0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5795 0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5795 1.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2805 1.5611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8784 -0.0578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1774 0.3470 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1774 1.1564 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8784 1.5611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8784 -0.7559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5233 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2378 1.1485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9523 1.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9523 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2378 2.7985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5233 2.3860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6823 1.5610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3193 -0.1431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2805 -0.7358 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6169 2.7697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8115 -1.5517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0771 -1.1277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3101 -1.9431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0771 -2.7633 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8219 -3.2102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5785 -2.3720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6982 -0.4258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0719 -2.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6438 -3.8328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2211 0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8944 -0.5496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6782 -0.5289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3382 0.0486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2863 0.4404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8944 0.0294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6782 0.0089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2847 -3.3602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9652 -4.2770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0511 -4.2770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7081 -3.6415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9327 -2.7501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8469 -2.7499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1899 -3.3857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7837 -1.9873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1752 -2.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0244 -3.2272 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7421 -4.6237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3711 -3.8556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2378 3.4929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7939 4.6237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6782 -1.0862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6823 -1.3560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 15 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 19 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 31 1 0 0 0 0
32 36 1 0 0 0 0
33 37 1 0 0 0 0
28 31 1 0 0 0 0
25 38 1 0 0 0 0
40 39 1 1 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 1 0 0 0
44 39 1 1 0 0 0
43 45 1 0 0 0 0
42 46 1 0 0 0 0
44 47 1 0 0 0 0
39 48 1 0 0 0 0
40 49 1 0 0 0 0
49 38 1 0 0 0 0
50 51 1 0 0 0 0
16 50 1 0 0 0 0
52 53 1 0 0 0 0
33 52 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 56
M SMT 1 OCH3
M SBV 1 56 0.0000 -0.6945
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 52 53
M SBL 2 1 58
M SMT 2 CH2OH
M SBV 2 58 0.0000 0.5573
S SKP 5
ID FL5FADGL0020
FORMULA C33H40O20
EXACTMASS 756.21129372
AVERAGEMASS 756.6587
SMILES C(OC(O6)C(C(O)(CO)C6)O)(C1OC(C(=O)4)=C(c(c5)ccc(O)c5OC)Oc(c43)cc(O)cc(O)3)C(O)C(C(COC(C(O)2)OC(C)C(O)C(O)2)O1)O
M END