Mol:FL5FCAGA0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.1547 -4.8607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1547 -4.8607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4780 -4.7492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4780 -4.7492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6976 -4.1455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6976 -4.1455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2848 -3.6534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2848 -3.6534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3479 -3.7649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3479 -3.7649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5676 -4.3685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5676 -4.3685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5044 -3.0498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5044 -3.0498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0916 -2.5577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0916 -2.5577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5411 -2.6692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5411 -2.6692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7608 -3.2729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7608 -3.2729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9976 -2.9627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9976 -2.9627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9539 -2.1773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9539 -2.1773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7299 -1.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7299 -1.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1508 -1.0607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1508 -1.0607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7955 -1.1743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7955 -1.1743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0194 -1.7896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0194 -1.7896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5986 -2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5986 -2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3299 -4.0339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3299 -4.0339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5209 -1.8239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5209 -1.8239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9716 -0.4733 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.9716 -0.4733 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5840 -1.1448 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.5840 -1.1448 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3294 -0.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3294 -0.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0794 -1.1448 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.0794 -1.1448 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4671 -0.4733 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4671 -0.4733 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.7216 -0.6863 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.7216 -0.6863 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.2445 -0.6682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2445 -0.6682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9862 -0.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9862 -0.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2311 0.4076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2311 0.4076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2162 -0.6730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2162 -0.6730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7020 -5.3199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7020 -5.3199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4681 -5.9627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4681 -5.9627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5899 -1.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5899 -1.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3044 -1.5310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3044 -1.5310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 19 8 1 0 0 0 0 | + | 19 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 15 29 1 0 0 0 0 | + | 15 29 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 23 32 1 0 0 0 0 | + | 23 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 32 33 | + | M SAL 2 2 32 33 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 35 2.5899 -1.1185 | + | M SVB 2 35 2.5899 -1.1185 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 33 -3.3044 0.8467 | + | M SVB 1 33 -3.3044 0.8467 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FCAGA0001 | + | ID FL5FCAGA0001 |
| − | KNApSAcK_ID C00005270 | + | KNApSAcK_ID C00005270 |
| − | NAME Rhamnocitrin 3-galactoside | + | NAME Rhamnocitrin 3-galactoside |
| − | CAS_RN 99878-05-4 | + | CAS_RN 99878-05-4 |
| − | FORMULA C22H22O11 | + | FORMULA C22H22O11 |
| − | EXACTMASS 462.116211546 | + | EXACTMASS 462.116211546 |
| − | AVERAGEMASS 462.40348000000006 | + | AVERAGEMASS 462.40348000000006 |
| − | SMILES c(c4)(ccc(c4)C(O2)=C(C(=O)c(c3O)c2cc(c3)OC)O[C@H](O1)C(O)C([C@H]([C@@H](CO)1)O)O)O | + | SMILES c(c4)(ccc(c4)C(O2)=C(C(=O)c(c3O)c2cc(c3)OC)O[C@H](O1)C(O)C([C@H]([C@@H](CO)1)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-0.1547 -4.8607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4780 -4.7492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6976 -4.1455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2848 -3.6534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3479 -3.7649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5676 -4.3685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5044 -3.0498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0916 -2.5577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5411 -2.6692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7608 -3.2729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9976 -2.9627 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9539 -2.1773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7299 -1.5622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1508 -1.0607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7955 -1.1743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0194 -1.7896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5986 -2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3299 -4.0339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5209 -1.8239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9716 -0.4733 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.5840 -1.1448 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3294 -0.9317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0794 -1.1448 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4671 -0.4733 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.7216 -0.6863 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.2445 -0.6682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9862 -0.2281 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2311 0.4076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2162 -0.6730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7020 -5.3199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4681 -5.9627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5899 -1.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3044 -1.5310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
21 19 1 0 0 0 0
19 8 1 0 0 0 0
23 22 1 1 0 0 0
15 29 1 0 0 0 0
1 30 1 0 0 0 0
30 31 1 0 0 0 0
23 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 32 33
M SBL 2 1 35
M SMT 2 CH2OH
M SVB 2 35 2.5899 -1.1185
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 33
M SMT 1 OCH3
M SVB 1 33 -3.3044 0.8467
S SKP 8
ID FL5FCAGA0001
KNApSAcK_ID C00005270
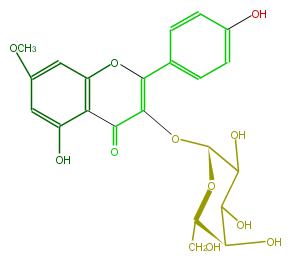
NAME Rhamnocitrin 3-galactoside
CAS_RN 99878-05-4
FORMULA C22H22O11
EXACTMASS 462.116211546
AVERAGEMASS 462.40348000000006
SMILES c(c4)(ccc(c4)C(O2)=C(C(=O)c(c3O)c2cc(c3)OC)O[C@H](O1)C(O)C([C@H]([C@@H](CO)1)O)O)O
M END