Mol:FL5FCAGL0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2694 1.3287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2694 1.3287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2694 0.6864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2694 0.6864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7131 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7131 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1568 0.6864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1568 0.6864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1568 1.3287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1568 1.3287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7131 1.6499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7131 1.6499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6005 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6005 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0442 0.6864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0442 0.6864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0442 1.3287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0442 1.3287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6005 1.6499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6005 1.6499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6005 -0.1357 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6005 -0.1357 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5119 1.6498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5119 1.6498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0789 1.3224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0789 1.3224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6459 1.6498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6459 1.6498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6459 2.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6459 2.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0789 2.6318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0789 2.6318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5119 2.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5119 2.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3500 0.2975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3500 0.2975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7131 -0.2770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7131 -0.2770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2798 2.6705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2798 2.6705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7406 -0.8203 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.7406 -0.8203 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.1578 -0.4838 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.1578 -0.4838 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.3427 -1.1309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3427 -1.1309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1578 -1.7819 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.1578 -1.7819 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.7406 -2.1184 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.7406 -2.1184 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5557 -1.4713 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.5557 -1.4713 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5731 -0.1954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5731 -0.1954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0172 -1.7377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0172 -1.7377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0764 -3.0603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0764 -3.0603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7064 -0.8729 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.7064 -0.8729 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.4318 -0.8729 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4318 -0.8729 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.9104 -0.3685 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.9104 -0.3685 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.7322 0.3389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7322 0.3389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0067 0.3390 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.0067 0.3390 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.5281 -0.1655 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.5281 -0.1655 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1952 0.2197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1952 0.2197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1472 -1.3137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1472 -1.3137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5834 -1.4386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5834 -1.4386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6266 -0.3685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6266 -0.3685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6266 1.9475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6266 1.9475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1265 2.8136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1265 2.8136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0169 -2.1298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0169 -2.1298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9735 -2.2681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9735 -2.2681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 18 1 0 0 0 0 | + | 22 18 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 30 37 1 0 0 0 0 | + | 30 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 27 34 1 0 0 0 0 | + | 27 34 1 0 0 0 0 |
| − | 1 40 1 0 0 0 0 | + | 1 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 24 42 1 0 0 0 0 | + | 24 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 0.0794 -2.1491 | + | M SVB 2 46 0.0794 -2.1491 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 44 -2.6266 1.9475 | + | M SVB 1 44 -2.6266 1.9475 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FCAGL0003 | + | ID FL5FCAGL0003 |
| − | KNApSAcK_ID C00005278 | + | KNApSAcK_ID C00005278 |
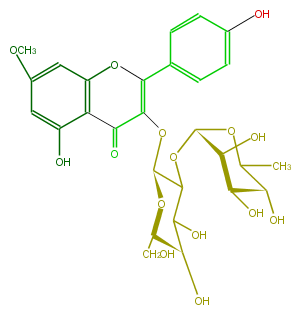
| − | NAME Rhamnocitrin 3-neohesperidoside | + | NAME Rhamnocitrin 3-neohesperidoside |
| − | CAS_RN 129885-17-2 | + | CAS_RN 129885-17-2 |
| − | FORMULA C28H32O15 | + | FORMULA C28H32O15 |
| − | EXACTMASS 608.174120354 | + | EXACTMASS 608.174120354 |
| − | AVERAGEMASS 608.54468 | + | AVERAGEMASS 608.54468 |
| − | SMILES [C@@H]([C@H](O)1)([C@@H](C(C)O[C@@H]1OC(C(O)5)[C@@H](O[C@H](CO)[C@H](O)5)OC(C(=O)2)=C(c(c4)ccc(c4)O)Oc(c3)c(c(O)cc3OC)2)O)O | + | SMILES [C@@H]([C@H](O)1)([C@@H](C(C)O[C@@H]1OC(C(O)5)[C@@H](O[C@H](CO)[C@H](O)5)OC(C(=O)2)=C(c(c4)ccc(c4)O)Oc(c3)c(c(O)cc3OC)2)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.2694 1.3287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2694 0.6864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7131 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1568 0.6864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1568 1.3287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7131 1.6499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6005 0.3652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0442 0.6864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0442 1.3287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6005 1.6499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6005 -0.1357 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5119 1.6498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0789 1.3224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6459 1.6498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6459 2.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0789 2.6318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5119 2.3045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3500 0.2975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7131 -0.2770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2798 2.6705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7406 -0.8203 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.1578 -0.4838 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.3427 -1.1309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1578 -1.7819 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.7406 -2.1184 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5557 -1.4713 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.5731 -0.1954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0172 -1.7377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0764 -3.0603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7064 -0.8729 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.4318 -0.8729 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.9104 -0.3685 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.7322 0.3389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0067 0.3390 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.5281 -0.1655 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1952 0.2197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1472 -1.3137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5834 -1.4386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6266 -0.3685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6266 1.9475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1265 2.8136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0169 -2.1298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9735 -2.2681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
8 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 18 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
35 36 1 0 0 0 0
30 37 1 0 0 0 0
31 38 1 0 0 0 0
32 39 1 0 0 0 0
27 34 1 0 0 0 0
1 40 1 0 0 0 0
40 41 1 0 0 0 0
24 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 0.0794 -2.1491
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 OCH3
M SVB 1 44 -2.6266 1.9475
S SKP 8
ID FL5FCAGL0003
KNApSAcK_ID C00005278
NAME Rhamnocitrin 3-neohesperidoside
CAS_RN 129885-17-2
FORMULA C28H32O15
EXACTMASS 608.174120354
AVERAGEMASS 608.54468
SMILES [C@@H]([C@H](O)1)([C@@H](C(C)O[C@@H]1OC(C(O)5)[C@@H](O[C@H](CO)[C@H](O)5)OC(C(=O)2)=C(c(c4)ccc(c4)O)Oc(c3)c(c(O)cc3OC)2)O)O
M END