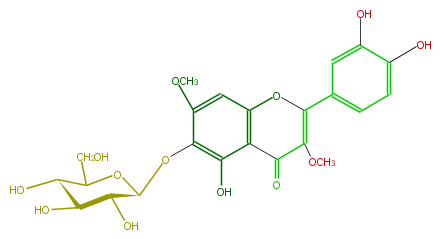
Mol:FL5FECGS0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5489 0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5489 0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5489 -0.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5489 -0.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0074 -0.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0074 -0.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5637 -0.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5637 -0.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5637 0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5637 0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0074 0.4870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0074 0.4870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1200 -0.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1200 -0.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6763 -0.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6763 -0.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6763 0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6763 0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1200 0.4870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1200 0.4870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1200 -1.2985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1200 -1.2985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2324 0.4869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2324 0.4869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7993 0.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7993 0.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3663 0.4869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3663 0.4869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3663 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3663 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7993 1.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7993 1.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2324 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2324 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0074 -1.4398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0074 -1.4398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0002 1.5076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0002 1.5076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7993 2.1235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7993 2.1235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1050 -0.7976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1050 -0.7976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2617 -1.2076 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.2617 -1.2076 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.8348 -1.7712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.8348 -1.7712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.2200 -1.5321 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.2200 -1.5321 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6268 -1.5257 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.6268 -1.5257 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.0578 -1.0945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0578 -1.0945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6859 -1.3200 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.6859 -1.3200 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.0002 -1.4055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0002 -1.4055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5003 -1.8036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5003 -1.8036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8677 -2.1235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8677 -2.1235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8114 -0.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8114 -0.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9546 0.0803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9546 0.0803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9062 0.7846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9062 0.7846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4063 1.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4063 1.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8369 -0.8390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8369 -0.8390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6484 -0.6904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6484 -0.6904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 21 1 0 0 0 0 | + | 25 21 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 1 33 1 0 0 0 0 | + | 1 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 8 35 1 0 0 0 0 | + | 8 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 31 32 | + | M SAL 3 2 31 32 |
| − | M SBL 3 1 34 | + | M SBL 3 1 34 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 34 -3.1259 -0.9105 | + | M SVB 3 34 -3.1259 -0.9105 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 35 36 | + | M SAL 2 2 35 36 |
| − | M SBL 2 1 38 | + | M SBL 2 1 38 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 38 1.6686 -0.873 | + | M SVB 2 38 1.6686 -0.873 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 33 34 | + | M SAL 1 2 33 34 |
| − | M SBL 1 1 36 | + | M SBL 1 1 36 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 36 -0.9062 0.7846 | + | M SVB 1 36 -0.9062 0.7846 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FECGS0016 | + | ID FL5FECGS0016 |
| − | KNApSAcK_ID C00005658 | + | KNApSAcK_ID C00005658 |
| − | NAME Tomentin 6-glucoside | + | NAME Tomentin 6-glucoside |
| − | CAS_RN 64190-90-5 | + | CAS_RN 64190-90-5 |
| − | FORMULA C23H24O13 | + | FORMULA C23H24O13 |
| − | EXACTMASS 508.121690854 | + | EXACTMASS 508.121690854 |
| − | AVERAGEMASS 508.42886 | + | AVERAGEMASS 508.42886 |
| − | SMILES O=C(C(OC)=3)c(c(O)1)c(OC3c(c4)ccc(c4O)O)cc(OC)c1O[C@H](O2)[C@@H](O)[C@H]([C@H](C(CO)2)O)O | + | SMILES O=C(C(OC)=3)c(c(O)1)c(OC3c(c4)ccc(c4O)O)cc(OC)c1O[C@H](O2)[C@@H](O)[C@H]([C@H](C(CO)2)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-0.5489 0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5489 -0.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0074 -0.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5637 -0.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5637 0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0074 0.4870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1200 -0.7977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6763 -0.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6763 0.1659 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1200 0.4870 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1200 -1.2985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2324 0.4869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7993 0.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3663 0.4869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3663 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7993 1.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2324 1.1416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0074 -1.4398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0002 1.5076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7993 2.1235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1050 -0.7976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2617 -1.2076 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.8348 -1.7712 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.2200 -1.5321 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6268 -1.5257 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.0578 -1.0945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6859 -1.3200 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.0002 -1.4055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5003 -1.8036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8677 -2.1235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8114 -0.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9546 0.0803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9062 0.7846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4063 1.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8369 -0.8390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6484 -0.6904 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
16 20 1 0 0 0 0
2 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 21 1 0 0 0 0
27 31 1 0 0 0 0
31 32 1 0 0 0 0
1 33 1 0 0 0 0
33 34 1 0 0 0 0
8 35 1 0 0 0 0
35 36 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 31 32
M SBL 3 1 34
M SMT 3 CH2OH
M SVB 3 34 -3.1259 -0.9105
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 35 36
M SBL 2 1 38
M SMT 2 OCH3
M SVB 2 38 1.6686 -0.873
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 33 34
M SBL 1 1 36
M SMT 1 OCH3
M SVB 1 36 -0.9062 0.7846
S SKP 8
ID FL5FECGS0016
KNApSAcK_ID C00005658
NAME Tomentin 6-glucoside
CAS_RN 64190-90-5
FORMULA C23H24O13
EXACTMASS 508.121690854
AVERAGEMASS 508.42886
SMILES O=C(C(OC)=3)c(c(O)1)c(OC3c(c4)ccc(c4O)O)cc(OC)c1O[C@H](O2)[C@@H](O)[C@H]([C@H](C(CO)2)O)O
M END