Mol:FL5FECNI0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 31 33 0 0 0 0 0 0 0 0999 V2000 | + | 31 33 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2400 -0.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2400 -0.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9685 -0.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9685 -0.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3286 -0.8652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3286 -0.8652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0399 -0.3390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0399 -0.3390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2316 0.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2316 0.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8715 0.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8715 0.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6799 -0.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6799 -0.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0483 0.1313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0483 0.1313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7769 0.7134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7769 0.7134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1369 0.7694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1369 0.7694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8916 -0.8489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8916 -0.8489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1451 1.2394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1451 1.2394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7973 1.1823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7973 1.1823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1728 1.7186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1728 1.7186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8961 2.3120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8961 2.3120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2439 2.3690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2439 2.3690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8684 1.8327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8684 1.8327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0572 -1.4471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0572 -1.4471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8024 2.7346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8024 2.7346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8797 -0.1711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8797 -0.1711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2711 -0.7468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2711 -0.7468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9094 -0.6910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9094 -0.6910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2761 -1.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2761 -1.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9131 -1.1590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9131 -1.1590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0059 -1.7942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0059 -1.7942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0445 0.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0445 0.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0407 -0.0429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0407 -0.0429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2573 3.0109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2573 3.0109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2781 4.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2781 4.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6546 -1.0283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6546 -1.0283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8328 -1.1002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8328 -1.1002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 21 20 1 0 0 0 0 | + | 21 20 1 0 0 0 0 |
| − | 8 26 1 0 0 0 0 | + | 8 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 16 28 1 0 0 0 0 | + | 16 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 2 30 1 0 0 0 0 | + | 2 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 30 31 | + | M SAL 3 2 30 31 |
| − | M SBL 3 1 32 | + | M SBL 3 1 32 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 32 -1.5472 -0.6877 | + | M SVB 3 32 -1.5472 -0.6877 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 28 29 | + | M SAL 2 2 28 29 |
| − | M SBL 2 1 30 | + | M SBL 2 1 30 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 30 2.7989 1.7424 | + | M SVB 2 30 2.7989 1.7424 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 26 27 | + | M SAL 1 2 26 27 |
| − | M SBL 1 1 28 | + | M SBL 1 1 28 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 28 1.6027 -0.9641 | + | M SVB 1 28 1.6027 -0.9641 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FECNI0001 | + | ID FL5FECNI0001 |
| − | KNApSAcK_ID C00005016 | + | KNApSAcK_ID C00005016 |
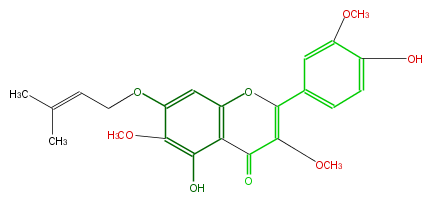
| − | NAME 5,4'-Dihydroxy-3,6,3'-trimethoxy-7-prenyloxyflavone | + | NAME 5,4'-Dihydroxy-3,6,3'-trimethoxy-7-prenyloxyflavone |
| − | CAS_RN 160036-27-1 | + | CAS_RN 160036-27-1 |
| − | FORMULA C23H24O8 | + | FORMULA C23H24O8 |
| − | EXACTMASS 428.14711774399996 | + | EXACTMASS 428.14711774399996 |
| − | AVERAGEMASS 428.43186000000003 | + | AVERAGEMASS 428.43186000000003 |
| − | SMILES O=C(c32)C(OC)=C(Oc(cc(c(c3O)OC)OCC=C(C)C)2)c(c1)cc(OC)c(O)c1 | + | SMILES O=C(c32)C(OC)=C(Oc(cc(c(c3O)OC)OCC=C(C)C)2)c(c1)cc(OC)c(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
31 33 0 0 0 0 0 0 0 0999 V2000
-1.2400 -0.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9685 -0.8092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3286 -0.8652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0399 -0.3390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2316 0.2431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8715 0.2991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6799 -0.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0483 0.1313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7769 0.7134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1369 0.7694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8916 -0.8489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1451 1.2394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7973 1.1823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1728 1.7186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8961 2.3120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2439 2.3690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8684 1.8327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0572 -1.4471 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8024 2.7346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8797 -0.1711 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2711 -0.7468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9094 -0.6910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2761 -1.2147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9131 -1.1590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0059 -1.7942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0445 0.0442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0407 -0.0429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2573 3.0109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2781 4.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6546 -1.0283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8328 -1.1002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
15 19 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
23 25 1 0 0 0 0
21 20 1 0 0 0 0
8 26 1 0 0 0 0
26 27 1 0 0 0 0
16 28 1 0 0 0 0
28 29 1 0 0 0 0
2 30 1 0 0 0 0
30 31 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 30 31
M SBL 3 1 32
M SMT 3 OCH3
M SVB 3 32 -1.5472 -0.6877
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 28 29
M SBL 2 1 30
M SMT 2 OCH3
M SVB 2 30 2.7989 1.7424
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 26 27
M SBL 1 1 28
M SMT 1 OCH3
M SVB 1 28 1.6027 -0.9641
S SKP 8
ID FL5FECNI0001
KNApSAcK_ID C00005016
NAME 5,4'-Dihydroxy-3,6,3'-trimethoxy-7-prenyloxyflavone
CAS_RN 160036-27-1
FORMULA C23H24O8
EXACTMASS 428.14711774399996
AVERAGEMASS 428.43186000000003
SMILES O=C(c32)C(OC)=C(Oc(cc(c(c3O)OC)OCC=C(C)C)2)c(c1)cc(OC)c(O)c1
M END