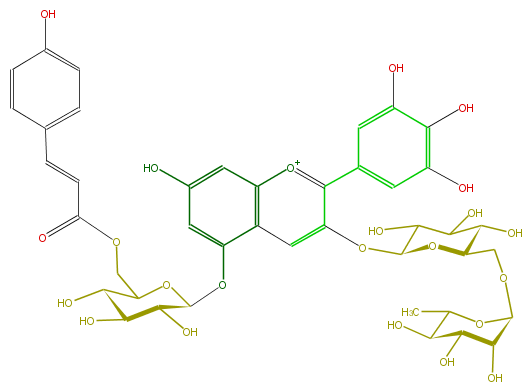
Mol:FL7AAGGA0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 65 71 0 0 0 0 0 0 0 0999 V2000 | + | 65 71 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5418 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5418 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5420 -0.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5420 -0.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8232 -1.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8232 -1.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1046 -0.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1046 -0.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1046 0.1804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1046 0.1804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8233 0.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8233 0.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6142 -1.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6142 -1.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3328 -0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3328 -0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3328 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3328 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6141 0.5954 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.6141 0.5954 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0512 0.5951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0512 0.5951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7838 0.1723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7838 0.1723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5163 0.5952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5163 0.5952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5162 1.4410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5162 1.4410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7837 1.8639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7837 1.8639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0512 1.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0512 1.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2603 0.5952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2603 0.5952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2484 1.8638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2484 1.8638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8232 -1.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8232 -1.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1444 -1.1084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1444 -1.1084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9655 -2.4705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9655 -2.4705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5680 -3.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5680 -3.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2484 -2.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2484 -2.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8997 -3.1547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8997 -3.1547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2972 -2.5870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2972 -2.5870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6168 -2.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6168 -2.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2807 -2.4584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2807 -2.4584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3116 -0.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3116 -0.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9651 -1.2488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9651 -1.2488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6315 -1.0583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6315 -1.0583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3020 -1.2487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3020 -1.2487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6485 -0.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6485 -0.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9822 -0.8390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9822 -0.8390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4916 -0.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4916 -0.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4452 -0.3570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4452 -0.3570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2912 -0.7842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2912 -0.7842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9195 -1.1483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9195 -1.1483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1356 -1.7489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1356 -1.7489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8930 -2.7381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8930 -2.7381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8507 -3.5143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8507 -3.5143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8708 -3.8623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8708 -3.8623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7837 2.7094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7837 2.7094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3551 -2.0057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3551 -2.0057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8754 -2.6386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8754 -2.6386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1849 -2.3700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1849 -2.3700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5187 -2.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5187 -2.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0029 -1.8787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0029 -1.8787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6070 -2.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6070 -2.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0820 -2.2662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0820 -2.2662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6202 -2.6571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6202 -2.6571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5817 -2.8912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5817 -2.8912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0687 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0687 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2484 0.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2484 0.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8882 -0.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8882 -0.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6410 -0.8939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6410 -0.8939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8937 0.2941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8937 0.2941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5743 0.7066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5743 0.7066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5802 1.4194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5802 1.4194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8709 1.8344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8709 1.8344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8749 2.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8749 2.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5878 3.0795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5878 3.0795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2972 2.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2972 2.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2933 1.8344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2933 1.8344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5878 3.8623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5878 3.8623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0742 -0.9861 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0742 -0.9861 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 24 41 1 0 0 0 0 | + | 24 41 1 0 0 0 0 |
| − | 29 20 1 0 0 0 0 | + | 29 20 1 0 0 0 0 |
| − | 15 42 1 0 0 0 0 | + | 15 42 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 48 52 1 0 0 0 0 | + | 48 52 1 0 0 0 0 |
| − | 46 19 1 0 0 0 0 | + | 46 19 1 0 0 0 0 |
| − | 13 53 1 0 0 0 0 | + | 13 53 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 54 56 1 0 0 0 0 | + | 54 56 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 2 0 0 0 0 | + | 58 59 2 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 2 0 0 0 0 | + | 60 61 2 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 62 63 2 0 0 0 0 | + | 62 63 2 0 0 0 0 |
| − | 63 58 1 0 0 0 0 | + | 63 58 1 0 0 0 0 |
| − | 61 64 1 0 0 0 0 | + | 61 64 1 0 0 0 0 |
| − | 52 65 1 0 0 0 0 | + | 52 65 1 0 0 0 0 |
| − | 65 54 1 0 0 0 0 | + | 65 54 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AAGGA0008 | + | ID FL7AAGGA0008 |
| − | FORMULA C42H47O23 | + | FORMULA C42H47O23 |
| − | EXACTMASS 919.25081281 | + | EXACTMASS 919.25081281 |
| − | AVERAGEMASS 919.8087800000001 | + | AVERAGEMASS 919.8087800000001 |
| − | SMILES O(C(Oc(c3)c(c5)c([o+1]c(c5OC(O6)C(C(C(O)C6COC(O7)C(C(C(O)C7C)O)O)O)O)c(c4)cc(c(c4O)O)O)cc3O)1)C(COC(=O)C=Cc(c2)ccc(O)c2)C(O)C(O)C1O | + | SMILES O(C(Oc(c3)c(c5)c([o+1]c(c5OC(O6)C(C(C(O)C6COC(O7)C(C(C(O)C7C)O)O)O)O)c(c4)cc(c(c4O)O)O)cc3O)1)C(COC(=O)C=Cc(c2)ccc(O)c2)C(O)C(O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
65 71 0 0 0 0 0 0 0 0999 V2000
-1.5418 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5420 -0.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8232 -1.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1046 -0.6494 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1046 0.1804 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8233 0.5954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6142 -1.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3328 -0.6495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3328 0.1805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6141 0.5954 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
2.0512 0.5951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7838 0.1723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5163 0.5952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5162 1.4410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7837 1.8639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0512 1.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2603 0.5952 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2484 1.8638 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8232 -1.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1444 -1.1084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9655 -2.4705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5680 -3.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2484 -2.9064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8997 -3.1547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2972 -2.5870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6168 -2.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2807 -2.4584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3116 -0.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9651 -1.2488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6315 -1.0583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3020 -1.2487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6485 -0.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9822 -0.8390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4916 -0.7359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4452 -0.3570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2912 -0.7842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9195 -1.1483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1356 -1.7489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8930 -2.7381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8507 -3.5143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8708 -3.8623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7837 2.7094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3551 -2.0057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8754 -2.6386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1849 -2.3700 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5187 -2.3629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0029 -1.8787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6070 -2.1976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0820 -2.2662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6202 -2.6571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5817 -2.8912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0687 -1.6653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2484 0.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8882 -0.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6410 -0.8939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8937 0.2941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5743 0.7066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5802 1.4194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8709 1.8344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8749 2.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5878 3.0795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2972 2.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2933 1.8344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5878 3.8623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0742 -0.9861 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
25 38 1 0 0 0 0
37 38 1 0 0 0 0
22 39 1 0 0 0 0
23 40 1 0 0 0 0
24 41 1 0 0 0 0
29 20 1 0 0 0 0
15 42 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
48 52 1 0 0 0 0
46 19 1 0 0 0 0
13 53 1 0 0 0 0
54 55 2 0 0 0 0
54 56 1 0 0 0 0
56 57 2 0 0 0 0
57 58 1 0 0 0 0
58 59 2 0 0 0 0
59 60 1 0 0 0 0
60 61 2 0 0 0 0
61 62 1 0 0 0 0
62 63 2 0 0 0 0
63 58 1 0 0 0 0
61 64 1 0 0 0 0
52 65 1 0 0 0 0
65 54 1 0 0 0 0
M CHG 1 10 1
S SKP 5
ID FL7AAGGA0008
FORMULA C42H47O23
EXACTMASS 919.25081281
AVERAGEMASS 919.8087800000001
SMILES O(C(Oc(c3)c(c5)c([o+1]c(c5OC(O6)C(C(C(O)C6COC(O7)C(C(C(O)C7C)O)O)O)O)c(c4)cc(c(c4O)O)O)cc3O)1)C(COC(=O)C=Cc(c2)ccc(O)c2)C(O)C(O)C1O
M END