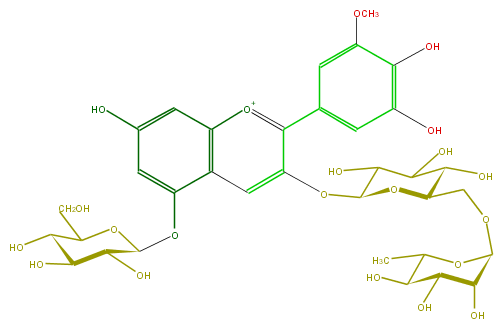
Mol:FL7AAHGL0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.2860 0.1012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2860 0.1012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2859 -0.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2859 -0.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4842 -1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4842 -1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6821 -0.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6821 -0.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6822 0.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6822 0.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4841 0.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4841 0.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1198 -1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1198 -1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9217 -0.8247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9217 -0.8247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9217 0.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9217 0.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1198 0.5642 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.1198 0.5642 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7233 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7233 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5405 0.0921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5405 0.0921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3579 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3579 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3580 1.5078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3580 1.5078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5405 1.9797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5405 1.9797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7233 1.5077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7233 1.5077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0877 0.5639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0877 0.5639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1296 1.9533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1296 1.9533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4840 -2.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4840 -2.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8061 -1.3180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8061 -1.3180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0347 -2.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0347 -2.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6481 -3.3248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6481 -3.3248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3916 -3.1122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3916 -3.1122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1396 -3.3248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1396 -3.3248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5264 -2.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5264 -2.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7827 -2.8675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7827 -2.8675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1932 -2.7734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1932 -2.7734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0083 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0083 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6216 -1.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6216 -1.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3653 -1.1988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3653 -1.1988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1133 -1.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1133 -1.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5001 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5001 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7564 -0.9541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7564 -0.9541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1337 -0.8154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1337 -0.8154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4300 -0.3661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4300 -0.3661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3058 -0.9080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3058 -0.9080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8855 -1.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8855 -1.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3963 -1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3963 -1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9737 -3.1393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9737 -3.1393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9599 -3.8213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9599 -3.8213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1396 -3.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1396 -3.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1750 0.0923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1750 0.0923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2260 -2.2174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2260 -2.2174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7268 -2.8763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7268 -2.8763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0082 -2.5967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0082 -2.5967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2590 -2.6050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2590 -2.6050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8186 -2.0853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8186 -2.0853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5528 -2.3489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5528 -2.3489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9078 -2.4926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9078 -2.4926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4648 -2.8586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4648 -2.8586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2650 -3.1038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2650 -3.1038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1053 -1.6321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1053 -1.6321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5264 -1.3904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5264 -1.3904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5405 2.6839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5405 2.6839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1770 3.9772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1770 3.9772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 24 41 1 0 0 0 0 | + | 24 41 1 0 0 0 0 |
| − | 29 20 1 0 0 0 0 | + | 29 20 1 0 0 0 0 |
| − | 13 42 1 0 0 0 0 | + | 13 42 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 19 1 0 0 0 0 | + | 46 19 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 48 52 1 0 0 0 0 | + | 48 52 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 15 54 1 0 0 0 0 | + | 15 54 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 52 53 | + | M SAL 1 2 52 53 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 58 0.5524 -0.7167 | + | M SBV 1 58 0.5524 -0.7167 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 54 55 | + | M SAL 2 2 54 55 |
| − | M SBL 2 1 60 | + | M SBL 2 1 60 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 60 0.0000 -0.7041 | + | M SBV 2 60 0.0000 -0.7041 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AAHGL0008 | + | ID FL7AAHGL0008 |
| − | FORMULA C34H43O21 | + | FORMULA C34H43O21 |
| − | EXACTMASS 787.2296834379999 | + | EXACTMASS 787.2296834379999 |
| − | AVERAGEMASS 787.6926199999999 | + | AVERAGEMASS 787.6926199999999 |
| − | SMILES OC(C(O)1)C(C(CO)OC(Oc(c62)cc(cc2[o+1]c(c(c6)OC(C(O)5)OC(C(O)C(O)5)COC(O4)C(C(C(C4C)O)O)O)c(c3)cc(c(O)c3OC)O)O)1)O | + | SMILES OC(C(O)1)C(C(CO)OC(Oc(c62)cc(cc2[o+1]c(c(c6)OC(C(O)5)OC(C(O)C(O)5)COC(O4)C(C(C(C4C)O)O)O)c(c3)cc(c(O)c3OC)O)O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
-2.2860 0.1012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2859 -0.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4842 -1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6821 -0.8248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6822 0.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4841 0.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1198 -1.2879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9217 -0.8247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9217 0.1013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1198 0.5642 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.7233 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5405 0.0921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3579 0.5640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3580 1.5078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5405 1.9797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7233 1.5077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0877 0.5639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1296 1.9533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4840 -2.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8061 -1.3180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0347 -2.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6481 -3.3248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3916 -3.1122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1396 -3.3248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5264 -2.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7827 -2.8675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1932 -2.7734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0083 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6216 -1.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3653 -1.1988 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1133 -1.4113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5001 -0.7417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7564 -0.9541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1337 -0.8154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4300 -0.3661 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3058 -0.9080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8855 -1.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3963 -1.8654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9737 -3.1393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9599 -3.8213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1396 -3.9772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1750 0.0923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2260 -2.2174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7268 -2.8763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0082 -2.5967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2590 -2.6050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8186 -2.0853 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5528 -2.3489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9078 -2.4926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4648 -2.8586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2650 -3.1038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1053 -1.6321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5264 -1.3904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5405 2.6839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1770 3.9772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
25 38 1 0 0 0 0
37 38 1 0 0 0 0
22 39 1 0 0 0 0
23 40 1 0 0 0 0
24 41 1 0 0 0 0
29 20 1 0 0 0 0
13 42 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
46 19 1 0 0 0 0
52 53 1 0 0 0 0
48 52 1 0 0 0 0
54 55 1 0 0 0 0
15 54 1 0 0 0 0
M CHG 1 10 1
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 52 53
M SBL 1 1 58
M SMT 1 ^ CH2OH
M SBV 1 58 0.5524 -0.7167
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 54 55
M SBL 2 1 60
M SMT 2 OCH3
M SBV 2 60 0.0000 -0.7041
S SKP 5
ID FL7AAHGL0008
FORMULA C34H43O21
EXACTMASS 787.2296834379999
AVERAGEMASS 787.6926199999999
SMILES OC(C(O)1)C(C(CO)OC(Oc(c62)cc(cc2[o+1]c(c(c6)OC(C(O)5)OC(C(O)C(O)5)COC(O4)C(C(C(C4C)O)O)O)c(c3)cc(c(O)c3OC)O)O)1)O
M END