Mol:FL7AAIGL0019
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 57 62 0 0 0 0 0 0 0 0999 V2000 | + | 57 62 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.8271 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8271 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8271 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8271 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2708 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2708 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7145 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7145 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7145 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7145 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2708 1.4708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2708 1.4708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1582 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1582 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6019 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6019 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6019 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6019 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1582 1.4708 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -2.1582 1.4708 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0458 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0458 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4788 1.1434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4788 1.1434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0882 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0882 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0882 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0882 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4788 2.4528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4788 2.4528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0458 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0458 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3832 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3832 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9542 2.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9542 2.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2708 -0.4560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2708 -0.4560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1858 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1858 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3171 -0.2372 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.3171 -0.2372 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.0488 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.0488 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5647 -0.5543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5647 -0.5543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0836 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.0836 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.3519 -0.2372 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.3519 -0.2372 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.8360 -0.3845 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.8360 -0.3845 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.0474 0.1275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0474 0.1275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9777 0.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9777 0.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6136 0.0747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6136 0.0747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4859 -1.1041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4859 -1.1041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2648 -1.9295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2648 -1.9295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6552 -2.2815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6552 -2.2815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0107 -2.2814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0107 -2.2814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9427 -2.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9427 -2.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5437 -3.3651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5437 -3.3651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8967 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8967 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5919 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5919 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8967 -5.0324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8967 -5.0324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5063 -5.0325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5063 -5.0325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8112 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8112 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5064 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5064 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8109 -5.5600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8109 -5.5600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1408 -0.8539 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -5.1408 -0.8539 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -4.7696 -1.3439 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.7696 -1.3439 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -4.2351 -1.1360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.2351 -1.1360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.7194 -1.1305 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.7194 -1.1305 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -4.0941 -0.7556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0941 -0.7556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5618 -1.0024 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -4.5618 -1.0024 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -5.6545 -0.5573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6545 -0.5573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1199 -1.8051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1199 -1.8051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9289 -1.6502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9289 -1.6502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9048 -0.4321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9048 -0.4321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6925 0.2675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6925 0.2675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1954 3.0289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1954 3.0289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2460 3.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2460 3.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0882 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0882 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0882 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0882 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 19 1 0 0 0 0 | + | 46 19 1 0 0 0 0 |
| − | 48 52 1 0 0 0 0 | + | 48 52 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 15 54 1 0 0 0 0 | + | 15 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 13 56 1 0 0 0 0 | + | 13 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 52 53 | + | M SAL 3 2 52 53 |
| − | M SBL 3 1 57 | + | M SBL 3 1 57 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 57 -4.9986 -0.5003 | + | M SVB 3 57 -4.9986 -0.5003 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 56 57 | + | M SAL 2 2 56 57 |
| − | M SBL 2 1 61 | + | M SBL 2 1 61 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 61 0.2977 1.2645 | + | M SVB 2 61 0.2977 1.2645 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 54 55 | + | M SAL 1 2 54 55 |
| − | M SBL 1 1 59 | + | M SBL 1 1 59 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 59 -0.1954 3.0289 | + | M SVB 1 59 -0.1954 3.0289 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAIGL0019 | + | ID FL7AAIGL0019 |
| − | KNApSAcK_ID C00006910 | + | KNApSAcK_ID C00006910 |
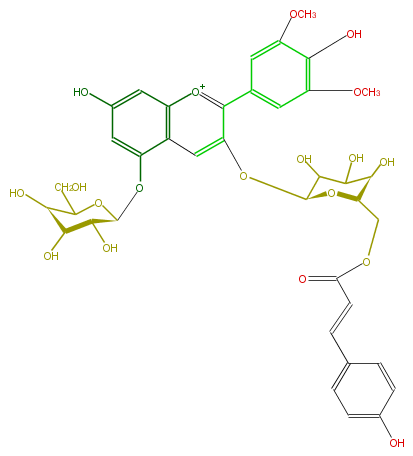
| − | NAME Tibouchinin;Malvidin-3-(6-O-p-coumarylglucoside)-5-glucoside | + | NAME Tibouchinin;Malvidin-3-(6-O-p-coumarylglucoside)-5-glucoside |
| − | CAS_RN 144940-56-7,51939-51-6 | + | CAS_RN 144940-56-7,51939-51-6 |
| − | FORMULA C38H41O19 | + | FORMULA C38H41O19 |
| − | EXACTMASS 801.22420413 | + | EXACTMASS 801.22420413 |
| − | AVERAGEMASS 801.72074 | + | AVERAGEMASS 801.72074 |
| − | SMILES O([C@H](O6)[C@@H](O)[C@H]([C@@H](O)C6CO)O)c(c5)c(c2)c(cc5O)[o+1]c(c2O[C@H](O3)C(O)C([C@@H](O)[C@H]3COC(C=Cc(c4)ccc(O)c4)=O)O)c(c1)cc(OC)c(c(OC)1)O | + | SMILES O([C@H](O6)[C@@H](O)[C@H]([C@@H](O)C6CO)O)c(c5)c(c2)c(cc5O)[o+1]c(c2O[C@H](O3)C(O)C([C@@H](O)[C@H]3COC(C=Cc(c4)ccc(O)c4)=O)O)c(c1)cc(OC)c(c(OC)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
57 62 0 0 0 0 0 0 0 0999 V2000
-3.8271 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8271 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2708 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7145 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7145 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2708 1.4708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1582 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6019 0.5073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6019 1.1497 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1582 1.4708 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-1.0458 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4788 1.1434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0882 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0882 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4788 2.4528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0458 2.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3832 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9542 2.6254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2708 -0.4560 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1858 -0.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3171 -0.2372 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.0488 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5647 -0.5543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0836 -0.7017 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.3519 -0.2372 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.8360 -0.3845 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.0474 0.1275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9777 0.1442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6136 0.0747 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4859 -1.1041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2648 -1.9295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6552 -2.2815 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0107 -2.2814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9427 -2.7775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5437 -3.3651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8967 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5919 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8967 -5.0324 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5063 -5.0325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8112 -4.5045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5064 -3.9766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8109 -5.5600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1408 -0.8539 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-4.7696 -1.3439 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-4.2351 -1.1360 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.7194 -1.1305 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-4.0941 -0.7556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5618 -1.0024 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-5.6545 -0.5573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1199 -1.8051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9289 -1.6502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9048 -0.4321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6925 0.2675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1954 3.0289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2460 3.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0882 1.4707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0882 1.4707 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
24 30 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
46 19 1 0 0 0 0
48 52 1 0 0 0 0
52 53 1 0 0 0 0
15 54 1 0 0 0 0
54 55 1 0 0 0 0
13 56 1 0 0 0 0
56 57 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 52 53
M SBL 3 1 57
M SMT 3 CH2OH
M SVB 3 57 -4.9986 -0.5003
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 56 57
M SBL 2 1 61
M SMT 2 OCH3
M SVB 2 61 0.2977 1.2645
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 54 55
M SBL 1 1 59
M SMT 1 OCH3
M SVB 1 59 -0.1954 3.0289
S SKP 8
ID FL7AAIGL0019
KNApSAcK_ID C00006910
NAME Tibouchinin;Malvidin-3-(6-O-p-coumarylglucoside)-5-glucoside
CAS_RN 144940-56-7,51939-51-6
FORMULA C38H41O19
EXACTMASS 801.22420413
AVERAGEMASS 801.72074
SMILES O([C@H](O6)[C@@H](O)[C@H]([C@@H](O)C6CO)O)c(c5)c(c2)c(cc5O)[o+1]c(c2O[C@H](O3)C(O)C([C@@H](O)[C@H]3COC(C=Cc(c4)ccc(O)c4)=O)O)c(c1)cc(OC)c(c(OC)1)O
M END