Mol:BMCCPUAP0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 35 37 0 0 1 0 0 0 0 0999 V2000 | + | 35 37 0 0 1 0 0 0 0 0999 V2000 |
| − | 3.7321 3.0970 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 3.0970 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5981 1.5970 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5981 1.5970 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7321 1.0970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7321 1.0970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 1.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 1.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8660 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8660 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1229 0.9279 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1229 0.9279 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5296 0.0143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5296 0.0143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5241 0.1188 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5241 0.1188 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 3.0970 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 3.0970 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1933 -0.6243 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.1933 -0.6243 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.9854 -1.6025 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.9854 -1.6025 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.8514 -2.1025 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.8514 -2.1025 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 5.5945 -1.4333 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 5.5945 -1.4333 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 6.5727 -1.6412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5727 -1.6412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0718 -2.0092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0718 -2.0092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9559 -3.0970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9559 -3.0970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1878 -0.5198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1878 -0.5198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2418 -0.8981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2418 -0.8981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2200 -1.1060 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2200 -1.1060 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.0120 -2.0842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.0120 -2.0842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.4279 -0.1279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.4279 -0.1279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1981 -1.3139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1981 -1.3139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8672 -0.5708 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8672 -0.5708 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.6104 -1.2399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.6104 -1.2399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.1241 0.0984 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.1241 0.0984 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5364 0.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5364 0.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.5145 -0.0355 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.5145 -0.0355 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.3066 -1.0137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.3066 -1.0137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.7224 0.9426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.7224 0.9426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.4927 -0.2435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.4927 -0.2435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.1618 0.4997 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.1618 0.4997 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.9049 -0.1694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.9049 -0.1694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.4186 1.1688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.4186 1.1688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.8309 1.2428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.8309 1.2428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 3 2 2 0 0 0 0 | + | 3 2 2 0 0 0 0 |
| − | 2 1 1 0 0 0 0 | + | 2 1 1 0 0 0 0 |
| − | 1 6 2 0 0 0 0 | + | 1 6 2 0 0 0 0 |
| − | 6 5 1 0 0 0 0 | + | 6 5 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 4 1 0 0 0 0 | + | 9 4 1 0 0 0 0 |
| − | 6 10 1 0 0 0 0 | + | 6 10 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 18 1 6 0 0 0 | + | 14 18 1 6 0 0 0 |
| − | 11 18 1 6 0 0 0 | + | 11 18 1 6 0 0 0 |
| − | 12 16 1 1 0 0 0 | + | 12 16 1 1 0 0 0 |
| − | 13 17 1 1 0 0 0 | + | 13 17 1 1 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 11 9 1 0 0 0 0 | + | 11 9 1 0 0 0 0 |
| − | 31 28 1 0 0 0 0 | + | 31 28 1 0 0 0 0 |
| − | 28 27 1 0 0 0 0 | + | 28 27 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 28 30 2 0 0 0 0 | + | 28 30 2 0 0 0 0 |
| − | 27 24 1 0 0 0 0 | + | 27 24 1 0 0 0 0 |
| − | 24 23 1 0 0 0 0 | + | 24 23 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 24 26 2 0 0 0 0 | + | 24 26 2 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 20 19 1 0 0 0 0 | + | 20 19 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 20 22 2 0 0 0 0 | + | 20 22 2 0 0 0 0 |
| − | 33 32 1 0 0 0 0 | + | 33 32 1 0 0 0 0 |
| − | 32 31 1 0 0 0 0 | + | 32 31 1 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 32 35 2 0 0 0 0 | + | 32 35 2 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCPUAP0016 | + | ID BMCCPUAP0016 |
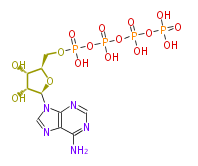
| − | NAME Adenosine 5'-tetraphosphate | + | NAME Adenosine 5'-tetraphosphate |
| − | FORMULA C10H17N5O16P4 | + | FORMULA C10H17N5O16P4 |
| − | EXACTMASS 586.962 | + | EXACTMASS 586.962 |
| − | AVERAGEMASS 587.1611 | + | AVERAGEMASS 587.1611 |
| − | SMILES Nc(n3)c(n2)c(nc3)n(c2)[C@H](O1)[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(=O)OP(O)(O)=O)1 | + | SMILES Nc(n3)c(n2)c(nc3)n(c2)[C@H](O1)[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(=O)OP(O)(O)=O)1 |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C03483 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C03483 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
35 37 0 0 1 0 0 0 0 0999 V2000
3.7321 3.0970 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5981 1.5970 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.7321 1.0970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 1.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8660 2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1229 0.9279 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.5296 0.0143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5241 0.1188 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 3.0970 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
4.1933 -0.6243 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.9854 -1.6025 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.8514 -2.1025 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
5.5945 -1.4333 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
6.5727 -1.6412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0718 -2.0092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9559 -3.0970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1878 -0.5198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.2418 -0.8981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.2200 -1.1060 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
8.0120 -2.0842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.4279 -0.1279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.1981 -1.3139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.8672 -0.5708 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
10.6104 -1.2399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
9.1241 0.0984 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.5364 0.1724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.5145 -0.0355 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
11.3066 -1.0137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.7224 0.9426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.4927 -0.2435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.1618 0.4997 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
13.9049 -0.1694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.4186 1.1688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.8309 1.2428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4 3 1 0 0 0 0
3 2 2 0 0 0 0
2 1 1 0 0 0 0
1 6 2 0 0 0 0
6 5 1 0 0 0 0
4 5 2 0 0 0 0
5 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 4 1 0 0 0 0
6 10 1 0 0 0 0
11 12 1 0 0 0 0
12 13 1 0 0 0 0
13 14 1 0 0 0 0
14 18 1 6 0 0 0
11 18 1 6 0 0 0
12 16 1 1 0 0 0
13 17 1 1 0 0 0
14 15 1 0 0 0 0
15 19 1 0 0 0 0
11 9 1 0 0 0 0
31 28 1 0 0 0 0
28 27 1 0 0 0 0
28 29 1 0 0 0 0
28 30 2 0 0 0 0
27 24 1 0 0 0 0
24 23 1 0 0 0 0
24 25 1 0 0 0 0
24 26 2 0 0 0 0
23 20 1 0 0 0 0
20 19 1 0 0 0 0
20 21 1 0 0 0 0
20 22 2 0 0 0 0
33 32 1 0 0 0 0
32 31 1 0 0 0 0
32 34 1 0 0 0 0
32 35 2 0 0 0 0
S SKP 7
ID BMCCPUAP0016
NAME Adenosine 5'-tetraphosphate
FORMULA C10H17N5O16P4
EXACTMASS 586.962
AVERAGEMASS 587.1611
SMILES Nc(n3)c(n2)c(nc3)n(c2)[C@H](O1)[C@H](O)[C@H](O)[C@@H](COP(O)(=O)OP(O)(=O)OP(O)(=O)OP(O)(O)=O)1
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C03483
M END