Mol:BMMCBZ4Sa013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 63 66 0 0 1 0 0 0 0 0999 V2000 | + | 63 66 0 0 1 0 0 0 0 0999 V2000 |
| − | 4.9344 -3.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9344 -3.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6254 -3.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6254 -3.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6473 -4.2064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6473 -4.2064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9781 -3.4632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9781 -3.4632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2872 -2.5122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2872 -2.5122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2653 -2.3043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2653 -2.3043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9126 -3.2553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9126 -3.2553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5817 -2.5122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5817 -2.5122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.5599 -2.7201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.5599 -2.7201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8689 -3.6712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8689 -3.6712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3383 -5.1574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3383 -5.1574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0074 -5.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0074 -5.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -3.6712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -3.6712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6180 -1.7690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6180 -1.7690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9271 -0.8180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9271 -0.8180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.9170 -1.6902 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.9170 -1.6902 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.0510 -1.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.0510 -1.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.0510 -0.1902 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.0510 -0.1902 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.9170 0.3098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.9170 0.3098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.7830 -0.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.7830 -0.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.7830 -1.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.7830 -1.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.5262 0.4790 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.5262 0.4790 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.1194 1.3925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.1194 1.3925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.1249 1.2880 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.1249 1.2880 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 26.6490 -1.6902 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 26.6490 -1.6902 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.4558 2.0311 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 24.4558 2.0311 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 24.6637 3.0093 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 24.6637 3.0093 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 23.7976 3.5093 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 23.7976 3.5093 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 23.0545 2.8401 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 23.0545 2.8401 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 22.0764 3.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.0764 3.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.5772 3.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.5772 3.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.6931 4.5038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.6931 4.5038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.4612 1.9266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.4612 1.9266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.4072 2.3049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.4072 2.3049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.5021 5.0916 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.5021 5.0916 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.0899 4.2826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.0899 4.2826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.9144 5.9006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.9144 5.9006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 25.3112 5.6794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 25.3112 5.6794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.4291 2.5128 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.4291 2.5128 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.2212 1.5347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.2212 1.5347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 20.6370 3.4910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 20.6370 3.4910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.4509 2.7207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.4509 2.7207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.7818 1.9776 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.7818 1.9776 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.5249 1.3085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.5249 1.3085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.0387 2.6467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.0387 2.6467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.1127 1.2344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.1127 1.2344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.1345 1.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.1345 1.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.4654 0.6992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.4654 0.6992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.2085 0.0301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.2085 0.0301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.7222 1.3683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.7222 1.3683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.7963 -0.0439 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 15.7963 -0.0439 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 14.8181 0.1640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.8181 0.1640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.1490 -0.5792 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.1490 -0.5792 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.1708 -0.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.1708 -0.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.5017 -1.1144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5017 -1.1144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.5236 -0.9065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.5236 -0.9065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.8544 -1.6496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.8544 -1.6496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.8763 -1.4417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.8763 -1.4417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2071 -2.1849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2071 -2.1849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.2290 -1.9770 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.2290 -1.9770 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.1053 -0.9950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.1053 -0.9950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.5091 1.1150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.5091 1.1150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.2145 0.0446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.2145 0.0446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16 21 2 0 0 0 0 | + | 16 21 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 21 20 1 0 0 0 0 | + | 21 20 1 0 0 0 0 |
| − | 20 19 2 0 0 0 0 | + | 20 19 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 23 22 2 0 0 0 0 | + | 23 22 2 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 21 25 1 0 0 0 0 | + | 21 25 1 0 0 0 0 |
| − | 29 33 1 6 0 0 0 | + | 29 33 1 6 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 26 33 1 6 0 0 0 | + | 26 33 1 6 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 34 30 1 0 0 0 0 | + | 34 30 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 46 43 1 0 0 0 0 | + | 46 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 48 50 1 0 0 0 0 | + | 48 50 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 51 61 1 1 0 0 0 | + | 51 61 1 1 0 0 0 |
| − | 52 62 2 0 0 0 0 | + | 52 62 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 56 63 2 0 0 0 0 | + | 56 63 2 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 27 31 1 1 0 0 0 | + | 27 31 1 1 0 0 0 |
| − | 28 32 1 1 0 0 0 | + | 28 32 1 1 0 0 0 |
| − | 36 35 1 0 0 0 0 | + | 36 35 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 35 37 2 0 0 0 0 | + | 35 37 2 0 0 0 0 |
| − | 32 35 1 0 0 0 0 | + | 32 35 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 6 5 1 0 0 0 0 | + | 6 5 1 0 0 0 0 |
| − | 5 4 2 0 0 0 0 | + | 5 4 2 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 3 2 2 0 0 0 0 | + | 3 2 2 0 0 0 0 |
| − | 2 1 1 0 0 0 0 | + | 2 1 1 0 0 0 0 |
| − | 1 7 1 0 0 0 0 | + | 1 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 9 60 1 0 0 0 0 | + | 9 60 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 5 14 1 0 0 0 0 | + | 5 14 1 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 3 11 1 0 0 0 0 | + | 3 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 4 13 1 0 0 0 0 | + | 4 13 1 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMMCBZ4Sa013 | + | ID BMMCBZ4Sa013 |
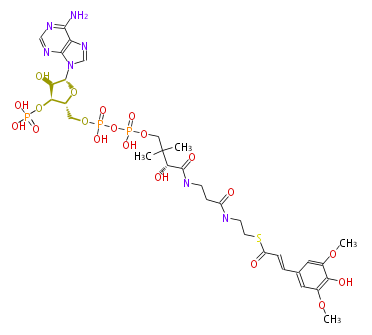
| − | NAME Sinapoyl-CoA | + | NAME Sinapoyl-CoA |
| − | FORMULA C32H46N7O20P3S | + | FORMULA C32H46N7O20P3S |
| − | EXACTMASS 973.1731 | + | EXACTMASS 973.1731 |
| − | AVERAGEMASS 973.7301 | + | AVERAGEMASS 973.7301 |
| − | SMILES C(CNC(CCNC([C@@H](C(C)(C)COP(OP(O)(=O)OC[C@@H](O2)[C@@H](OP(O)(O)=O)[C@H]([C@@H]2n(c34)cnc3c(ncn4)N)O)(O)=O)O)=O)=O)SC(C=Cc(c1)cc(c(O)c1OC)OC)=O | + | SMILES C(CNC(CCNC([C@@H](C(C)(C)COP(OP(O)(=O)OC[C@@H](O2)[C@@H](OP(O)(O)=O)[C@H]([C@@H]2n(c34)cnc3c(ncn4)N)O)(O)=O)O)=O)=O)SC(C=Cc(c1)cc(c(O)c1OC)OC)=O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00411 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00411 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
63 66 0 0 1 0 0 0 0 0999 V2000
4.9344 -3.0474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6254 -3.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6473 -4.2064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9781 -3.4632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2872 -2.5122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2653 -2.3043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9126 -3.2553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.5817 -2.5122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5599 -2.7201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.8689 -3.6712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3383 -5.1574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0074 -5.9006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -3.6712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6180 -1.7690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9271 -0.8180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
24.9170 -1.6902 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
24.0510 -1.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
24.0510 -0.1902 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
24.9170 0.3098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.7830 -0.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.7830 -1.1902 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
26.5262 0.4790 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
26.1194 1.3925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.1249 1.2880 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
26.6490 -1.6902 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
24.4558 2.0311 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
24.6637 3.0093 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
23.7976 3.5093 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
23.0545 2.8401 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
22.0764 3.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
25.5772 3.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.6931 4.5038 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.4612 1.9266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.4072 2.3049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
24.5021 5.0916 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
25.0899 4.2826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.9144 5.9006 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
25.3112 5.6794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.4291 2.5128 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
20.2212 1.5347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
20.6370 3.4910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.4509 2.7207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.7818 1.9776 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
19.5249 1.3085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.0387 2.6467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.1127 1.2344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.1345 1.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.4654 0.6992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
17.2085 0.0301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.7222 1.3683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.7963 -0.0439 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
14.8181 0.1640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.1490 -0.5792 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
13.1708 -0.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.5017 -1.1144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.5236 -0.9065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.8544 -1.6496 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
9.8763 -1.4417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.2071 -2.1849 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.2290 -1.9770 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
16.1053 -0.9950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.5091 1.1150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.2145 0.0446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16 21 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
21 20 1 0 0 0 0
20 19 2 0 0 0 0
23 24 1 0 0 0 0
23 22 2 0 0 0 0
20 22 1 0 0 0 0
19 24 1 0 0 0 0
21 25 1 0 0 0 0
29 33 1 6 0 0 0
28 29 1 0 0 0 0
26 33 1 6 0 0 0
27 28 1 0 0 0 0
26 27 1 0 0 0 0
29 30 1 0 0 0 0
34 30 1 0 0 0 0
24 26 1 0 0 0 0
34 39 1 0 0 0 0
39 41 1 0 0 0 0
39 40 2 0 0 0 0
39 42 1 0 0 0 0
46 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
46 47 1 0 0 0 0
42 43 1 0 0 0 0
47 48 1 0 0 0 0
48 51 1 0 0 0 0
48 49 1 0 0 0 0
48 50 1 0 0 0 0
51 52 1 0 0 0 0
51 61 1 1 0 0 0
52 62 2 0 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
55 56 1 0 0 0 0
56 57 1 0 0 0 0
56 63 2 0 0 0 0
57 58 1 0 0 0 0
58 59 1 0 0 0 0
59 60 1 0 0 0 0
27 31 1 1 0 0 0
28 32 1 1 0 0 0
36 35 1 0 0 0 0
35 38 1 0 0 0 0
35 37 2 0 0 0 0
32 35 1 0 0 0 0
6 1 2 0 0 0 0
6 5 1 0 0 0 0
5 4 2 0 0 0 0
4 3 1 0 0 0 0
3 2 2 0 0 0 0
2 1 1 0 0 0 0
1 7 1 0 0 0 0
7 8 2 0 0 0 0
9 10 2 0 0 0 0
9 60 1 0 0 0 0
8 9 1 0 0 0 0
5 14 1 0 0 0 0
14 15 1 0 0 0 0
3 11 1 0 0 0 0
11 12 1 0 0 0 0
4 13 1 0 0 0 0
S SKP 7
ID BMMCBZ4Sa013
NAME Sinapoyl-CoA
FORMULA C32H46N7O20P3S
EXACTMASS 973.1731
AVERAGEMASS 973.7301
SMILES C(CNC(CCNC([C@@H](C(C)(C)COP(OP(O)(=O)OC[C@@H](O2)[C@@H](OP(O)(O)=O)[C@H]([C@@H]2n(c34)cnc3c(ncn4)N)O)(O)=O)O)=O)=O)SC(C=Cc(c1)cc(c(O)c1OC)OC)=O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C00411
M END