Mol:FL1C3CGS0017
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 45 0 0 0 0 0 0 0 0999 V2000 | + | 42 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.0832 -1.0619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0832 -1.0619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0832 -1.8927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0832 -1.8927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3637 -2.3082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3637 -2.3082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3559 -1.8927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3559 -1.8927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3559 -1.0619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3559 -1.0619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3637 -0.6465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3637 -0.6465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0750 -2.3080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0750 -2.3080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7926 -1.8937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7926 -1.8937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5088 -2.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5088 -2.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2232 -1.8946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2232 -1.8946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9225 -2.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9225 -2.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6218 -1.8946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6218 -1.8946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6218 -1.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6218 -1.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9225 -0.6834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9225 -0.6834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2232 -1.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2232 -1.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0750 -3.1366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0750 -3.1366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3637 -3.1387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3637 -3.1387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8023 -0.6467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8023 -0.6467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7130 -2.3740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7130 -2.3740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3208 -2.2982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3208 -2.2982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2811 0.8225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2811 0.8225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5434 0.3967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5434 0.3967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7774 1.2155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7774 1.2155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5434 2.0395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5434 2.0395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2811 2.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2811 2.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0470 1.6465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0470 1.6465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8924 3.1387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8924 3.1387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5434 2.7297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5434 2.7297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6338 2.1291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6338 2.1291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3317 0.7698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3317 0.7698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5192 1.6019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5192 1.6019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0694 1.0084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0694 1.0084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4218 1.2602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4218 1.2602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7469 1.2528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7469 1.2528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2510 1.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2510 1.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9125 1.4836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9125 1.4836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2488 1.0848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2488 1.0848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7705 0.9742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7705 0.9742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8286 0.9177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8286 0.9177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2516 1.7480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2516 1.7480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3208 -0.6836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3208 -0.6836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2488 -1.2193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2488 -1.2193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 19 2 1 0 0 0 0 | + | 19 2 1 0 0 0 0 |
| − | 12 20 1 0 0 0 0 | + | 12 20 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 21 1 1 0 0 0 | + | 26 21 1 1 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 22 18 1 0 0 0 0 | + | 22 18 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 40 29 1 0 0 0 0 | + | 40 29 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 13 41 1 0 0 0 0 | + | 13 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 45 -0.6990 -0.4035 | + | M SBV 1 45 -0.6990 -0.4035 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL1C3CGS0017 | + | ID FL1C3CGS0017 |
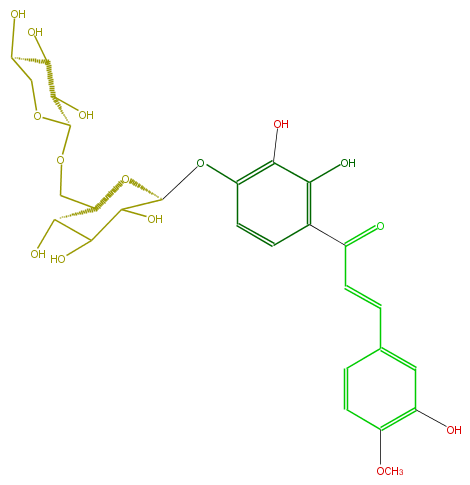
| − | FORMULA C27H32O15 | + | FORMULA C27H32O15 |
| − | EXACTMASS 596.174120354 | + | EXACTMASS 596.174120354 |
| − | AVERAGEMASS 596.5339799999999 | + | AVERAGEMASS 596.5339799999999 |
| − | SMILES O(c(c4)c(cc(c4)C=CC(c(c3)c(O)c(O)c(c3)OC(O1)C(O)C(O)C(O)C(COC(O2)C(O)C(C(O)C2)O)1)=O)O)C | + | SMILES O(c(c4)c(cc(c4)C=CC(c(c3)c(O)c(O)c(c3)OC(O1)C(O)C(O)C(O)C(COC(O2)C(O)C(C(O)C2)O)1)=O)O)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 45 0 0 0 0 0 0 0 0999 V2000
-1.0832 -1.0619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0832 -1.8927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3637 -2.3082 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3559 -1.8927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3559 -1.0619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3637 -0.6465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0750 -2.3080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7926 -1.8937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5088 -2.3071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2232 -1.8946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9225 -2.2982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6218 -1.8946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6218 -1.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9225 -0.6834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2232 -1.0871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0750 -3.1366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3637 -3.1387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8023 -0.6467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7130 -2.3740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3208 -2.2982 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2811 0.8225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5434 0.3967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7774 1.2155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5434 2.0395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2811 2.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0470 1.6465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8924 3.1387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5434 2.7297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6338 2.1291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3317 0.7698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5192 1.6019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0694 1.0084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4218 1.2602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7469 1.2528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2510 1.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9125 1.4836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.2488 1.0848 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7705 0.9742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8286 0.9177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2516 1.7480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3208 -0.6836 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.2488 -1.2193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
3 17 1 0 0 0 0
1 18 1 0 0 0 0
19 2 1 0 0 0 0
12 20 1 0 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 21 1 1 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
26 29 1 0 0 0 0
23 30 1 0 0 0 0
22 18 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
40 29 1 0 0 0 0
41 42 1 0 0 0 0
13 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 OCH3
M SBV 1 45 -0.6990 -0.4035
S SKP 5
ID FL1C3CGS0017
FORMULA C27H32O15
EXACTMASS 596.174120354
AVERAGEMASS 596.5339799999999
SMILES O(c(c4)c(cc(c4)C=CC(c(c3)c(O)c(O)c(c3)OC(O1)C(O)C(O)C(O)C(COC(O2)C(O)C(C(O)C2)O)1)=O)O)C
M END