Mol:FL2FACGS0019
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.8266 -0.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8266 -0.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5393 -0.0856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5393 -0.0856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8298 -1.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8298 -1.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5466 -1.7356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5466 -1.7356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2592 -1.3199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2592 -1.3199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2556 -0.4949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2556 -0.4949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9755 -1.7293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9755 -1.7293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6882 -1.3136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6882 -1.3136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6845 -0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6845 -0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9682 -0.0793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9682 -0.0793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3383 -0.1078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3383 -0.1078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0543 -0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0543 -0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7672 -0.1023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7672 -0.1023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7640 0.7227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7640 0.7227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0480 1.1325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0480 1.1325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3351 0.7172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3351 0.7172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9766 -2.3725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9766 -2.3725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2012 -0.1402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2012 -0.1402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5466 -2.4602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5466 -2.4602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4199 1.1014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4199 1.1014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3978 -0.4663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3978 -0.4663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5693 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5693 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0038 -0.9830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0038 -0.9830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2747 -0.5964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2747 -0.5964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4504 -0.6353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4504 -0.6353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0158 -0.0342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0158 -0.0342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7450 -0.4207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7450 -0.4207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0136 0.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0136 0.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8861 -0.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8861 -0.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4715 -0.9687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4715 -0.9687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7554 -1.1484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7554 -1.1484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3528 0.3279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3528 0.3279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9461 1.0457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9461 1.0457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3644 1.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3644 1.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1894 1.7501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1894 1.7501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5961 1.0322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5961 1.0322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1778 0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1778 0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5839 -0.3957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5839 -0.3957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.4199 1.0255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.4199 1.0255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6072 2.4602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6072 2.4602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1223 1.0524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1223 1.0524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7162 1.7692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7162 1.7692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7045 0.3423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7045 0.3423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 1 3 1 0 0 0 0 | + | 1 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 2 1 0 0 0 0 | + | 6 2 1 0 0 0 0 |
| − | 5 7 1 0 0 0 0 | + | 5 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 6 1 0 0 0 0 | + | 10 6 1 0 0 0 0 |
| − | 9 11 1 6 0 0 0 | + | 9 11 1 6 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 4 19 1 0 0 0 0 | + | 4 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 43 28 1 0 0 0 0 | + | 43 28 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL2FACGS0019 | + | ID FL2FACGS0019 |
| − | KNApSAcK_ID C00014340 | + | KNApSAcK_ID C00014340 |
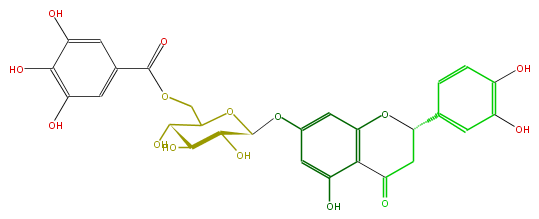
| − | NAME (2S)-5,7,3',4'-Tetrahydroxyflavanone 7-(6-galloylglucoside) | + | NAME (2S)-5,7,3',4'-Tetrahydroxyflavanone 7-(6-galloylglucoside) |
| − | CAS_RN 467437-60-1 | + | CAS_RN 467437-60-1 |
| − | FORMULA C28H26O15 | + | FORMULA C28H26O15 |
| − | EXACTMASS 602.127170162 | + | EXACTMASS 602.127170162 |
| − | AVERAGEMASS 602.49704 | + | AVERAGEMASS 602.49704 |
| − | SMILES C(O4)(C(O)C(C(O)C4COC(=O)c(c5)cc(O)c(c(O)5)O)O)Oc(c1)cc(O2)c(C(=O)CC(c(c3)cc(O)c(O)c3)2)c1O | + | SMILES C(O4)(C(O)C(C(O)C4COC(=O)c(c5)cc(O)c(c(O)5)O)O)Oc(c1)cc(O2)c(C(=O)CC(c(c3)cc(O)c(O)c3)2)c1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.8266 -0.5013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5393 -0.0856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8298 -1.3263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5466 -1.7356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2592 -1.3199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2556 -0.4949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9755 -1.7293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6882 -1.3136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6845 -0.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9682 -0.0793 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3383 -0.1078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0543 -0.5175 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7672 -0.1023 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7640 0.7227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0480 1.1325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3351 0.7172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9766 -2.3725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2012 -0.1402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5466 -2.4602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.4199 1.1014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.3978 -0.4663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5693 -0.3819 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0038 -0.9830 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2747 -0.5964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4504 -0.6353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0158 -0.0342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7450 -0.4207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0136 0.0791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8861 -0.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4715 -0.9687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7554 -1.1484 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3528 0.3279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9461 1.0457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3644 1.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1894 1.7501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5961 1.0322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1778 0.3212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5839 -0.3957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.4199 1.0255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6072 2.4602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1223 1.0524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7162 1.7692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7045 0.3423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
1 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 2 1 0 0 0 0
5 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 6 1 0 0 0 0
9 11 1 6 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
1 18 1 0 0 0 0
4 19 1 0 0 0 0
14 20 1 0 0 0 0
13 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 18 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
33 41 1 0 0 0 0
41 42 2 0 0 0 0
41 43 1 0 0 0 0
43 28 1 0 0 0 0
S SKP 8
ID FL2FACGS0019
KNApSAcK_ID C00014340
NAME (2S)-5,7,3',4'-Tetrahydroxyflavanone 7-(6-galloylglucoside)
CAS_RN 467437-60-1
FORMULA C28H26O15
EXACTMASS 602.127170162
AVERAGEMASS 602.49704
SMILES C(O4)(C(O)C(C(O)C4COC(=O)c(c5)cc(O)c(c(O)5)O)O)Oc(c1)cc(O2)c(C(=O)CC(c(c3)cc(O)c(O)c3)2)c1O
M END