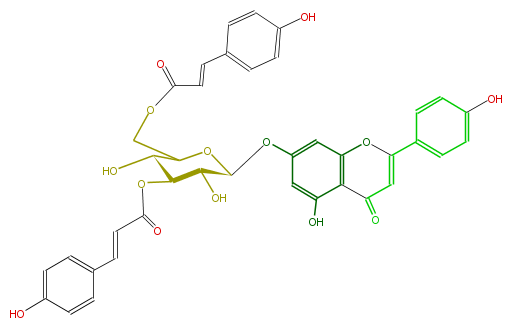
Mol:FL3FAAGS0046
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.7126 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7126 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7126 -0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7126 -0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2088 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2088 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7051 -0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7051 -0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7051 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7051 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2088 0.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2088 0.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2013 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2013 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6975 -0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6975 -0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6975 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6975 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2013 0.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2013 0.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3902 -0.8294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3902 -0.8294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1935 0.7213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1935 0.7213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6993 0.4293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6993 0.4293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2050 0.7213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2050 0.7213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2050 1.3052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2050 1.3052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6993 1.5972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6993 1.5972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1935 1.3052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1935 1.3052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2166 0.7213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2166 0.7213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7106 1.5971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7106 1.5971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1313 -0.8647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1313 -0.8647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0670 0.4378 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.0670 0.4378 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.6793 -0.0740 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.6793 -0.0740 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.1209 0.1431 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.1209 0.1431 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.5086 0.0386 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5086 0.0386 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.9737 0.5405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9737 0.5405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5440 0.3357 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.5440 0.3357 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.8594 0.1405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8594 0.1405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2837 -0.1034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2837 -0.1034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8010 -0.3939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8010 -0.3939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4619 0.7325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4619 0.7325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1048 1.3686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1048 1.3686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6427 1.8306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6427 1.8306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9098 2.2931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9098 2.2931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1102 1.8306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1102 1.8306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0723 2.2642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0723 2.2642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6019 2.4836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6019 2.4836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1261 2.1505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1261 2.1505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4003 2.3959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4003 2.3959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4510 2.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4510 2.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0249 3.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0249 3.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5512 3.0622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5512 3.0622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9768 3.2197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9768 3.2197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2654 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2654 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9613 -1.0609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9613 -1.0609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8188 -1.0763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8188 -1.0763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8188 -1.6183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8188 -1.6183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2574 -1.8716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2574 -1.8716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7419 -1.5919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7419 -1.5919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2263 -1.8716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2263 -1.8716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2263 -2.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2263 -2.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7419 -2.7106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7419 -2.7106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2574 -2.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2574 -2.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7106 -2.7106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7106 -2.7106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 20 3 1 0 0 0 0 | + | 20 3 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 18 24 1 0 0 0 0 | + | 18 24 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 43 28 1 0 0 0 0 | + | 43 28 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAAGS0046 | + | ID FL3FAAGS0046 |
| − | KNApSAcK_ID C00004186 | + | KNApSAcK_ID C00004186 |
| − | NAME Apigenin 7-(3'',6''-di-p-coumarylglucoside) | + | NAME Apigenin 7-(3'',6''-di-p-coumarylglucoside) |
| − | CAS_RN 83529-71-9 | + | CAS_RN 83529-71-9 |
| − | FORMULA C39H32O14 | + | FORMULA C39H32O14 |
| − | EXACTMASS 724.179205732 | + | EXACTMASS 724.179205732 |
| − | AVERAGEMASS 724.6629800000001 | + | AVERAGEMASS 724.6629800000001 |
| − | SMILES O=C(C=Cc(c6)ccc(O)c6)O[C@H]([C@H](O)2)[C@@H](O)C(O[C@H]2Oc(c3)cc(c(C5=O)c(OC(=C5)c(c4)ccc(O)c4)3)O)COC(C=Cc(c1)ccc(c1)O)=O | + | SMILES O=C(C=Cc(c6)ccc(O)c6)O[C@H]([C@H](O)2)[C@@H](O)C(O[C@H]2Oc(c3)cc(c(C5=O)c(OC(=C5)c(c4)ccc(O)c4)3)O)COC(C=Cc(c1)ccc(c1)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
0.7126 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7126 -0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2088 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7051 -0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7051 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2088 0.7214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2013 -0.4246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6975 -0.1381 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6975 0.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2013 0.7214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3902 -0.8294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1935 0.7213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6993 0.4293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2050 0.7213 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2050 1.3052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6993 1.5972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1935 1.3052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2166 0.7213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7106 1.5971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1313 -0.8647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0670 0.4378 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.6793 -0.0740 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.1209 0.1431 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.5086 0.0386 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.9737 0.5405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5440 0.3357 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.8594 0.1405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2837 -0.1034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8010 -0.3939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4619 0.7325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1048 1.3686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6427 1.8306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9098 2.2931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1102 1.8306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0723 2.2642 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6019 2.4836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1261 2.1505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4003 2.3959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4510 2.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0249 3.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5512 3.0622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9768 3.2197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2654 -0.7568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9613 -1.0609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8188 -1.0763 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8188 -1.6183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2574 -1.8716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7419 -1.5919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2263 -1.8716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2263 -2.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7419 -2.7106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2574 -2.4310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7106 -2.7106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
20 3 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
26 30 1 0 0 0 0
18 24 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 36 1 0 0 0 0
39 42 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
43 28 1 0 0 0 0
S SKP 8
ID FL3FAAGS0046
KNApSAcK_ID C00004186
NAME Apigenin 7-(3'',6''-di-p-coumarylglucoside)
CAS_RN 83529-71-9
FORMULA C39H32O14
EXACTMASS 724.179205732
AVERAGEMASS 724.6629800000001
SMILES O=C(C=Cc(c6)ccc(O)c6)O[C@H]([C@H](O)2)[C@@H](O)C(O[C@H]2Oc(c3)cc(c(C5=O)c(OC(=C5)c(c4)ccc(O)c4)3)O)COC(C=Cc(c1)ccc(c1)O)=O
M END