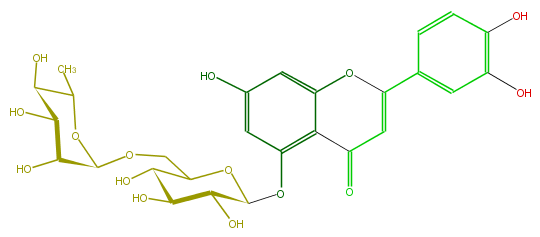
Mol:FL3FACGS0072
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5035 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5035 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5035 -0.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5035 -0.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2109 -0.7032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2109 -0.7032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9254 -0.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9254 -0.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9254 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9254 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2109 0.9467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2109 0.9467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6399 -0.7032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6399 -0.7032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3543 -0.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3543 -0.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3543 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3543 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6399 0.9467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6399 0.9467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0687 0.9467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0687 0.9467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7832 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7832 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4977 0.9467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4977 0.9467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4977 1.7717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4977 1.7717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7832 2.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7832 2.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0687 1.7717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0687 1.7717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6399 -1.5282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6399 -1.5282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2230 0.8812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2230 0.8812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2109 -1.5611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2109 -1.5611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1206 2.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1206 2.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2034 0.5393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2034 0.5393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4688 -1.0478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4688 -1.0478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0561 -1.7625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0561 -1.7625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2578 -1.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2578 -1.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4643 -1.7803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4643 -1.7803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8769 -1.0655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8769 -1.0655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6753 -1.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6753 -1.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7904 -2.1842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7904 -2.1842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9902 -1.3067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9902 -1.3067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6467 -1.6495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6467 -1.6495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1868 -0.7996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1868 -0.7996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9283 -0.7613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9283 -0.7613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1161 0.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1161 0.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9236 0.6392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9236 0.6392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4616 -0.0446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4616 -0.0446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4126 -0.8684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4126 -0.8684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6051 -1.0388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6051 -1.0388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0671 -0.3549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0671 -0.3549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8629 1.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8629 1.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3631 1.0357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3631 1.0357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2034 0.1378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2034 0.1378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0572 -1.0545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0572 -1.0545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACGS0072 | + | ID FL3FACGS0072 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C2Oc(c34)cc(O)cc3OC(c(c5)cc(c(c5)O)O)=CC4=O)O)C(C(O)C(O)1)O | + | SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C2Oc(c34)cc(O)cc3OC(c(c5)cc(c(c5)O)O)=CC4=O)O)C(C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.5035 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5035 -0.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2109 -0.7032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9254 -0.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9254 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2109 0.9467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6399 -0.7032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3543 -0.2907 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3543 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6399 0.9467 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0687 0.9467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7832 0.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4977 0.9467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4977 1.7717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7832 2.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0687 1.7717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6399 -1.5282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2230 0.8812 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2109 -1.5611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1206 2.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2034 0.5393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4688 -1.0478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0561 -1.7625 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2578 -1.5534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4643 -1.7803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8769 -1.0655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6753 -1.2746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7904 -2.1842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9902 -1.3067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6467 -1.6495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1868 -0.7996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9283 -0.7613 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1161 0.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9236 0.6392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4616 -0.0446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4126 -0.8684 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6051 -1.0388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0671 -0.3549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8629 1.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3631 1.0357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2034 0.1378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0572 -1.0545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
14 20 1 0 0 0 0
13 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
24 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
27 31 1 0 0 0 0
31 32 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
34 39 1 0 0 0 0
33 40 1 0 0 0 0
35 41 1 0 0 0 0
36 42 1 0 0 0 0
32 37 1 0 0 0 0
25 19 1 0 0 0 0
S SKP 5
ID FL3FACGS0072
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C2Oc(c34)cc(O)cc3OC(c(c5)cc(c(c5)O)O)=CC4=O)O)C(C(O)C(O)1)O
M END