Mol:FL3FACGS0082
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | 3.8967 2.2486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8967 2.2486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8967 1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8967 1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6111 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6111 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3256 1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3256 1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3256 2.2486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3256 2.2486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6111 2.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6111 2.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1822 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1822 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4677 1.4236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4677 1.4236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7533 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7533 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7533 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7533 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4677 -0.2264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4677 -0.2264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1822 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1822 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0388 1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0388 1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3243 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3243 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3243 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3243 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0388 -0.2264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0388 -0.2264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4677 -0.9446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4677 -0.9446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3902 1.4236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3902 1.4236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0388 -0.9123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0388 -0.9123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8497 2.5512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8497 2.5512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9001 1.0919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9001 1.0919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9090 1.5781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9090 1.5781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4965 0.8636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4965 0.8636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6985 1.0727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6985 1.0727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9052 0.8459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9052 0.8459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3177 1.5605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3177 1.5605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1158 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1158 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2727 1.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2727 1.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7524 2.2140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7524 2.2140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3921 1.0950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3921 1.0950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9451 1.1226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9451 1.1226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8699 0.4330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8699 0.4330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4170 2.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4170 2.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0045 2.0516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0045 2.0516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2065 2.2607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2065 2.2607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4132 2.0339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4132 2.0339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8257 2.7485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8257 2.7485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6238 2.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6238 2.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7807 3.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7807 3.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2604 3.4020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2604 3.4020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9001 2.2830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9001 2.2830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4531 2.3106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4531 2.3106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3779 1.6210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3779 1.6210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0770 0.6411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0770 0.6411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7420 1.0251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7420 1.0251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0770 -0.0412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0770 -0.0412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4841 -0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4841 -0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4841 -1.0590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4841 -1.0590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1986 -1.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1986 -1.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1986 -2.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1986 -2.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4841 -2.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4841 -2.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7696 -2.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7696 -2.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7696 -1.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7696 -1.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4841 -3.4020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4841 -3.4020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1809 -2.6364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1809 -2.6364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 4 21 1 0 0 0 0 | + | 4 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 29 1 0 0 0 0 | + | 36 29 1 0 0 0 0 |
| − | 30 44 1 0 0 0 0 | + | 30 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 48 1 0 0 0 0 | + | 53 48 1 0 0 0 0 |
| − | 51 54 1 0 0 0 0 | + | 51 54 1 0 0 0 0 |
| − | 52 55 1 0 0 0 0 | + | 52 55 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FACGS0082 | + | ID FL3FACGS0082 |
| − | KNApSAcK_ID C00013667 | + | KNApSAcK_ID C00013667 |
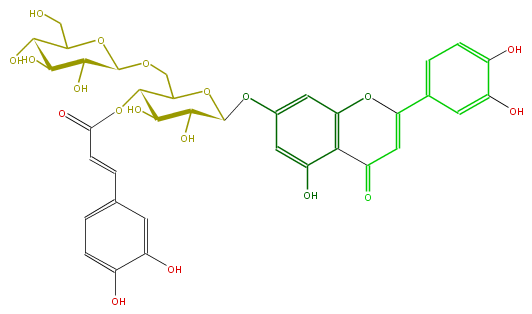
| − | NAME Luteolin 7-glucosyl-(1->6)-(4'''-caffeoylglucoside) | + | NAME Luteolin 7-glucosyl-(1->6)-(4'''-caffeoylglucoside) |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C36H36O19 | + | FORMULA C36H36O19 |
| − | EXACTMASS 772.18507897 | + | EXACTMASS 772.18507897 |
| − | AVERAGEMASS 772.6596400000001 | + | AVERAGEMASS 772.6596400000001 |
| − | SMILES OCC(C1O)OC(OCC(O3)C(C(C(C(Oc(c4)cc(c(C(=O)5)c(OC(c(c6)ccc(O)c6O)=C5)4)O)3)O)O)OC(=O)C=Cc(c2)cc(c(c2)O)O)C(C(O)1)O | + | SMILES OCC(C1O)OC(OCC(O3)C(C(C(C(Oc(c4)cc(c(C(=O)5)c(OC(c(c6)ccc(O)c6O)=C5)4)O)3)O)O)OC(=O)C=Cc(c2)cc(c(c2)O)O)C(C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
3.8967 2.2486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8967 1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6111 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3256 1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3256 2.2486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6111 2.6611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1822 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4677 1.4236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7533 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7533 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4677 -0.2264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1822 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0388 1.4236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3243 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3243 0.1861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0388 -0.2264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4677 -0.9446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3902 1.4236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0388 -0.9123 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8497 2.5512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.9001 1.0919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9090 1.5781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4965 0.8636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6985 1.0727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9052 0.8459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3177 1.5605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1158 1.3515 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2727 1.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7524 2.2140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3921 1.0950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9451 1.1226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8699 0.4330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4170 2.7661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0045 2.0516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2065 2.2607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4132 2.0339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8257 2.7485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6238 2.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7807 3.1251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2604 3.4020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9001 2.2830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4531 2.3106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3779 1.6210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0770 0.6411 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7420 1.0251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0770 -0.0412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4841 -0.3835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4841 -1.0590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1986 -1.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1986 -2.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4841 -2.7090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7696 -2.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7696 -1.4715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4841 -3.4020 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1809 -2.6364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
16 19 1 0 0 0 0
5 20 1 0 0 0 0
4 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 18 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 29 1 0 0 0 0
30 44 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 52 1 0 0 0 0
52 53 2 0 0 0 0
53 48 1 0 0 0 0
51 54 1 0 0 0 0
52 55 1 0 0 0 0
S SKP 8
ID FL3FACGS0082
KNApSAcK_ID C00013667
NAME Luteolin 7-glucosyl-(1->6)-(4'''-caffeoylglucoside)
CAS_RN -
FORMULA C36H36O19
EXACTMASS 772.18507897
AVERAGEMASS 772.6596400000001
SMILES OCC(C1O)OC(OCC(O3)C(C(C(C(Oc(c4)cc(c(C(=O)5)c(OC(c(c6)ccc(O)c6O)=C5)4)O)3)O)O)OC(=O)C=Cc(c2)cc(c(c2)O)O)C(C(O)1)O
M END