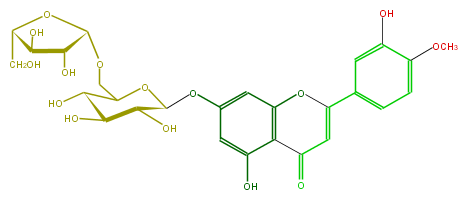
Mol:FL3FAEGS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.2311 -0.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2311 -0.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2311 -1.0318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2311 -1.0318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4698 -1.4365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4698 -1.4365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1707 -1.0318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1707 -1.0318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1707 -0.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1707 -0.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4698 0.1823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4698 0.1823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8717 -1.4365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8717 -1.4365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5725 -1.0318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5725 -1.0318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5725 -0.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5725 -0.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8717 0.1823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8717 0.1823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8717 -2.2148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8717 -2.2148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2732 0.1821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2732 0.1821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9877 -0.2303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9877 -0.2303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7021 0.1821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7021 0.1821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7021 1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7021 1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9877 1.4194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9877 1.4194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2732 1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2732 1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9877 2.2442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9877 2.2442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9306 0.1813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9306 0.1813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4698 -2.2442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4698 -2.2442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7723 0.1896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7723 0.1896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2115 -0.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2115 -0.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4038 -0.2366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4038 -0.2366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6244 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6244 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1907 0.3381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1907 0.3381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8973 -0.0347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8973 -0.0347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4223 -0.0965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4223 -0.0965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0006 -0.4723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0006 -0.4723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6288 -0.7275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6288 -0.7275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3625 0.3910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3625 0.3910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3588 0.9349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3588 0.9349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6254 1.7768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6254 1.7768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1200 1.2556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1200 1.2556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2688 1.2594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2688 1.2594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6623 1.7544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6623 1.7544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7335 2.2158 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7335 2.2158 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0939 1.7584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0939 1.7584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2675 0.7165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2675 0.7165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3819 1.3995 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3819 1.3995 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4922 0.7585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4922 0.7585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6254 0.9253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6254 0.9253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.4922 1.1575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.4922 1.1575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 16 18 1 0 0 0 0 | + | 16 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 31 30 1 0 0 0 0 | + | 31 30 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 32 1 0 0 0 0 | + | 36 32 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 35 31 1 0 0 0 0 | + | 35 31 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 15 39 1 0 0 0 0 | + | 15 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 44 -0.6798 -0.3925 | + | M SBV 1 44 -0.6798 -0.3925 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 46 0.0000 0.8515 | + | M SBV 2 46 0.0000 0.8515 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAEGS0004 | + | ID FL3FAEGS0004 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES O(CC(O2)C(O)C(C(O)C(Oc(c5)cc(O)c(c45)C(=O)C=C(O4)c(c3)cc(O)c(OC)c3)2)O)C(C(O)1)OC(CO)C1O | + | SMILES O(CC(O2)C(O)C(C(O)C(Oc(c5)cc(O)c(c45)C(=O)C=C(O4)c(c3)cc(O)c(OC)c3)2)O)C(C(O)1)OC(CO)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.2311 -0.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2311 -1.0318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4698 -1.4365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1707 -1.0318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1707 -0.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4698 0.1823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8717 -1.4365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5725 -1.0318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5725 -0.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8717 0.1823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8717 -2.2148 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2732 0.1821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9877 -0.2303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7021 0.1821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7021 1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9877 1.4194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2732 1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9877 2.2442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9306 0.1813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4698 -2.2442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7723 0.1896 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2115 -0.5508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4038 -0.2366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6244 -0.2283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1907 0.3381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8973 -0.0347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4223 -0.0965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0006 -0.4723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6288 -0.7275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3625 0.3910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3588 0.9349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6254 1.7768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1200 1.2556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2688 1.2594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6623 1.7544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7335 2.2158 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0939 1.7584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2675 0.7165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.3819 1.3995 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.4922 0.7585 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6254 0.9253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.4922 1.1575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
16 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 19 1 0 0 0 0
26 30 1 0 0 0 0
31 30 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 32 1 0 0 0 0
33 37 1 0 0 0 0
34 38 1 0 0 0 0
35 31 1 0 0 0 0
39 40 1 0 0 0 0
15 39 1 0 0 0 0
41 42 1 0 0 0 0
32 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 OCH3
M SBV 1 44 -0.6798 -0.3925
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 CH2OH
M SBV 2 46 0.0000 0.8515
S SKP 5
ID FL3FAEGS0004
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES O(CC(O2)C(O)C(C(O)C(Oc(c5)cc(O)c(c45)C(=O)C=C(O4)c(c3)cc(O)c(OC)c3)2)O)C(C(O)1)OC(CO)C1O
M END