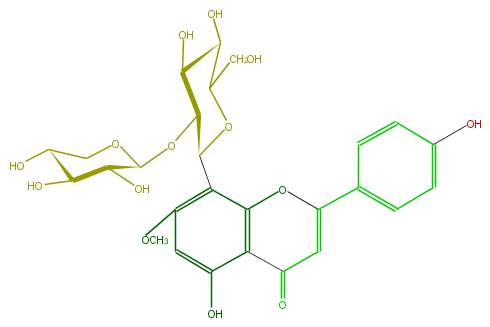
Mol:FL3FCACS0010
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4052 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4052 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4052 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4052 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6907 -2.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6907 -2.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0237 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0237 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0237 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0237 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6907 -0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6907 -0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7382 -2.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7382 -2.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4526 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4526 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4526 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4526 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7382 -0.5246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7382 -0.5246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7382 -2.8178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7382 -2.8178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2463 -0.4982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2463 -0.4982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9992 -0.9330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9992 -0.9330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7521 -0.4982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7521 -0.4982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7521 0.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7521 0.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9992 0.8057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9992 0.8057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2463 0.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2463 0.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5045 0.8055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5045 0.8055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5052 2.4107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5052 2.4107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2829 1.8215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2829 1.8215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9530 0.9731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9530 0.9731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9441 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9441 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3491 0.7492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3491 0.7492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7408 1.4915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7408 1.4915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6910 2.9993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6910 2.9993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4789 0.3617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4789 0.3617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6907 -2.9993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6907 -2.9993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9452 0.2737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9452 0.2737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4686 -0.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4686 -0.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7822 -0.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7822 -0.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1197 -0.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1197 -0.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6011 0.4000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6011 0.4000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2016 0.0831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2016 0.0831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5045 -0.0317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5045 -0.0317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1440 -0.4336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1440 -0.4336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1399 -0.5028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1399 -0.5028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2576 2.5869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2576 2.5869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2633 2.0991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2633 2.0991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6544 1.5693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6544 1.5693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1082 -1.5268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1082 -1.5268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0164 -2.4350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0164 -2.4350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 12 9 1 0 0 0 0 | + | 12 9 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 21 26 1 0 0 0 0 | + | 21 26 1 0 0 0 0 |
| − | 6 22 1 0 0 0 0 | + | 6 22 1 0 0 0 0 |
| − | 3 27 1 0 0 0 0 | + | 3 27 1 0 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 20 37 1 0 0 0 0 | + | 20 37 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 24 38 1 0 0 0 0 | + | 24 38 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 1 40 1 0 0 0 0 | + | 1 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 38 39 | + | M SAL 1 2 38 39 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 43 -0.4776 -0.6076 | + | M SBV 1 43 -0.4776 -0.6076 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 40 41 | + | M SAL 2 2 40 41 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 45 0.7030 0.5898 | + | M SBV 2 45 0.7030 0.5898 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FCACS0010 | + | ID FL3FCACS0010 |
| − | FORMULA C27H30O14 | + | FORMULA C27H30O14 |
| − | EXACTMASS 578.163555668 | + | EXACTMASS 578.163555668 |
| − | AVERAGEMASS 578.5187000000001 | + | AVERAGEMASS 578.5187000000001 |
| − | SMILES O(C)c(c3)c(c(O4)c(C(=O)C=C(c(c5)ccc(O)c5)4)c(O)3)C(C(OC(O2)C(C(O)C(O)C2)O)1)OC(CO)C(O)C(O)1 | + | SMILES O(C)c(c3)c(c(O4)c(C(=O)C=C(c(c5)ccc(O)c5)4)c(O)3)C(C(OC(O2)C(C(O)C(O)C2)O)1)OC(CO)C(O)C(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.4052 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4052 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6907 -2.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0237 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0237 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6907 -0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7382 -2.1745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4526 -1.7621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4526 -0.9371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7382 -0.5246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7382 -2.8178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2463 -0.4982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9992 -0.9330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7521 -0.4982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7521 0.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9992 0.8057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2463 0.3711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5045 0.8055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5052 2.4107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2829 1.8215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9530 0.9731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9441 0.1543 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3491 0.7492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7408 1.4915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6910 2.9993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4789 0.3617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6907 -2.9993 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9452 0.2737 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4686 -0.3555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7822 -0.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1197 -0.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6011 0.4000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2016 0.0831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5045 -0.0317 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1440 -0.4336 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1399 -0.5028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2576 2.5869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2633 2.0991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6544 1.5693 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1082 -1.5268 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0164 -2.4350 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
12 9 1 0 0 0 0
15 18 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
21 26 1 0 0 0 0
6 22 1 0 0 0 0
3 27 1 0 0 0 0
28 29 1 1 0 0 0
29 30 1 1 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 26 1 0 0 0 0
20 37 1 0 0 0 0
38 39 1 0 0 0 0
24 38 1 0 0 0 0
40 41 1 0 0 0 0
1 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 38 39
M SBL 1 1 43
M SMT 1 CH2OH
M SBV 1 43 -0.4776 -0.6076
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 40 41
M SBL 2 1 45
M SMT 2 ^ OCH3
M SBV 2 45 0.7030 0.5898
S SKP 5
ID FL3FCACS0010
FORMULA C27H30O14
EXACTMASS 578.163555668
AVERAGEMASS 578.5187000000001
SMILES O(C)c(c3)c(c(O4)c(C(=O)C=C(c(c5)ccc(O)c5)4)c(O)3)C(C(OC(O2)C(C(O)C(O)C2)O)1)OC(CO)C(O)C(O)1
M END