Mol:FL5FAAGL0110
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6773 -1.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6773 -1.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6773 -1.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6773 -1.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9628 -2.3871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9628 -2.3871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2483 -1.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2483 -1.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2483 -1.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2483 -1.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9628 -0.7370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9628 -0.7370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4663 -2.3871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4663 -2.3871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1808 -1.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1808 -1.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1808 -1.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1808 -1.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4663 -0.7370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4663 -0.7370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4663 -3.0304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4663 -3.0304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0806 -0.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0806 -0.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8088 -0.9986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8088 -0.9986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5371 -0.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5371 -0.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5371 0.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5371 0.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8088 0.6832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8088 0.6832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0806 0.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0806 0.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9628 -3.2119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9628 -3.2119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3165 0.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3165 0.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9043 -2.5005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9043 -2.5005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3452 -0.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3452 -0.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8746 -0.5762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8746 -0.5762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3978 -1.2056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3978 -1.2056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7112 -0.9386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7112 -0.9386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0489 -0.9314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0489 -0.9314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5301 -0.4500 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5301 -0.4500 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1307 -0.7670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1307 -0.7670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4259 -0.7587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4259 -0.7587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0733 -1.1600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0733 -1.1600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0883 -1.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0883 -1.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0123 -1.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0123 -1.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6678 -2.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6678 -2.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3303 -2.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3303 -2.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9968 -2.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9968 -2.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3414 -1.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3414 -1.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6789 -2.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6789 -2.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4120 -2.0523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4120 -2.0523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0785 -1.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0785 -1.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9029 -2.0825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9029 -2.0825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1528 3.1500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1528 3.1500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6546 2.6518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6546 2.6518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0034 2.0397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0034 2.0397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0034 1.3353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0034 1.3353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5017 1.8334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5017 1.8334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1528 2.4456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1528 2.4456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9074 3.7532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9074 3.7532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6433 3.2913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6433 3.2913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5940 1.4021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5940 1.4021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5082 3.1255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5082 3.1255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4259 2.5957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4259 2.5957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5639 -0.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5639 -0.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9263 0.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9263 0.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6429 -2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6429 -2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6154 -3.7532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6154 -3.7532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 21 1 0 0 0 0 | + | 25 21 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 32 20 1 0 0 0 0 | + | 32 20 1 0 0 0 0 |
| − | 40 41 1 1 0 0 0 | + | 40 41 1 1 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 19 1 0 0 0 0 | + | 43 19 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 45 49 1 0 0 0 0 | + | 45 49 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 27 51 1 0 0 0 0 | + | 27 51 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 34 53 1 0 0 0 0 | + | 34 53 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 49 50 | + | M SAL 1 2 49 50 |
| − | M SBL 1 1 55 | + | M SBL 1 1 55 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 55 -0.3554 -0.6799 | + | M SBV 1 55 -0.3554 -0.6799 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 51 52 | + | M SAL 2 2 51 52 |
| − | M SBL 2 1 57 | + | M SBL 2 1 57 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 57 0.4331 -0.4876 | + | M SBV 2 57 0.4331 -0.4876 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 53 54 | + | M SAL 3 2 53 54 |
| − | M SBL 3 1 59 | + | M SBL 3 1 59 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SBV 3 59 -0.6461 -0.0959 | + | M SBV 3 59 -0.6461 -0.0959 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0110 | + | ID FL5FAAGL0110 |
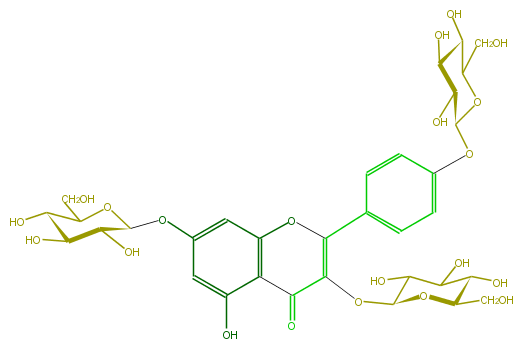
| − | FORMULA C33H40O21 | + | FORMULA C33H40O21 |
| − | EXACTMASS 772.206208342 | + | EXACTMASS 772.206208342 |
| − | AVERAGEMASS 772.6581 | + | AVERAGEMASS 772.6581 |
| − | SMILES O(C1CO)C(OC(=C4c(c5)ccc(OC(O6)C(C(C(O)C6CO)O)O)c5)C(c(c3O4)c(cc(c3)OC(C(O)2)OC(CO)C(O)C2O)O)=O)C(C(C(O)1)O)O | + | SMILES O(C1CO)C(OC(=C4c(c5)ccc(OC(O6)C(C(C(O)C6CO)O)O)c5)C(c(c3O4)c(cc(c3)OC(C(O)2)OC(CO)C(O)C2O)O)=O)C(C(C(O)1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-1.6773 -1.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6773 -1.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9628 -2.3871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2483 -1.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2483 -1.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9628 -0.7370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4663 -2.3871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1808 -1.9746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1808 -1.1496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4663 -0.7370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4663 -3.0304 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0806 -0.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8088 -0.9986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5371 -0.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5371 0.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8088 0.6832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0806 0.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9628 -3.2119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3165 0.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9043 -2.5005 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3452 -0.7186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8746 -0.5762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3978 -1.2056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7112 -0.9386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0489 -0.9314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5301 -0.4500 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1307 -0.7670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4259 -0.7587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0733 -1.1600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0883 -1.4160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0123 -1.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6678 -2.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3303 -2.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9968 -2.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3414 -1.9682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6789 -2.1575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4120 -2.0523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0785 -1.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9029 -2.0825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1528 3.1500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6546 2.6518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0034 2.0397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0034 1.3353 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5017 1.8334 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1528 2.4456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9074 3.7532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6433 3.2913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5940 1.4021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5082 3.1255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4259 2.5957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5639 -0.2794 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9263 0.7164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6429 -2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6154 -3.7532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 21 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
32 20 1 0 0 0 0
40 41 1 1 0 0 0
41 42 1 1 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
40 46 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 19 1 0 0 0 0
49 50 1 0 0 0 0
45 49 1 0 0 0 0
51 52 1 0 0 0 0
27 51 1 0 0 0 0
53 54 1 0 0 0 0
34 53 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 49 50
M SBL 1 1 55
M SMT 1 CH2OH
M SBV 1 55 -0.3554 -0.6799
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 51 52
M SBL 2 1 57
M SMT 2 ^ CH2OH
M SBV 2 57 0.4331 -0.4876
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 53 54
M SBL 3 1 59
M SMT 3 CH2OH
M SBV 3 59 -0.6461 -0.0959
S SKP 5
ID FL5FAAGL0110
FORMULA C33H40O21
EXACTMASS 772.206208342
AVERAGEMASS 772.6581
SMILES O(C1CO)C(OC(=C4c(c5)ccc(OC(O6)C(C(C(O)C6CO)O)O)c5)C(c(c3O4)c(cc(c3)OC(C(O)2)OC(CO)C(O)C2O)O)=O)C(C(C(O)1)O)O
M END