Mol:FL5FAAGS0103
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.1286 4.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1286 4.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1286 3.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1286 3.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8431 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8431 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5575 3.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5575 3.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5575 4.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5575 4.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8431 4.5822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8431 4.5822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4141 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4141 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3004 3.3447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3004 3.3447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0148 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0148 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0148 2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0148 2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3004 1.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3004 1.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4141 2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4141 2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7293 3.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7293 3.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4438 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4438 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4438 2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4438 2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7293 1.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7293 1.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3004 0.9764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3004 0.9764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1582 3.3447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1582 3.3447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2487 1.5907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2487 1.5907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1582 4.5165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1582 4.5165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7293 0.9951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7293 0.9951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4993 -1.2807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4993 -1.2807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9107 -1.9590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9107 -1.9590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2152 -1.2962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2152 -1.2962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6821 -1.9958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6821 -1.9958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2974 -2.6280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2974 -2.6280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7265 -3.3326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7265 -3.3326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3309 -4.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3309 -4.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4939 -4.0758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4939 -4.0758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9231 -3.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9231 -3.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5274 -2.6473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5274 -2.6473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7706 -4.5822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7706 -4.5822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2329 0.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2329 0.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6454 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6454 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1478 0.2387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1478 0.2387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9459 0.0296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9459 0.0296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3585 0.7440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3585 0.7440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5652 0.5174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5652 0.5174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9412 -0.6475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9412 -0.6475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4300 -0.3643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4300 -0.3643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0939 0.1048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0939 0.1048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9259 0.5818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9259 0.5818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 22 24 1 0 0 0 0 | + | 22 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 2 0 0 0 0 | + | 28 29 2 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 29 32 1 0 0 0 0 | + | 29 32 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 19 37 1 0 0 0 0 | + | 19 37 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
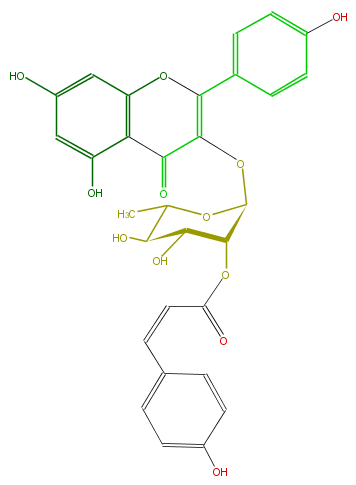
| − | ID FL5FAAGS0103 | + | ID FL5FAAGS0103 |
| − | FORMULA C30H26O12 | + | FORMULA C30H26O12 |
| − | EXACTMASS 578.1424262959999 | + | EXACTMASS 578.1424262959999 |
| − | AVERAGEMASS 578.5202400000001 | + | AVERAGEMASS 578.5202400000001 |
| − | SMILES c(C(=C(OC(O5)C(C(O)C(C5C)O)OC(=O)C=Cc(c4)ccc(c4)O)2)Oc(c3)c(c(O)cc3O)C2=O)(c1)ccc(c1)O | + | SMILES c(C(=C(OC(O5)C(C(O)C(C5C)O)OC(=O)C=Cc(c4)ccc(c4)O)2)Oc(c3)c(c(O)cc3O)C2=O)(c1)ccc(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
1.1286 4.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1286 3.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8431 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5575 3.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5575 4.1697 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8431 4.5822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4141 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3004 3.3447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0148 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0148 2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3004 1.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4141 2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7293 3.3447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4438 2.9322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4438 2.1072 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7293 1.6947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3004 0.9764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1582 3.3447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2487 1.5907 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1582 4.5165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7293 0.9951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4993 -1.2807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9107 -1.9590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2152 -1.2962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6821 -1.9958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2974 -2.6280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7265 -3.3326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3309 -4.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4939 -4.0758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9231 -3.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5274 -2.6473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7706 -4.5822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2329 0.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6454 0.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1478 0.2387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9459 0.0296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3585 0.7440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5652 0.5174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9412 -0.6475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4300 -0.3643 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0939 0.1048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9259 0.5818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 2 0 0 0 0
22 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 28 1 0 0 0 0
28 29 2 0 0 0 0
29 30 1 0 0 0 0
30 31 2 0 0 0 0
31 26 1 0 0 0 0
29 32 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
34 41 1 0 0 0 0
33 42 1 0 0 0 0
22 39 1 0 0 0 0
19 37 1 0 0 0 0
S SKP 5
ID FL5FAAGS0103
FORMULA C30H26O12
EXACTMASS 578.1424262959999
AVERAGEMASS 578.5202400000001
SMILES c(C(=C(OC(O5)C(C(O)C(C5C)O)OC(=O)C=Cc(c4)ccc(c4)O)2)Oc(c3)c(c(O)cc3O)C2=O)(c1)ccc(c1)O
M END