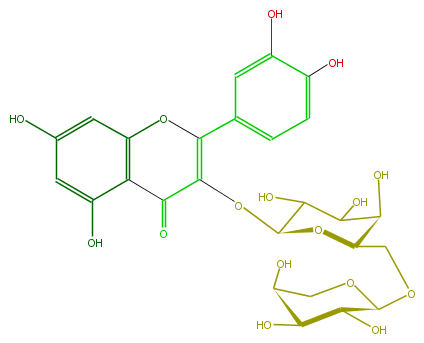
Mol:FL5FACGA0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1910 0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1910 0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1910 -0.1767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1910 -0.1767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4765 -0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4765 -0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7621 -0.1767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7621 -0.1767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7621 0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7621 0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4765 1.0607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4765 1.0607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0476 -0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0476 -0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3332 -0.1767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3332 -0.1767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3332 0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3332 0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0476 1.0607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0476 1.0607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0476 -1.2325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0476 -1.2325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3811 1.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3811 1.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1093 0.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1093 0.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8375 1.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8375 1.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8375 1.9014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8375 1.9014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1093 2.3218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1093 2.3218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3811 1.9014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3811 1.9014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9052 1.0606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9052 1.0606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4765 -1.4139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4765 -1.4139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3250 2.1829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3250 2.1829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7646 -0.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7646 -0.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2475 -1.2445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2475 -1.2445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0476 -1.1611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0476 -1.1611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7757 -1.5140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7757 -1.5140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2930 -0.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2930 -0.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4927 -0.9810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4927 -0.9810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0792 -0.5073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0792 -0.5073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8210 -0.5975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8210 -0.5975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3797 -1.4990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3797 -1.4990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1480 -2.3547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1480 -2.3547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6962 -3.0785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6962 -3.0785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4858 -2.7714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4858 -2.7714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2477 -2.7632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2477 -2.7632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6940 -2.2094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6940 -2.2094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8875 -2.4990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8875 -2.4990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2948 -1.8395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2948 -1.8395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0284 -3.0596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0284 -3.0596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1884 -3.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1884 -3.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9052 -2.4389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9052 -2.4389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4626 -0.6680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4626 -0.6680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1093 3.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1093 3.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2420 -0.0505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2420 -0.0505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 39 29 1 0 0 0 0 | + | 39 29 1 0 0 0 0 |
| − | 22 40 1 0 0 0 0 | + | 22 40 1 0 0 0 0 |
| − | 40 8 1 0 0 0 0 | + | 40 8 1 0 0 0 0 |
| − | 16 41 1 0 0 0 0 | + | 16 41 1 0 0 0 0 |
| − | 25 42 1 0 0 0 0 | + | 25 42 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGA0003 | + | ID FL5FACGA0003 |
| − | FORMULA C26H28O16 | + | FORMULA C26H28O16 |
| − | EXACTMASS 596.137734848 | + | EXACTMASS 596.137734848 |
| − | AVERAGEMASS 596.49092 | + | AVERAGEMASS 596.49092 |
| − | SMILES C(O)(C(O)4)C(O)C(OC(COC(O5)C(O)C(C(O)C5)O)4)OC(=C1c(c3)ccc(O)c3O)C(c(c(O)2)c(cc(O)c2)O1)=O | + | SMILES C(O)(C(O)4)C(O)C(OC(COC(O5)C(O)C(C(O)C5)O)4)OC(=C1c(c3)ccc(O)c3O)C(c(c(O)2)c(cc(O)c2)O1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-3.1910 0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1910 -0.1767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4765 -0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7621 -0.1767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7621 0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4765 1.0607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0476 -0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3332 -0.1767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3332 0.6482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0476 1.0607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0476 -1.2325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3811 1.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1093 0.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8375 1.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8375 1.9014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1093 2.3218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3811 1.9014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9052 1.0606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4765 -1.4139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3250 2.1829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7646 -0.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2475 -1.2445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0476 -1.1611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7757 -1.5140 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2930 -0.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4927 -0.9810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0792 -0.5073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8210 -0.5975 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3797 -1.4990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1480 -2.3547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6962 -3.0785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4858 -2.7714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2477 -2.7632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6940 -2.2094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8875 -2.4990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2948 -1.8395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0284 -3.0596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1884 -3.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9052 -2.4389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4626 -0.6680 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1093 3.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2420 -0.0505 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
24 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
39 29 1 0 0 0 0
22 40 1 0 0 0 0
40 8 1 0 0 0 0
16 41 1 0 0 0 0
25 42 1 0 0 0 0
S SKP 5
ID FL5FACGA0003
FORMULA C26H28O16
EXACTMASS 596.137734848
AVERAGEMASS 596.49092
SMILES C(O)(C(O)4)C(O)C(OC(COC(O5)C(O)C(C(O)C5)O)4)OC(=C1c(c3)ccc(O)c3O)C(c(c(O)2)c(cc(O)c2)O1)=O
M END