Mol:FL5FACGL0017
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6582 -0.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6582 -0.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6582 -1.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6582 -1.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9436 -1.5242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9436 -1.5242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2291 -1.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2291 -1.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2291 -0.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2291 -0.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9436 0.1259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9436 0.1259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4854 -1.5242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4854 -1.5242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1999 -1.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1999 -1.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1999 -0.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1999 -0.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4854 0.1259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4854 0.1259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4854 -2.1675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4854 -2.1675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9346 0.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9346 0.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6629 -0.3214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6629 -0.3214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3911 0.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3911 0.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3911 0.9399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3911 0.9399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6629 1.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6629 1.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9346 0.9399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9346 0.9399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9436 -2.2010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9436 -2.2010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1705 1.3899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1705 1.3899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9330 -1.5783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9330 -1.5783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5116 0.2060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5116 0.2060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6629 2.2010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6629 2.2010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9529 -0.9836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9529 -0.9836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5664 -1.6528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5664 -1.6528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3095 -1.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3095 -1.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0570 -1.6528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0570 -1.6528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4435 -0.9836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4435 -0.9836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7004 -1.1959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7004 -1.1959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2372 -1.1754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2372 -1.1754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0064 -0.6660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0064 -0.6660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1355 -1.1691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1355 -1.1691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0133 0.2456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0133 0.2456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5366 -0.3838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5366 -0.3838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8501 -0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8501 -0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1876 -0.1097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1876 -0.1097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6690 0.3718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6690 0.3718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2696 0.0549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2696 0.0549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6733 -0.1355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6733 -0.1355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2798 -0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2798 -0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4567 -0.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4567 -0.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6215 0.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6215 0.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6812 0.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6812 0.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0917 1.5821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0917 1.5821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7634 -1.5717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7634 -1.5717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6812 -2.1015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6812 -2.1015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 21 1 0 0 0 0 | + | 35 21 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 26 44 1 0 0 0 0 | + | 26 44 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 41 42 43 | + | M SAL 1 3 41 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^COOH | + | M SMT 1 ^COOH |
| − | M SBV 1 47 0.3519 -0.6096 | + | M SBV 1 47 0.3519 -0.6096 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 44 45 | + | M SAL 2 2 44 45 |
| − | M SBL 2 1 49 | + | M SBL 2 1 49 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 49 -0.7064 -0.0811 | + | M SBV 2 49 -0.7064 -0.0811 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGL0017 | + | ID FL5FACGL0017 |
| − | FORMULA C27H28O18 | + | FORMULA C27H28O18 |
| − | EXACTMASS 640.1275640920001 | + | EXACTMASS 640.1275640920001 |
| − | AVERAGEMASS 640.5004200000001 | + | AVERAGEMASS 640.5004200000001 |
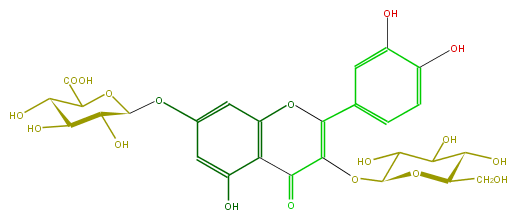
| − | SMILES O=C(c21)C(OC(C(O)5)OC(C(C(O)5)O)CO)=C(c(c4)ccc(O)c4O)Oc1cc(OC(O3)C(C(C(O)C3C(O)=O)O)O)cc2O | + | SMILES O=C(c21)C(OC(C(O)5)OC(C(C(O)5)O)CO)=C(c(c4)ccc(O)c4O)Oc1cc(OC(O3)C(C(C(O)C3C(O)=O)O)O)cc2O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
-1.6582 -0.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6582 -1.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9436 -1.5242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2291 -1.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2291 -0.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9436 0.1259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4854 -1.5242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1999 -1.1118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1999 -0.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4854 0.1259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4854 -2.1675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9346 0.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6629 -0.3214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3911 0.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3911 0.9399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6629 1.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9346 0.9399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9436 -2.2010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1705 1.3899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9330 -1.5783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5116 0.2060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6629 2.2010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9529 -0.9836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5664 -1.6528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3095 -1.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0570 -1.6528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4435 -0.9836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7004 -1.1959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2372 -1.1754 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0064 -0.6660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1355 -1.1691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0133 0.2456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5366 -0.3838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8501 -0.1167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1876 -0.1097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6690 0.3718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2696 0.0549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6733 -0.1355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2798 -0.4200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4567 -0.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6215 0.6644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6812 0.6644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0917 1.5821 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7634 -1.5717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6812 -2.1015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
1 21 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
28 30 1 0 0 0 0
27 31 1 0 0 0 0
24 20 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 21 1 0 0 0 0
41 42 2 0 0 0 0
41 43 1 0 0 0 0
37 41 1 0 0 0 0
44 45 1 0 0 0 0
26 44 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 41 42 43
M SBL 1 1 47
M SMT 1 ^COOH
M SBV 1 47 0.3519 -0.6096
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 44 45
M SBL 2 1 49
M SMT 2 CH2OH
M SBV 2 49 -0.7064 -0.0811
S SKP 5
ID FL5FACGL0017
FORMULA C27H28O18
EXACTMASS 640.1275640920001
AVERAGEMASS 640.5004200000001
SMILES O=C(c21)C(OC(C(O)5)OC(C(C(O)5)O)CO)=C(c(c4)ccc(O)c4O)Oc1cc(OC(O3)C(C(C(O)C3C(O)=O)O)O)cc2O
M END