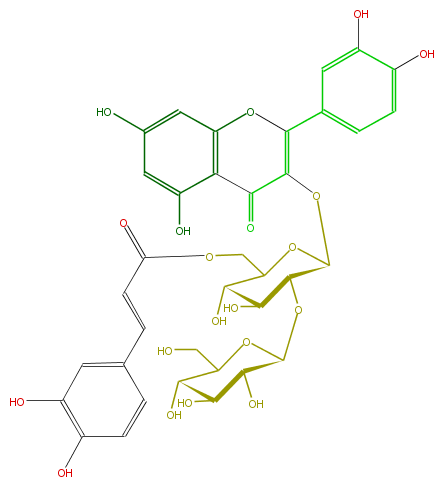
Mol:FL5FACGS0105
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.7207 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7207 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0062 2.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0062 2.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0062 3.4509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0062 3.4509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7207 3.8634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7207 3.8634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4351 3.4509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4351 3.4509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4351 2.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4351 2.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2917 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2917 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5773 2.6259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5773 2.6259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1372 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1372 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1372 1.3884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1372 1.3884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5773 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5773 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2917 1.3884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2917 1.3884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8517 2.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8517 2.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5662 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5662 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5662 1.3884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5662 1.3884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8517 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8517 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5773 0.3151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5773 0.3151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2806 2.6259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2806 2.6259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8907 0.9429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8907 0.9429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8517 0.2492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8517 0.2492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0270 3.7926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0270 3.7926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7207 4.5892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7207 4.5892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0453 -0.8837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0453 -0.8837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7687 -1.2808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7687 -1.2808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3428 -0.6879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3428 -0.6879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1389 -0.4703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1389 -0.4703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4154 -0.0732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4154 -0.0732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8413 -0.6660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8413 -0.6660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4032 -0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4032 -0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2435 -0.2623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2435 -0.2623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1173 -1.5475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1173 -1.5475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2508 -1.2920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2508 -1.2920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5281 -1.3240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5281 -1.3240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8740 -2.7734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8740 -2.7734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1506 -3.1705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1506 -3.1705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4235 -2.5776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4235 -2.5776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2196 -2.3600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2196 -2.3600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4962 -1.9629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4962 -1.9629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0780 -2.5557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0780 -2.5557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5161 -2.1363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5161 -2.1363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0700 -2.1483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0700 -2.1483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0366 -3.4372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0366 -3.4372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6685 -3.1817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6685 -3.1817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6088 -3.2137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6088 -3.2137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5616 -0.3017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5616 -0.3017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9755 0.4107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9755 0.4107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9715 -1.0164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9715 -1.0164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5576 -1.7287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5576 -1.7287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9676 -2.4434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9676 -2.4434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7926 -2.4456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7926 -2.4456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2031 -3.1612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2031 -3.1612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7887 -3.8746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7887 -3.8746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9637 -3.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9637 -3.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5531 -3.1567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5531 -3.1567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1987 -4.5892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1987 -4.5892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0270 -3.1635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0270 -3.1635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 33 1 0 0 0 0 | + | 37 33 1 0 0 0 0 |
| − | 26 19 1 0 0 0 0 | + | 26 19 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 54 49 1 0 0 0 0 | + | 54 49 1 0 0 0 0 |
| − | 52 55 1 0 0 0 0 | + | 52 55 1 0 0 0 0 |
| − | 51 56 1 0 0 0 0 | + | 51 56 1 0 0 0 0 |
| − | 45 30 1 0 0 0 0 | + | 45 30 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGS0105 | + | ID FL5FACGS0105 |
| − | KNApSAcK_ID C00013877 | + | KNApSAcK_ID C00013877 |
| − | NAME Quercetin 3-(6''-caffeoylsophoroside);2-(3,4-Dihydroxyphenyl)-3-[[6-O-[(2E)-3-(3,4-dihydroxyphenyl)-1-oxo-2-propenyl]-2-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl]oxy]-5,7-dihydroxy-4H-1-benzopyran-4-one | + | NAME Quercetin 3-(6''-caffeoylsophoroside);2-(3,4-Dihydroxyphenyl)-3-[[6-O-[(2E)-3-(3,4-dihydroxyphenyl)-1-oxo-2-propenyl]-2-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl]oxy]-5,7-dihydroxy-4H-1-benzopyran-4-one |
| − | CAS_RN 365544-18-9 | + | CAS_RN 365544-18-9 |
| − | FORMULA C36H36O20 | + | FORMULA C36H36O20 |
| − | EXACTMASS 788.179993592 | + | EXACTMASS 788.179993592 |
| − | AVERAGEMASS 788.65904 | + | AVERAGEMASS 788.65904 |
| − | SMILES c(c6O)(cc(cc6)C(O5)=C(C(=O)c(c54)c(cc(c4)O)O)OC(C(OC(C(O)3)OC(CO)C(O)C(O)3)2)OC(C(O)C2O)COC(C=Cc(c1)cc(O)c(O)c1)=O)O | + | SMILES c(c6O)(cc(cc6)C(O5)=C(C(=O)c(c54)c(cc(c4)O)O)OC(C(OC(C(O)3)OC(CO)C(O)C(O)3)2)OC(C(O)C2O)COC(C=Cc(c1)cc(O)c(O)c1)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
2.7207 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0062 2.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0062 3.4509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7207 3.8634 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4351 3.4509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4351 2.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2917 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5773 2.6259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1372 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1372 1.3884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5773 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2917 1.3884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8517 2.6259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5662 2.2134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5662 1.3884 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8517 0.9759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5773 0.3151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2806 2.6259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8907 0.9429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8517 0.2492 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0270 3.7926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7207 4.5892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0453 -0.8837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7687 -1.2808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3428 -0.6879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1389 -0.4703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4154 -0.0732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8413 -0.6660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4032 -0.2466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2435 -0.2623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1173 -1.5475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2508 -1.2920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5281 -1.3240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8740 -2.7734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1506 -3.1705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4235 -2.5776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2196 -2.3600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4962 -1.9629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0780 -2.5557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5161 -2.1363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0700 -2.1483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0366 -3.4372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6685 -3.1817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6088 -3.2137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5616 -0.3017 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9755 0.4107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9715 -1.0164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5576 -1.7287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9676 -2.4434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7926 -2.4456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2031 -3.1612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7887 -3.8746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9637 -3.8723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5531 -3.1567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1987 -4.5892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0270 -3.1635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
37 33 1 0 0 0 0
26 19 1 0 0 0 0
45 46 2 0 0 0 0
45 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 53 1 0 0 0 0
53 54 2 0 0 0 0
54 49 1 0 0 0 0
52 55 1 0 0 0 0
51 56 1 0 0 0 0
45 30 1 0 0 0 0
S SKP 8
ID FL5FACGS0105
KNApSAcK_ID C00013877
NAME Quercetin 3-(6''-caffeoylsophoroside);2-(3,4-Dihydroxyphenyl)-3-[[6-O-[(2E)-3-(3,4-dihydroxyphenyl)-1-oxo-2-propenyl]-2-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl]oxy]-5,7-dihydroxy-4H-1-benzopyran-4-one
CAS_RN 365544-18-9
FORMULA C36H36O20
EXACTMASS 788.179993592
AVERAGEMASS 788.65904
SMILES c(c6O)(cc(cc6)C(O5)=C(C(=O)c(c54)c(cc(c4)O)O)OC(C(OC(C(O)3)OC(CO)C(O)C(O)3)2)OC(C(O)C2O)COC(C=Cc(c1)cc(O)c(O)c1)=O)O
M END