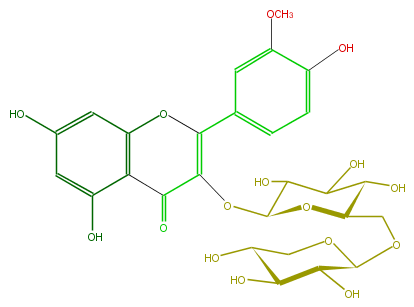
Mol:FL5FADGL0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.0452 -0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0452 -0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0452 -1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0452 -1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3307 -1.4245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3307 -1.4245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6163 -1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6163 -1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6163 -0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6163 -0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3307 0.2255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3307 0.2255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9018 -1.4245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9018 -1.4245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1873 -1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1873 -1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1873 -0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1873 -0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9018 0.2255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9018 0.2255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9018 -2.0678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9018 -2.0678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5269 0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5269 0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2551 -0.1951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2551 -0.1951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9833 0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9833 0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9833 1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9833 1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2551 1.4865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2551 1.4865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5269 1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5269 1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7594 0.2253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7594 0.2253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3307 -2.2493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3307 -2.2493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6844 1.5420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6844 1.5420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5803 -1.1617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5803 -1.1617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1780 -1.8584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1780 -1.8584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9516 -1.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9516 -1.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7299 -1.8584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7299 -1.8584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1322 -1.1617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1322 -1.1617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3586 -1.3827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3586 -1.3827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1332 -1.1424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1332 -1.1424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9104 -0.7906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9104 -0.7906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7248 -1.2571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7248 -1.2571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4674 -1.8584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4674 -1.8584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8605 -2.4680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8605 -2.4680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4088 -3.1919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4088 -3.1919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1984 -2.8848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1984 -2.8848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9604 -2.8766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9604 -2.8766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4067 -2.3228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4067 -2.3228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6002 -2.6124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6002 -2.6124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1341 -2.6622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1341 -2.6622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6516 -3.1013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6516 -3.1013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7966 -3.3775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7966 -3.3775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7594 -2.4052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7594 -2.4052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3873 -1.6127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3873 -1.6127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2492 2.2251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2492 2.2251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8161 3.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8161 3.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 40 30 1 0 0 0 0 | + | 40 30 1 0 0 0 0 |
| − | 22 41 1 0 0 0 0 | + | 22 41 1 0 0 0 0 |
| − | 41 8 1 0 0 0 0 | + | 41 8 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 47 0.0059 -0.7385 | + | M SBV 1 47 0.0059 -0.7385 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FADGL0004 | + | ID FL5FADGL0004 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES C(O)(C(O)1)C(O)C(OC(C4=O)=C(Oc(c5)c(c(O)cc5O)4)c(c3)ccc(c3OC)O)OC(COC(O2)C(O)C(C(O)C2)O)1 | + | SMILES C(O)(C(O)1)C(O)C(OC(C4=O)=C(Oc(c5)c(c(O)cc5O)4)c(c3)ccc(c3OC)O)OC(COC(O2)C(O)C(C(O)C2)O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-3.0452 -0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0452 -1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3307 -1.4245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6163 -1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6163 -0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3307 0.2255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9018 -1.4245 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1873 -1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1873 -0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9018 0.2255 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9018 -2.0678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5269 0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2551 -0.1951 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9833 0.2253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9833 1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2551 1.4865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5269 1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7594 0.2253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3307 -2.2493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6844 1.5420 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5803 -1.1617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1780 -1.8584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9516 -1.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7299 -1.8584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1322 -1.1617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3586 -1.3827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1332 -1.1424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9104 -0.7906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7248 -1.2571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4674 -1.8584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8605 -2.4680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4088 -3.1919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1984 -2.8848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9604 -2.8766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4067 -2.3228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6002 -2.6124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1341 -2.6622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6516 -3.1013 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7966 -3.3775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7594 -2.4052 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3873 -1.6127 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2492 2.2251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8161 3.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
40 30 1 0 0 0 0
22 41 1 0 0 0 0
41 8 1 0 0 0 0
42 43 1 0 0 0 0
16 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 OCH3
M SBV 1 47 0.0059 -0.7385
S SKP 5
ID FL5FADGL0004
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES C(O)(C(O)1)C(O)C(OC(C4=O)=C(Oc(c5)c(c(O)cc5O)4)c(c3)ccc(c3OC)O)OC(COC(O2)C(O)C(C(O)C2)O)1
M END