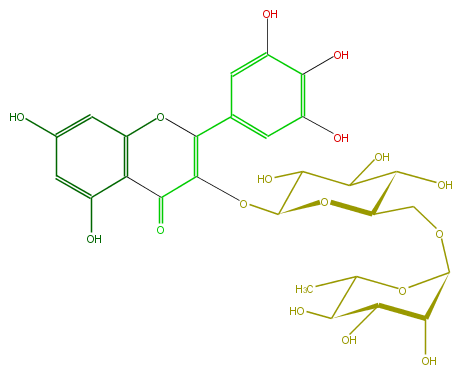
Mol:FL5FAGGL0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.5856 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5856 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5856 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5856 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8846 -0.2132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8846 -0.2132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1836 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1836 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1836 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1836 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8846 1.4057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8846 1.4057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4826 -0.2132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4826 -0.2132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7816 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7816 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7816 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7816 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4826 1.4057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4826 1.4057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4826 -0.8443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4826 -0.8443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0808 1.4056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0808 1.4056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6337 0.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6337 0.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3482 1.4056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3482 1.4056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3482 2.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3482 2.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6337 2.6430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6337 2.6430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0808 2.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0808 2.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8846 -1.0224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8846 -1.0224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4145 -1.7975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4145 -1.7975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9260 -2.6435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9260 -2.6435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8654 -2.3750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8654 -2.3750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8105 -2.6435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8105 -2.6435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2864 -1.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2864 -1.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3597 -2.0659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3597 -2.0659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5072 -2.0405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5072 -2.0405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3472 0.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3472 0.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8587 -0.5548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8587 -0.5548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7982 -0.2862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7982 -0.2862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7432 -0.5548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7432 -0.5548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2318 0.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2318 0.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2924 0.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2924 0.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1830 -0.3245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1830 -0.3245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8661 0.6090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8661 0.6090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1333 0.0499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1333 0.0499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5591 -0.3925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5591 -0.3925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1019 -0.9286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1019 -0.9286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3168 -2.4803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3168 -2.4803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2117 -3.0507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2117 -3.0507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8105 -3.4678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8105 -3.4678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6678 0.1648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6678 0.1648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2864 1.4056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2864 1.4056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0625 2.6429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0625 2.6429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6337 3.4678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6337 3.4678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0625 0.9932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0625 0.9932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 23 36 1 0 0 0 0 | + | 23 36 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 20 37 1 0 0 0 0 | + | 20 37 1 0 0 0 0 |
| − | 21 38 1 0 0 0 0 | + | 21 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 32 8 1 0 0 0 0 | + | 32 8 1 0 0 0 0 |
| − | 27 32 1 0 0 0 0 | + | 27 32 1 0 0 0 0 |
| − | 26 40 1 0 0 0 0 | + | 26 40 1 0 0 0 0 |
| − | 1 41 1 0 0 0 0 | + | 1 41 1 0 0 0 0 |
| − | 15 42 1 0 0 0 0 | + | 15 42 1 0 0 0 0 |
| − | 16 43 1 0 0 0 0 | + | 16 43 1 0 0 0 0 |
| − | 14 44 1 0 0 0 0 | + | 14 44 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAGGL0003 | + | ID FL5FAGGL0003 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES O(C1C)C(OCC(O2)C(C(O)C(C(OC(=C4c(c5)cc(c(c(O)5)O)O)C(=O)c(c3O)c(O4)cc(c3)O)2)O)O)C(C(O)C(O)1)O | + | SMILES O(C1C)C(OCC(O2)C(C(O)C(C(OC(=C4c(c5)cc(c(c(O)5)O)O)C(=O)c(c3O)c(O4)cc(c3)O)2)O)O)C(C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-3.5856 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5856 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8846 -0.2132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1836 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1836 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8846 1.4057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4826 -0.2132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7816 0.1915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7816 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4826 1.4057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4826 -0.8443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0808 1.4056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6337 0.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3482 1.4056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3482 2.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6337 2.6430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0808 2.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8846 -1.0224 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4145 -1.7975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9260 -2.6435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8654 -2.3750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8105 -2.6435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2864 -1.7147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3597 -2.0659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5072 -2.0405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3472 0.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8587 -0.5548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7982 -0.2862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7432 -0.5548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2318 0.2913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2924 0.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1830 -0.3245 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8661 0.6090 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1333 0.0499 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5591 -0.3925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1019 -0.9286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3168 -2.4803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2117 -3.0507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8105 -3.4678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6678 0.1648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2864 1.4056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0625 2.6429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6337 3.4678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0625 0.9932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 1 0 0 0
28 29 1 1 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
31 33 1 0 0 0 0
30 34 1 0 0 0 0
29 35 1 0 0 0 0
23 36 1 0 0 0 0
35 36 1 0 0 0 0
20 37 1 0 0 0 0
21 38 1 0 0 0 0
22 39 1 0 0 0 0
32 8 1 0 0 0 0
27 32 1 0 0 0 0
26 40 1 0 0 0 0
1 41 1 0 0 0 0
15 42 1 0 0 0 0
16 43 1 0 0 0 0
14 44 1 0 0 0 0
S SKP 5
ID FL5FAGGL0003
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES O(C1C)C(OCC(O2)C(C(O)C(C(OC(=C4c(c5)cc(c(c(O)5)O)O)C(=O)c(c3O)c(O4)cc(c3)O)2)O)O)C(C(O)C(O)1)O
M END