Mol:FL5FCAGL0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 63 69 0 0 0 0 0 0 0 0999 V2000 | + | 63 69 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.2558 -3.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2558 -3.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2558 -3.9869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2558 -3.9869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6995 -4.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6995 -4.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1432 -3.9869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1432 -3.9869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1432 -3.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1432 -3.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6995 -3.0234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6995 -3.0234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5869 -4.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5869 -4.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0305 -3.9869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0305 -3.9869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0305 -3.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0305 -3.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5869 -3.0234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5869 -3.0234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5869 -4.8089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5869 -4.8089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3301 -2.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3301 -2.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7631 -3.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7631 -3.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1961 -2.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1961 -2.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1961 -2.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1961 -2.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7631 -1.9177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7631 -1.9177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3301 -2.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3301 -2.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6995 -4.9502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6995 -4.9502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4107 -1.8947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4107 -1.8947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2033 -4.4171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2033 -4.4171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0445 -3.6857 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.0445 -3.6857 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.3453 -4.2067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.3453 -4.2067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2331 -4.0414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2331 -4.0414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8151 -4.2067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.8151 -4.2067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.1160 -3.6857 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.1160 -3.6857 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.5375 -3.8509 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.5375 -3.8509 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.6032 -3.8354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6032 -3.8354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5791 -3.3764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5791 -3.3764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5501 -3.9363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5501 -3.9363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2899 -0.7180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2899 -0.7180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2780 -1.0459 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.2780 -1.0459 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.0978 -0.4154 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.0978 -0.4154 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.2780 0.2190 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.2780 0.2190 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.2899 0.5469 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.2899 0.5469 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.1097 -0.0837 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.1097 -0.0837 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.5461 0.9906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5461 0.9906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5127 0.5542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5127 0.5542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5154 -0.6565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5154 -0.6565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2722 0.6881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2722 0.6881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6859 1.2788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6859 1.2788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6264 -0.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6264 -0.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4605 -0.1443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4605 -0.1443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8144 -0.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8144 -0.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9408 0.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9408 0.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7750 0.4686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7750 0.4686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2553 1.1546 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 4.2553 1.1546 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 4.8676 0.7259 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 4.8676 0.7259 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 5.5449 1.0417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5449 1.0417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6100 1.7862 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 5.6100 1.7862 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.9978 2.2149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9978 2.2149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3204 1.8991 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 4.3204 1.8991 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 3.4270 1.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4270 1.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7245 0.4395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7245 0.4395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0132 1.3656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0132 1.3656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1519 1.8818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1519 1.8818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9037 2.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9037 2.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8064 0.0268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8064 0.0268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7859 0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7859 0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5278 -0.5360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5278 -0.5360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6130 -2.7258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6130 -2.7258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1129 -1.8597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1129 -1.8597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0147 -4.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0147 -4.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0147 -5.3240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0147 -5.3240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 19 31 1 0 0 0 0 | + | 19 31 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 46 1 0 0 0 0 | + | 51 46 1 0 0 0 0 |
| − | 46 52 1 6 0 0 0 | + | 46 52 1 6 0 0 0 |
| − | 47 53 1 6 0 0 0 | + | 47 53 1 6 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 51 54 1 6 0 0 0 | + | 51 54 1 6 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 49 55 1 1 0 0 0 | + | 49 55 1 1 0 0 0 |
| − | 51 56 1 0 0 0 0 | + | 51 56 1 0 0 0 0 |
| − | 47 57 1 0 0 0 0 | + | 47 57 1 0 0 0 0 |
| − | 35 58 1 0 0 0 0 | + | 35 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 1 60 1 0 0 0 0 | + | 1 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 24 62 1 0 0 0 0 | + | 24 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 62 63 | + | M SAL 3 2 62 63 |
| − | M SBL 3 1 68 | + | M SBL 3 1 68 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SVB 3 68 1.1681 -4.18 | + | M SVB 3 68 1.1681 -4.18 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 58 59 | + | M SAL 2 2 58 59 |
| − | M SBL 2 1 64 | + | M SBL 2 1 64 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 64 -0.6303 0.3999 | + | M SVB 2 64 -0.6303 0.3999 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 60 61 | + | M SAL 1 2 60 61 |
| − | M SBL 1 1 66 | + | M SBL 1 1 66 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 66 -4.613 -2.7258 | + | M SVB 1 66 -4.613 -2.7258 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FCAGL0011 | + | ID FL5FCAGL0011 |
| − | KNApSAcK_ID C00005931 | + | KNApSAcK_ID C00005931 |
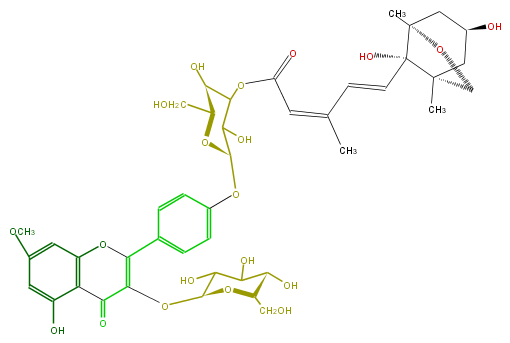
| − | NAME Rhamnocitrin 3-glucoside-4'-(3''-dihydrophaseoylglucoside) | + | NAME Rhamnocitrin 3-glucoside-4'-(3''-dihydrophaseoylglucoside) |
| − | CAS_RN 142449-94-3 | + | CAS_RN 142449-94-3 |
| − | FORMULA C43H52O20 | + | FORMULA C43H52O20 |
| − | EXACTMASS 888.305194104 | + | EXACTMASS 888.305194104 |
| − | AVERAGEMASS 888.8609799999999 | + | AVERAGEMASS 888.8609799999999 |
| − | SMILES [C@H]([C@H](CO)1)(C(OC(C=C(C)C=C[C@]([C@]7(C)6)(O)[C@@](CO7)(C[C@H](O)C6)C)=O)C([C@@H](Oc(c5)ccc(c5)C(O3)=C(O[C@H](O4)C(O)C(O)[C@@H](O)[C@H]4CO)C(c(c23)c(cc(c2)OC)O)=O)O1)O)O | + | SMILES [C@H]([C@H](CO)1)(C(OC(C=C(C)C=C[C@]([C@]7(C)6)(O)[C@@](CO7)(C[C@H](O)C6)C)=O)C([C@@H](Oc(c5)ccc(c5)C(O3)=C(O[C@H](O4)C(O)C(O)[C@@H](O)[C@H]4CO)C(c(c23)c(cc(c2)OC)O)=O)O1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
63 69 0 0 0 0 0 0 0 0999 V2000
-4.2558 -3.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2558 -3.9869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6995 -4.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1432 -3.9869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1432 -3.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6995 -3.0234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5869 -4.3081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0305 -3.9869 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0305 -3.3446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5869 -3.0234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5869 -4.8089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3301 -2.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7631 -3.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1961 -2.8997 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1961 -2.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7631 -1.9177 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3301 -2.2451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6995 -4.9502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4107 -1.8947 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2033 -4.4171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0445 -3.6857 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.3453 -4.2067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2331 -4.0414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8151 -4.2067 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.1160 -3.6857 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.5375 -3.8509 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.6032 -3.8354 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5791 -3.3764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5501 -3.9363 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2899 -0.7180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2780 -1.0459 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.0978 -0.4154 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.2780 0.2190 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.2899 0.5469 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.1097 -0.0837 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.5461 0.9906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5127 0.5542 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5154 -0.6565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2722 0.6881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6859 1.2788 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6264 -0.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4605 -0.1443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8144 -0.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9408 0.5416 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7750 0.4686 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2553 1.1546 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
4.8676 0.7259 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
5.5449 1.0417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6100 1.7862 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.9978 2.2149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3204 1.8991 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
3.4270 1.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7245 0.4395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0132 1.3656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1519 1.8818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9037 2.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8064 0.0268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7859 0.1346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5278 -0.5360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6130 -2.7258 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1129 -1.8597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0147 -4.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0147 -5.3240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
20 8 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
34 36 1 0 0 0 0
33 37 1 0 0 0 0
32 38 1 0 0 0 0
19 31 1 0 0 0 0
37 39 1 0 0 0 0
39 40 2 0 0 0 0
39 41 1 0 0 0 0
41 42 2 0 0 0 0
42 43 1 0 0 0 0
42 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
51 46 1 0 0 0 0
46 52 1 6 0 0 0
47 53 1 6 0 0 0
53 54 1 0 0 0 0
51 54 1 6 0 0 0
47 48 1 0 0 0 0
49 55 1 1 0 0 0
51 56 1 0 0 0 0
47 57 1 0 0 0 0
35 58 1 0 0 0 0
58 59 1 0 0 0 0
1 60 1 0 0 0 0
60 61 1 0 0 0 0
24 62 1 0 0 0 0
62 63 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 62 63
M SBL 3 1 68
M SMT 3 CH2OH
M SVB 3 68 1.1681 -4.18
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 58 59
M SBL 2 1 64
M SMT 2 CH2OH
M SVB 2 64 -0.6303 0.3999
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 60 61
M SBL 1 1 66
M SMT 1 OCH3
M SVB 1 66 -4.613 -2.7258
S SKP 8
ID FL5FCAGL0011
KNApSAcK_ID C00005931
NAME Rhamnocitrin 3-glucoside-4'-(3''-dihydrophaseoylglucoside)
CAS_RN 142449-94-3
FORMULA C43H52O20
EXACTMASS 888.305194104
AVERAGEMASS 888.8609799999999
SMILES [C@H]([C@H](CO)1)(C(OC(C=C(C)C=C[C@]([C@]7(C)6)(O)[C@@](CO7)(C[C@H](O)C6)C)=O)C([C@@H](Oc(c5)ccc(c5)C(O3)=C(O[C@H](O4)C(O)C(O)[C@@H](O)[C@H]4CO)C(c(c23)c(cc(c2)OC)O)=O)O1)O)O
M END