Mol:FL5FCCGS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.3553 1.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3553 1.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3553 0.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3553 0.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6407 0.2837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6407 0.2837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9262 0.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9262 0.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9262 1.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9262 1.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6407 1.9338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6407 1.9338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2117 0.2837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2117 0.2837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5028 0.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5028 0.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5028 1.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5028 1.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2117 1.9338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2117 1.9338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2117 -0.3595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2117 -0.3595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4025 2.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4025 2.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1307 1.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1307 1.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8589 2.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8589 2.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8589 2.9336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8589 2.9336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1307 3.3539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1307 3.3539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4025 2.9336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4025 2.9336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6407 -0.5410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6407 -0.5410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6383 3.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6383 3.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2689 0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2689 0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1307 4.1946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1307 4.1946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0514 -1.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0514 -1.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7391 -1.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7391 -1.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5209 -0.7248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5209 -0.7248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7391 0.0432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7391 0.0432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0514 0.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0514 0.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2695 -0.3232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2695 -0.3232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3140 -0.3243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3140 -0.3243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1263 -1.8138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1263 -1.8138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5672 -2.1593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5672 -2.1593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0362 -1.1182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0362 -1.1182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3272 -2.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3272 -2.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9985 -3.4726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9985 -3.4726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6304 -3.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6304 -3.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2663 -3.4726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2663 -3.4726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5950 -2.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5950 -2.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9629 -3.0839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9629 -3.0839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2343 -3.8715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2343 -3.8715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3952 -3.2058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3952 -3.2058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7251 -2.8576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7251 -2.8576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2225 -4.1946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2225 -4.1946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9961 1.8913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9961 1.8913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6383 3.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6383 3.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 20 1 0 0 0 0 | + | 26 20 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 1 42 1 0 0 0 0 | + | 1 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 47 0.6408 -0.3700 | + | M SBV 1 47 0.6408 -0.3700 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FCCGS0006 | + | ID FL5FCCGS0006 |
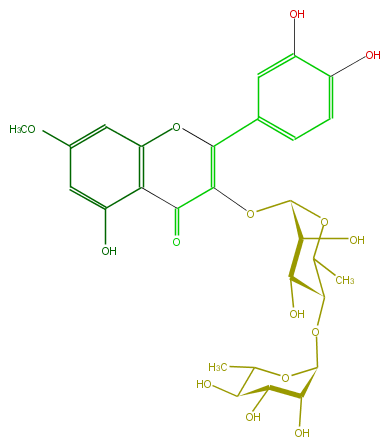
| − | FORMULA C28H32O15 | + | FORMULA C28H32O15 |
| − | EXACTMASS 608.174120354 | + | EXACTMASS 608.174120354 |
| − | AVERAGEMASS 608.54468 | + | AVERAGEMASS 608.54468 |
| − | SMILES OC(C1O)C(OC(O5)C(O)C(C(O)C(C)5)O)C(OC1OC(=C3c(c4)cc(c(c4)O)O)C(=O)c(c(O3)2)c(O)cc(OC)c2)C | + | SMILES OC(C1O)C(OC(O5)C(O)C(C(O)C(C)5)O)C(OC1OC(=C3c(c4)cc(c(c4)O)O)C(=O)c(c(O3)2)c(O)cc(OC)c2)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-2.3553 1.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3553 0.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6407 0.2837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9262 0.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9262 1.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6407 1.9338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2117 0.2837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5028 0.6962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5028 1.5214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2117 1.9338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2117 -0.3595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4025 2.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1307 1.6721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8589 2.0927 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8589 2.9336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1307 3.3539 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4025 2.9336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6407 -0.5410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6383 3.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2689 0.1895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1307 4.1946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0514 -1.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7391 -1.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5209 -0.7248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7391 0.0432 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0514 0.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2695 -0.3232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3140 -0.3243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1263 -1.8138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5672 -2.1593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0362 -1.1182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3272 -2.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9985 -3.4726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6304 -3.2920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2663 -3.4726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5950 -2.9033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9629 -3.0839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2343 -3.8715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3952 -3.2058 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7251 -2.8576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2225 -4.1946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9961 1.8913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6383 3.0036 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
16 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 20 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
34 38 1 0 0 0 0
33 39 1 0 0 0 0
32 40 1 0 0 0 0
35 41 1 0 0 0 0
30 36 1 0 0 0 0
42 43 1 0 0 0 0
1 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 ^ OCH3
M SBV 1 47 0.6408 -0.3700
S SKP 5
ID FL5FCCGS0006
FORMULA C28H32O15
EXACTMASS 608.174120354
AVERAGEMASS 608.54468
SMILES OC(C1O)C(OC(O5)C(O)C(C(O)C(C)5)O)C(OC1OC(=C3c(c4)cc(c(c4)O)O)C(=O)c(c(O3)2)c(O)cc(OC)c2)C
M END