Mol:FL7AACGL0014
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.9210 -0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9210 -0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9210 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9210 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3647 -1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3647 -1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8084 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8084 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8084 -0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8084 -0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3647 0.2367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3647 0.2367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2521 -1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2521 -1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6958 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6958 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6958 -0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6958 -0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2521 0.2367 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -2.2521 0.2367 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1397 0.2366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1397 0.2366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5728 -0.0908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5728 -0.0908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0058 0.2366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0058 0.2366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0058 0.8913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0058 0.8913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5728 1.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5728 1.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1397 0.8913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1397 0.8913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5610 1.2185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5610 1.2185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3647 -1.6901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3647 -1.6901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5728 1.8731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5728 1.8731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5115 -1.3390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5115 -1.3390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5340 -0.8476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5340 -0.8476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1463 -1.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1463 -1.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8918 -1.3060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8918 -1.3060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6417 -1.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6417 -1.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0295 -0.8476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0295 -0.8476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2840 -1.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2840 -1.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1860 -1.0405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1860 -1.0405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5909 -0.5289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5909 -0.5289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5888 -1.1705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5888 -1.1705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4771 0.2366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4771 0.2366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1427 1.5861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1427 1.5861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7551 0.9147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7551 0.9147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5005 1.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5005 1.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2505 0.9147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2505 0.9147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6382 1.5861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6382 1.5861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8927 1.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8927 1.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4227 1.3932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4227 1.3932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1997 1.9048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1997 1.9048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1976 1.2632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1976 1.2632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1539 -1.4923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1539 -1.4923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8684 -1.9048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8684 -1.9048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7627 0.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7627 0.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4771 0.5289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4771 0.5289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 17 32 1 0 0 0 0 | + | 17 32 1 0 0 0 0 |
| − | 24 40 1 0 0 0 0 | + | 24 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 44 -7.5297 5.0366 | + | M SBV 1 44 -7.5297 5.0366 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 46 -7.5297 5.0366 | + | M SBV 2 46 -7.5297 5.0366 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0014 | + | ID FL7AACGL0014 |
| − | KNApSAcK_ID C00006669 | + | KNApSAcK_ID C00006669 |
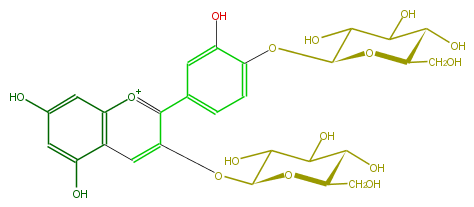
| − | NAME Cyanidin 3,4'-diglucoside | + | NAME Cyanidin 3,4'-diglucoside |
| − | CAS_RN 27459-78-5 | + | CAS_RN 27459-78-5 |
| − | FORMULA C27H31O16 | + | FORMULA C27H31O16 |
| − | EXACTMASS 611.161209944 | + | EXACTMASS 611.161209944 |
| − | AVERAGEMASS 611.52544 | + | AVERAGEMASS 611.52544 |
| − | SMILES O(C(O5)C(C(C(O)C(CO)5)O)O)c(c1)c(cc(c([o+1]2)c(OC(O4)C(C(O)C(C4CO)O)O)cc(c3O)c2cc(O)c3)c1)O | + | SMILES O(C(O5)C(C(C(O)C(CO)5)O)O)c(c1)c(cc(c([o+1]2)c(OC(O4)C(C(O)C(C4CO)O)O)cc(c3O)c2cc(O)c3)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-3.9210 -0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9210 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3647 -1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8084 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8084 -0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3647 0.2367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2521 -1.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6958 -0.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6958 -0.0845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2521 0.2367 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-1.1397 0.2366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5728 -0.0908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0058 0.2366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0058 0.8913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5728 1.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1397 0.8913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5610 1.2185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3647 -1.6901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5728 1.8731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5115 -1.3390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5340 -0.8476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1463 -1.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8918 -1.3060 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6417 -1.5190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0295 -0.8476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2840 -1.0606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1860 -1.0405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5909 -0.5289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5888 -1.1705 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4771 0.2366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1427 1.5861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7551 0.9147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5005 1.1278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2505 0.9147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6382 1.5861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8927 1.3732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4227 1.3932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1997 1.9048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1976 1.2632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1539 -1.4923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8684 -1.9048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7627 0.9414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4771 0.5289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
3 18 1 0 0 0 0
15 19 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
20 8 1 0 0 0 0
1 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
17 32 1 0 0 0 0
24 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SBV 1 44 -7.5297 5.0366
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SBV 2 46 -7.5297 5.0366
S SKP 8
ID FL7AACGL0014
KNApSAcK_ID C00006669
NAME Cyanidin 3,4'-diglucoside
CAS_RN 27459-78-5
FORMULA C27H31O16
EXACTMASS 611.161209944
AVERAGEMASS 611.52544
SMILES O(C(O5)C(C(C(O)C(CO)5)O)O)c(c1)c(cc(c([o+1]2)c(OC(O4)C(C(O)C(C4CO)O)O)cc(c3O)c2cc(O)c3)c1)O
M END