Mol:FLIA1LGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1158 0.9656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1158 0.9656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5595 1.2868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5595 1.2868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0034 0.9658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0034 0.9658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0034 0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0034 0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4267 -0.0332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4267 -0.0332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1501 0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1501 0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1501 0.9658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1501 0.9658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4267 1.2987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4267 1.2987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1158 0.3236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1158 0.3236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5595 0.0026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5595 0.0026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4267 -0.6987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4267 -0.6987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7264 -0.0329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7264 -0.0329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7264 -0.6696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7264 -0.6696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2778 -0.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2778 -0.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8291 -0.6696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8291 -0.6696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8291 -0.0329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8291 -0.0329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2778 0.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2778 0.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2997 -1.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2997 -1.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9866 -0.6932 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.9866 -0.6932 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 3.3578 -1.1831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3578 -1.1831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8923 -0.9753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.8923 -0.9753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.5066 -1.0821 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.5066 -1.0821 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.0332 -0.5948 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 4.0332 -0.5948 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 3.5656 -0.8416 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.5656 -0.8416 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 3.4413 -0.1366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4413 -0.1366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6107 -0.5948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6107 -0.5948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0154 -0.8069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0154 -0.8069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6270 1.2608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6270 1.2608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6150 1.1581 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -4.6150 1.1581 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -4.2568 0.6853 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -4.2568 0.6853 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.7409 0.8859 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.7409 0.8859 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.2432 0.8913 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.2432 0.8913 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.6049 1.2530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6049 1.2530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1318 1.0638 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -4.1318 1.0638 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -5.0755 1.1984 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0755 1.1984 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5949 0.2402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5949 0.2402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4454 0.3898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4454 0.3898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2778 0.9220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2778 0.9220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4041 1.7958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4041 1.7958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3901 1.9625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3901 1.9625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8921 -1.3833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8921 -1.3833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6066 -1.7958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6066 -1.7958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 2 0 0 0 0 | + | 6 7 2 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 3 1 0 0 0 0 | + | 8 3 1 0 0 0 0 |
| − | 1 9 1 0 0 0 0 | + | 1 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 2 0 0 0 0 | + | 5 11 2 0 0 0 0 |
| − | 6 12 1 0 0 0 0 | + | 6 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 15 1 0 0 0 0 | + | 18 15 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 23 26 1 0 0 0 0 | + | 23 26 1 0 0 0 0 |
| − | 22 27 1 0 0 0 0 | + | 22 27 1 0 0 0 0 |
| − | 1 28 1 0 0 0 0 | + | 1 28 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 17 38 1 0 0 0 0 | + | 17 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 21 41 1 0 0 0 0 | + | 21 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 45 | + | M SBL 2 1 45 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 45 3.8921 -1.3833 | + | M SVB 2 45 3.8921 -1.3833 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 43 | + | M SBL 1 1 43 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 43 -4.4041 1.7958 | + | M SVB 1 43 -4.4041 1.7958 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FLIA1LGS0002 | + | ID FLIA1LGS0002 |
| − | KNApSAcK_ID C00010141 | + | KNApSAcK_ID C00010141 |
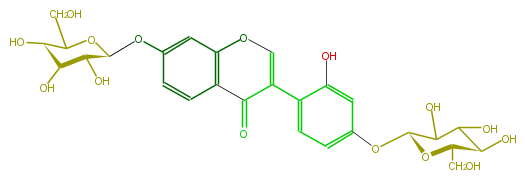
| − | NAME 2'-Hydroxydaidzein 7,4'-di-O-glucoside | + | NAME 2'-Hydroxydaidzein 7,4'-di-O-glucoside |
| − | CAS_RN 88048-22-0 | + | CAS_RN 88048-22-0 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES O[C@@H]([C@@H](O)1)[C@H](Oc(c2)ccc(C3=O)c(OC=C3c(c4)c(cc(O[C@@H](C5O)O[C@@H]([C@H](O)C(O)5)CO)c4)O)2)OC([C@H](O)1)CO | + | SMILES O[C@@H]([C@@H](O)1)[C@H](Oc(c2)ccc(C3=O)c(OC=C3c(c4)c(cc(O[C@@H](C5O)O[C@@H]([C@H](O)C(O)5)CO)c4)O)2)OC([C@H](O)1)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-2.1158 0.9656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5595 1.2868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0034 0.9658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0034 0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4267 -0.0332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1501 0.2998 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1501 0.9658 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4267 1.2987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1158 0.3236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5595 0.0026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4267 -0.6987 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7264 -0.0329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7264 -0.6696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2778 -0.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8291 -0.6696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8291 -0.0329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2778 0.2854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2997 -1.0202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9866 -0.6932 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
3.3578 -1.1831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8923 -0.9753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.5066 -1.0821 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.0332 -0.5948 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
3.5656 -0.8416 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
3.4413 -0.1366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6107 -0.5948 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0154 -0.8069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6270 1.2608 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6150 1.1581 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-4.2568 0.6853 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.7409 0.8859 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.2432 0.8913 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.6049 1.2530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1318 1.0638 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-5.0755 1.1984 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5949 0.2402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4454 0.3898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2778 0.9220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4041 1.7958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3901 1.9625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8921 -1.3833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6066 -1.7958 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 7 2 0 0 0 0
7 8 1 0 0 0 0
8 3 1 0 0 0 0
1 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 2 0 0 0 0
6 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 15 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
18 19 1 0 0 0 0
24 25 1 0 0 0 0
23 26 1 0 0 0 0
22 27 1 0 0 0 0
1 28 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
28 32 1 0 0 0 0
17 38 1 0 0 0 0
34 39 1 0 0 0 0
39 40 1 0 0 0 0
21 41 1 0 0 0 0
41 42 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 45
M SMT 2 CH2OH
M SVB 2 45 3.8921 -1.3833
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 43
M SMT 1 CH2OH
M SVB 1 43 -4.4041 1.7958
S SKP 8
ID FLIA1LGS0002
KNApSAcK_ID C00010141
NAME 2'-Hydroxydaidzein 7,4'-di-O-glucoside
CAS_RN 88048-22-0
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES O[C@@H]([C@@H](O)1)[C@H](Oc(c2)ccc(C3=O)c(OC=C3c(c4)c(cc(O[C@@H](C5O)O[C@@H]([C@H](O)C(O)5)CO)c4)O)2)OC([C@H](O)1)CO
M END